| Arraystar LncRNA芯片 人类 V4.0版本; 小鼠 V3.0版本 |
LncRNA高通量筛选利器,同时检测lncRNA和mRNA的表达量变化 |
优势
介绍 DNA转录形成的RNA中绝大部分是长链非编码RNA(lncRNAs),lncRNA与mRNA分子结构相似,但没有蛋白编码能力。这些lncRNAs有的从基因组中蛋白编码基因附近位置转录形成,有的位于基因间区,即lincRNA。(图1, 顶部). LncRNAs能够在多个层面(转录及转录后层面)调控基因的表达水平,参与了基因组印记、染色质表观修饰、转录激活、转录抑制等多种重要调控过程(图1, 底部)。越来越多的LncRNAs在疾病和生物学过程中发挥重要功能。一些悬而未决的谜团,例如染色体沉默、生长发育及分化过程、肿瘤的发生发展,已经被lncRNAs揭开。与mRNA相比,lncRNAs具有更好的组织特异性,是进行分子标志物研究的理想分子。目前基因表达谱检测技术不仅可以检测传统的蛋白编码mRNA,也可以同时检测非编码RNA分子。
|
| Arraystar是lncRNA芯片技术的领跑者,创新性的将lncRNAs与mRNAs设计在同一张芯片上进行检测。迄今为止,利用Arraystar lncRNA芯片已经发表了大量高水平文章,成为研究lncRNA强有力的工具。 目前Arraystar通过迅速整合学科前沿和数据库中的新lncRNA,发布了最新版本的人类V4.0 和小鼠V3.0 LncRNA表达谱芯片。
最全面的检测范围:金标准LncRNAs, 可靠的LncRNAs以及编码蛋白的mRNAs 与具有详尽注释的蛋白编码基因不同, lncRNAs常常缺乏注释,信息分散且收集不全。Arraystar拥有高质量的转录组和lncRNA数据库,通过生物信息学方法建立了科学、严谨的lncRNA筛选流程。 Arraystar人类LncRNA V4.0芯片共收录了40,173 个lncRNAs,主要分为两大类:7,506个金标准LncRNAs和32,667个可靠的LncRNAs,实现了对所有权威数据库(如Refseq, USCS Known Genes, GENCODE, lincRNA catalogs, lncRNAdb, T-UCRs, RNAdb, NRED 等)、高水平文章和超过47 Tb RNA-seq数据中的lncRNA最全面、最可靠和最及时的整理和收集。 金标准lncRNAs
除了金标准lncRNA外,其他的 lncRNA序列主要通过整合数据库和经典文献中的转录单位(Transcription Units)而获得。DNA链的转录起始于DNA模板的一个特殊起点,并在一个终点处终止,此转录区域称为转录单位。根据转录本长度、来源数据库和其他有效信息,每个转录单位挑选一个最具代表性的lncRNA进行检测。最终从308,525个lncRNA序列中筛选出32,667个可靠的 LncRNAs分子。 编码蛋白的mRNAs 根据与UniProt蛋白数据库的匹配程度,将RefSeq 及GENCODE数据库中收录并筛选的蛋白编码mRNA分为3类,依次为权威的、非权威的和不匹配的。Arraystar的人类V4 LncRNA芯片共挑选并收录了 20,730个编码蛋白的转录本。 LncRNAs的详细注释和功能分析 一站式芯片技术服务包含系统而详细的lncRNA注释、子类分析等重要分析项目,这些信息有助于揭示lncRNAs复杂的生物学功能。通过研究发现,lncRNAs在凋亡、分化、发育等多种生物学过程以及人类疾病,如癌症、神经系统疾病及心血管疾病中发挥重要功能。针对上述研究报道的所有LncRNAs,我们提供了全面的注释便于交叉引用,帮助您深入了解lncRNAs的生物功能和分子机制。
|
| 转录本特异性探针 与mRNA一样,LncRNAs是以转录本的形式发挥功能的。特定基因位点往往可以转录出多个没有开放阅读框,不同功能的转录本。大部分芯片平台只针对基因的3’端设计“基因特异性”探针,无法有效区分不同转录本。而Arraystar LncRNA芯片针对剪切连接位点或外显子序列设计了“转录本特异性”探针,能够实现对不同转录本的准确、特异性检测(图 2) 。
Arraystar LncRNA 芯片规格
|
|||||||||||||||||||||||||||||||||||||||||
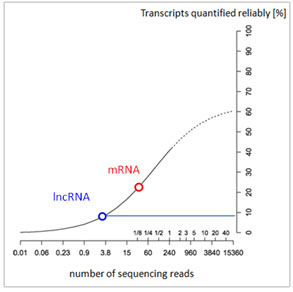
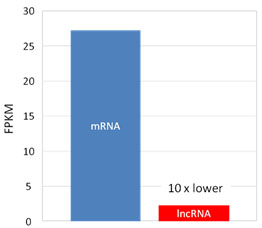
比RNA-seq更适合于LncRNA表达谱检测 利用RNA-seq进行lncRNA表达谱检测的一个主要问题在于绝大部分测序reads被表达丰度很高的RNA,如管家基因所占据。由于reads分布的这一统计学特征,使得RNA-seq对于低丰度RNA的定量不可靠 (图 3)。LncRNA的平均表达水平约为mRNA的1/10[1,5-6] (图 4),只有不到10%的lncRNAs能够被可靠的定量 (图 3)。
差异表达基因的qPCR验证 芯片筛选得到的差异表达基因需要经过qPCR验证才能进行后续研究。当挑选指标进行验证时,差异变化倍数 (fold change, FC),统计学显著性(p-values)以及初始信号值都会对结果产生影响。 FDR值 用来评估和控制假阳性出现的次数。 qPCR验证的一般指导原则包括Tm值、GC含量、是否形成引物二聚体、引物特异性等。除了上述原则外,lncRNA的qPCR验证还需要考虑以下因素:更建议选用数据库(source)中经过充分验证确定的lncRNA序列;位置关系(relationship)为Antisense类型的lncRNA 需要设计链特异性RT引物进行cDNA合成 (图1, 顶部);位置关系(relationship)为sense-overlapping 类型的lncRNAs引物设计在与mRNA的非重叠区域(图1, 顶端);引物位置尽可能靠近探针 ; qPCR扩增产物需横跨剪接位点以保证转录本特异性扩增。理论上应该使用与芯片检测相同的RNA样本进行qPCR验证。 |
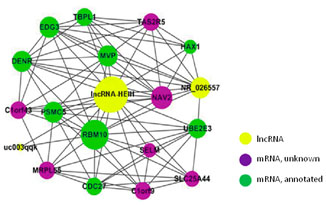
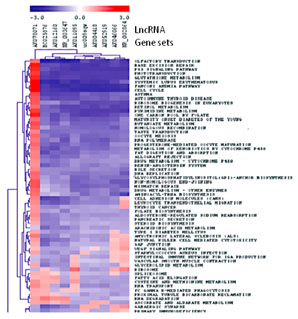
| LncRNA高级数据分析 深入的数据挖掘和高级分析项目(如图5、图6所示), 帮助您充分利用芯片所提供的海量信息。
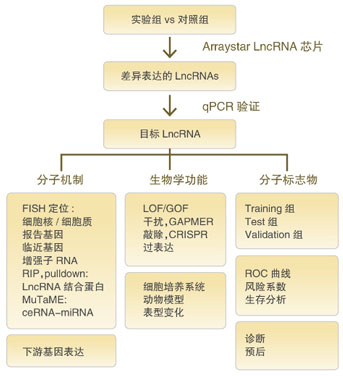
LncRNA技术路线图
引用 Arraystar LncRNA 芯片所发表的论文 Liu, B. et al. (2015) "A cytoplasmic NF-kappaB interacting long noncoding RNA blocks IkappaB phosphorylation and suppresses breast cancer metastasis." Cancer Cell 27(3):370-81 [PMID: 25759022] Xing, Z. et al. (2014) "lncRNA directs cooperative epigenetic regulation downstream of chemokine signals." Cell 159(5):1110-25 [PMID: 25416949] Yuan, J.H. et al. (2014) "A long noncoding RNA activated by TGF-beta promotes the invasion-metastasis cascade in hepatocellular carcinoma." Cancer Cell 25(5):666-81 [PMID: 24768205] Zhao, X.Y. et al. (2014) "A long noncoding RNA transcriptional regulatory circuit drives thermogenic adipocyte differentiation." Mol Cell 55(3):372-82 [PMID: 25002143] Ouyang, J. et al. (2014) "NRAV, a long noncoding RNA, modulates antiviral responses through suppression of interferon-stimulated gene transcription." Cell Host Microbe 16(5):616-26 [PMID: 25525793] Kumarswamy, R. et al. (2014) "Circulating long noncoding RNA, LIPCAR, predicts survival in patients with heart failure." Circ Res 114(10):1569-75 [PMID: 24663402] Garzon, R. et al. (2014) "Expression and prognostic impact of lncRNAs in acute myeloid leukemia." Proc Natl Acad Sci U S A 111(52):18679-84 [PMID: 25512507] Liu, Q. et al. (2014) "Long noncoding RNA related to cartilage injury promotes chondrocyte extracellular matrix degradation in osteoarthritis." Arthritis Rheumatol 66(4):969-78 [PMID: 24757148] Yang, F. et al. (2013) "Repression of the long noncoding RNA-LET by histone deacetylase 3 contributes to hypoxia-mediated metastasis." Mol Cell 49(6):1083-96 [PMID: 23395002] |
参考文献 1. Cabili, M.N. et al. (2011) Genes Dev 25(18):1915-27[PMID: 21890647]2. Labaj, P.P. et al. (2011) Bioinformatics 27(13):i383-91 [PMID: 21685096] 3. Kretz, M. et al. (2012) Genes Dev 26(4):338-43 [PMID: 22302877] 4. Xu, W. et al. (2011) Proc Natl Acad Sci U S A 108(9):3707-12 [PMID: 21317363] 5. Guttman, M. et al. (2010) Nat Biotechnol 28(5):503-10 [PMID: 20436462] 6. Derrien, T. et al. (2012) Genome Res 22(9):1775-89 [PMID: 22955988] 7. Khalil A.M. et al. (2009) Proc. Natl. Acad. Sci. U.S.A. 106(28):11667-72 [PMID: 19571010] 8. Cabili M.N. et al. (2011) Genes Dev. 25(18):1915-27 [PMID: 21890647] 9. Ørom U.A. et al. (2010) Cell 143(1):46-58 [PMID: 20887892] 10. Rinn J.L. et al. (2007) Cell 129(7):1311-23 [PMID: 17604720] 11. Bejerano G. et al. (2004) Science 304(5675):1321-5 [PMID: 15131266] 12. Pruitt K.D. et al. (2005) Nucleic Acids Res. 33(Database issue):D501-4 [PMID: 15608248] 13. Pang K.C. et al. (2005) Nucleic Acids Res. 33(Database issue):D125-30 [PMID: 15608161] 14. Pang K.C. et al. (2007) Nucleic Acids Res. 35(Database issue):D178-82 [PMID: 17145715] 15. Mercer T.R. et al. (2008) Proc. Natl. Acad. Sci. U.S.A. 105(2):716-21 [PMID: 18184812] 16. Hsu F. et al. (2006) Bioinformatics 22(9):1036-46 [PMID: 16500937] 17. Harrow J. et al. (2006) Genome Biol. 7 Suppl 1:S4.1-9 [PMID: 16925838] 18. Guttman M. et al. (2009) Nature 458(7235):223-7 [PMID: 19182780] 19. Dinger M.E. et al. (2009) Nucleic Acids Res. 37(Database issue):D122-6 [PMID: 18829717] 20. Benson D.A. et al. (2004) Nucleic Acids Res. 32(Database issue):D23-6 [PMID: 14681350] 21. Amaral P.P. et al. (2011) Nucleic Acids Res. 39(Database issue):D146-51 [PMID: 21112873] 22. Sun L. et al. (2012) BMC Bioinformatics 13:331 [PMID: 23237380] 23. Ramos A.D. et al. (2013) Cell Stem Cell 12(5):616-28 [PMID: 23583100] 24. Ponjavic J. et al. (2009) PLoS Genet. 5(8):e1000617 [PMID: 19696892] 25. Willingham A.T. et al. (2005) Science 309(5740) :1570-3 [PMID: 16141075] 26. The ENCODE Consortium. . https://genome.ucsc.edu/ENCODE/protocols/dataStandards/ ENCODE_RNAseq_Standards_V1.0.pdf 27. Clark M.B. et al. (2015) Nat. Methods 12(4):339-42 [PMID: 25751143] 28. Iyer M.K. et al. (2015) Nat. Genet. 47(3):199-208 [PMID: 25599403] 29. Quek X.C. et al. (2015) Nucleic Acids Res. 43(Database issue):D168-73 [PMID: 25332394] 30. Skroblin P. and M. Mayr (2014) Circ. Res. 115(7):607-9 [PMID: 25214572] 31. Kurian L. et al. (2015) Circulation 131(14):1278-90[PMID: 25739401] 32. Hu Y. et al. (2014) Cancer Res. 74(23):6890-902 [PMID: 25277524] 33. Fang X.Y. et al. (2015) Cancer Lett. 356(2 Pt B):357-66 [PMID: 25444905] 34. Howald C. et al. (2012) Genome Res. 22(9):1698-710 [PMID: 22955982] 35. Miura P. et al. (2013) Genome Res. 23(5):812-25 [PMID: 23520388] 36. St Laurent G. et al. (2015) Trends Genet. 31(5):239-51 [PMID: 25869999] 37. Yan X. et al. (2015) Cancer Cell 28(4):529-40 [PMID: 26461095] 38. Hackermuller J. et al. (2014) Genome Biol. 15(3):R48 [PMID: 24594072] 39. St Laurent G. et al. (2013) Genome Biol. 14(7):R73 [PMID: 23876380] 40. Chen G. et al. (2013) Nucleic Acids Res. 41(Database issue):D983-6 [PMID: 23175614] 41. Ferdin J. et al. (2013) Cell Death Differ. 20(12):675-87 [PMID: 24037088] 42. Silva J.M. et al. (2010) Genomics 95(6):355-62[PMID: 20214974] 康成生物独家提供技术服务 康成生物国内独家提供Arraystar LncRNA芯片全程优质技术服务;目前康成客户LncRNA芯片研究文章已达160余篇,其中多篇发表在国际顶尖杂志Cancer Cell, Molecular Cell, Hepatology等上。 |