聚焦品牌优势
 |
基于circRNA研究领域顶尖期刊和权威公共数据库,运用强大的信息筛选系统,挑选出可靠的circRNA。 |
 |
剪接位点特异性探针与Rnase R预处理双重保障,即便在相应线性RNA存在的情况下也能够特异性检测circRNA。 |
 |
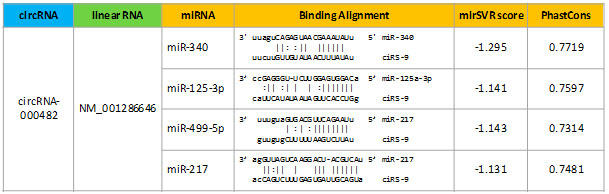
数据报告提供circRNA可能结合的miRNA以及对应的host gene和线性RNA,便于从不同角度研究circRNA的作用机制。 |
 |
circRNA的特征决定了通过高通量测序的方法检测结果不准确,Arraystar circRNA芯片可以恰到好处地破解这个难题。 |
circRNA介绍
circRNA是一类由特殊剪接机制形成的、具有闭合环状结构的非编码RNA,也是目前生命科学和医学领域的研究热点分子。circRNA分子的组织特异性、疾病特异性、时序特异性及高稳定性等特征,使得circRNA作为临床疾病的biomarker具有明显的优势。circRNA作为相应线性RNA的补充,表达量水平高,可以在细胞核中稳定存在并在细胞质中富集;circRNA具有大量miRNA结合位点,由于不完全配对,故不会被miRNA靶定降解;由于这些特征,circRNA被公认为高效的天然miRNA海绵,在基因调节中发挥竞争性内源RNA(ceRNA)作用。此外,一些内含子类型的circRNA会促进宿主基因转录。生物学功能强大的circRNA分子犹如一座亟待开发的“金矿”。 为了能够帮助科研工作者对circRNA进行更好地研究,美国Arraystar公司在其首款circRNA芯片基础上迅即升级版本为V2.0,以便于研究者对不同生理和病理状态下对 circRNA的表达变化进行系统的研究。
circRNA竞争性吸附miRNA
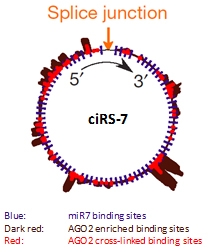
circRNA序列上含有大量的miRNA结合位点,当circRNA与miRNA同处于细胞浆时会引起circRNA对miRNA的吸附(miRNA sponge)。例如,通过PAR-CLIP实验证实了ciRS-7与miR-7以及RNA诱导沉默复合体(RISC)重要组成成分AGO2蛋白结合;对生物素偶联的miRNA进行pull down实验,进一步验证了ciRS-7吸附miR-7功能的存在。(图1)
|
图1. ciRS-7序列上含有七十多个miR-7结合位点,它可与miR-7以及AGO2蛋白结合。 |
circRNA调节RNA结合蛋白(RBP)
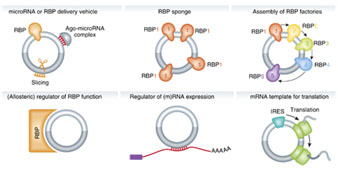
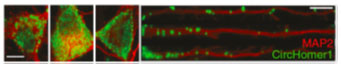
circRNA可以与多种RNA结合蛋白(RBP)作用,进而发挥相应的分子生物学功能(图2);例如circRNA可以充当RBP的亚细胞转移或定位的载体(图3)、作为RBP的分子海绵,组装RBP复合物以及作为酶活性调节的辅因子等。Antisense类型的circRNA还可以通过碱基互补配对的方式与相应的mRNA结合,进行调控mRNA的稳定性。
|
图2. circRNA与RNA结合蛋白(RBP)之间的相互作用 |
|
|
|
图3. circRNA在神经元胞体及树突动态分布示意图 |
circRNA的生物学功能
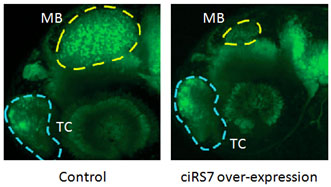
miRNA分子调控生物体内1/3左右的基因,circRNA对miRNA的吸附会影响基因的表达,进而引起生物学表型或状态的改变(图4)。相当大一部分环状RNA的靶标基因是介导信号传导的蛋白激酶。研究还表明,circRNA的异常表达往往和临床疾病相关,比如肿瘤、阿尔茨海默症、动脉粥样硬化等。
|
图4. 斑马鱼胚胎过表达ciRS-7(右图)会导致中脑(MB)缩小,而端脑(TC)大小不受影响。 |
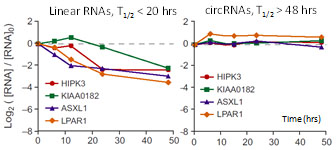
circRNA的高稳定性
circRNA分子独特的闭合环状结构,不受核酸外切酶影响,使circRNA的稳定性要远远高于同一个基因转录的相应线性RNA分子(图5)。circRNA的高稳定性预示其非常适用于临床biomarker研究。
|
图5. circRNA在细胞内稳定性远高于同一个基因转录的相应线性RNA,前者半衰期>48h(右图),而后者半衰期<20h(左图)。 |
circRNA高通量筛选的利器:RNA-seq or microarray?
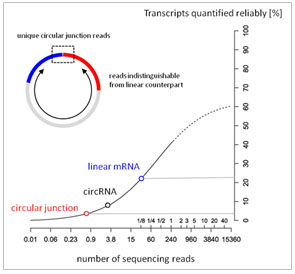
circRNA在细胞内含量极低,约占线性RNA的5-10%,通过测序获得的circular junction reads 数目更低。RNA测序仅仅能有效检测低于5% 的circRNAs(图6),即便将测序量提高到300M reads,对于检测准确度的影响也是微乎其微。
测序不是circRNA高通量筛选的优质检测手段,circRNA-seq需要双端模式和极高的测序量。此外,后续的数据分析(如mapping所需数据库、circRNA转录本组装、特殊生物信息学算法、计算机筛选程序等)也存在一系列难题。由于上述因素所限,通过circRNA-seq发现新的circRNA的可能性几乎为零。
circRNA芯片采用特异性剪接位点探针与外切酶预处理双重保障,能够准确检测样本中circRNA的表达。芯片杂交不受RNA丰度的影响,即便在一个细胞中只含一个circRNA拷贝也能够做到特异性检测。此外,芯片避免了circRNA-seq复杂的建库过程,其高效成熟的技术是circRNA高通量筛选的优质平台。
|
图6. 定量可靠的转录本比例与测序深度关系图。RNA测序一般产生低于30M mRNA reads(蓝圈),低于0.5M circular junction reads(红圈),低于5%的circular junction真正得到有效检测。(Adopted from Labaj et al, (2011) Bioinformatics [PMID 21685096].) |
国际尖端circRNA研究利器
Arraystar circRNA芯片是准确检测样本中circRNA含量理想的工具。剪接位点特异性探针与Rnase R预处理双重保障,即便在相应线性RNA存在的情况下也能够做到特异性检测circRNA。数据报告中详尽的circRNA注释信息十分便于客户进行circRNA机制研究。
|
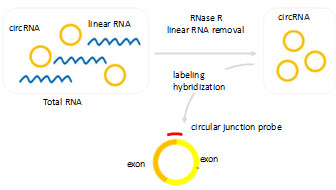
图7. circRNA芯片实验大体流程(上图)及详细的circRNA注释(下图): circRNA对应的线性RNA及miRNA的结合位点。 |
研究技术路线
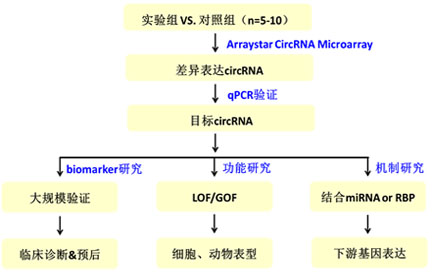
通过芯片筛选获得组间差异表达的circRNA,接下来通过低通量circRNA-PCR的验证(图8)锁定目标circRNA分子。目标circRNA分子可以展开不同方向的研究:临床biomarker或功能机制(图9)。
|
|
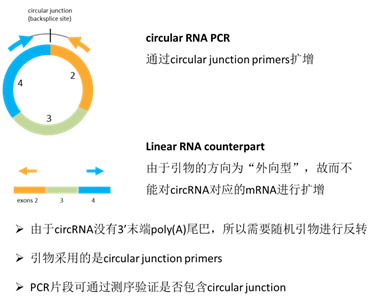
图8. circRNA PCR验证引物设计示意图 |
图9. circRNA基本研究思路示意图 |
环状RNA芯片参数
|
Human |
Mouse |
Rat |
| Total Number of unique circRNAs |
13,617 |
14,236 |
14,145 |
| Probe Length |
60nt |
60nt |
60nt |
| Probe Region |
Circular junctions of circRNA |
| Probe Specificity |
Transcript-specific |
| circRNA Enrichment |
RNase R treatment |
| Labeling Method |
Labeling by random primers |
| Circular RNA Sources |
| Salzman’s circRNAs (2013) |
8,529 |
|
|
| Memczak’s circRNAs (2013) |
1,601 |
1,750 |
|
| Zhang’s circRNAs (2013) |
93 |
|
|
| Zhang’s circRNAs (2014) |
4,980 |
|
|
| Jeck’s circRNAs (2014) |
3,769 |
|
|
| Guo’s circRNAs (2014) |
5,536 |
570 |
|
| You's circRNAs (2015) |
|
13,300 |
12,298 |
| Mouse circRNA orthologs |
|
|
1,668 |
| Human circRNA orthologs |
|
|
179 |
| Array Format |
8 * 15K |
8 * 15K |
8 * 15K |
参考文献
[1] Guo, J. U., V. Agarwal, et al. (2014). "Expanded identification and characterization of mammalian circular RNAs." Genome Biol 15(7): 409.
[2] Jeck, W. R., J. A. Sorrentino, et al. (2013). "Circular RNAs are abundant, conserved, and associated with ALU repeats." RNA 19(2): 141-157.
[3] Memczak, S., M. Jens, et al. (2013). "Circular RNAs are a large class of animal RNAs with regulatory potency." Nature 495(7441): 333-338.
[4] Salzman, J., R. E. Chen, et al. (2013). "Cell-type specific features of circular RNA expression." PLoS Genet 9(9): e1003777.
[5] Zhang, X. O., H. B. Wang, et al. (2014). "Complementary sequence-mediated exon circularization." Cell 159(1): 134-147.
[6] Zhang, Y., X. O. Zhang, et al. (2013). "Circular intronic long noncoding RNAs." Mol Cell 51(6): 792-806.
[7] You X. et. al. (2015) “Neural circular RNAs are derived from synaptic genes and regulated by development and plasticity.” Nat Neurosci.
2015 Apr;18(4):603-10
[8] Hentze and Preiss (2013) “Circular RNAs: splicing's enigma variations.” EMBO J 3;32(7):923-5.
康成生物是Arraystar公司在中国地区的唯一代理商,提供从样品到数据分析的全程技术服务。
|