
-

生物通官微
陪你抓住生命科技
跳动的脉搏
Science:通过全基因组测序揭示DNM突变与强迫症中的染色质修饰有关
【字体: 大 中 小 】 时间:2022年01月14日 来源:AAAS
编辑推荐:
本研究出于对识别神经精神疾病发病机制背后的超罕见、潜在的高度外显性遗传变异的日益增长的兴趣,首次通过WGS进行全基因组 DNM 分析描述了患有强迫症的家族。研究结果发布在2022年1月12日的Science杂志上。
强迫症(OCD)是一种慢性焦虑症,具有重要的遗传基础和广泛未被发现的病因。最近对强迫症的从头突变 (DNM) 外显子组测序研究加强了罕见变异导致风险的假设。据我们所知,我们对 53 个受强迫症影响的后代的亲子家族进行了第一次全基因组测序,以调查所有罕见的(DNM)变异和插入/缺失。我们观察到启动子锚定染色质环(经验P = 0.0015)和组蛋白标记频率高的区域(经验P= 0.0001)。影响编码区的突变在人类大脑发育过程中参与染色质修饰的基因的共表达模块中显着富集。四genes- SETD5,KDM3B,ASXL3和FBL -had强有力的证据汇总和功能融合转录的表观遗传调控,这意味着一个重要的OCD风险机制。我们的数据表征了不同的全基因组 DNM,并强调了染色质修饰在 OCD 病因学中的影响。
介绍
强迫症(OCD)是一种严重的神经精神障碍,其特征在于重复的,强迫性的想法,和/或强迫运动或仪式,大约1-3%会出现终生患病(1 - 3)。强迫症的诊断通常基于观察到的行为、症状持续时间和功能损害,而不是基于对疾病病因的生物学理解。这明显阻碍了在开发更精确的诊断和更好的治疗以改善患者预后方面的进展。流行病学研究已经证明遗传因素对强迫症病因的重要影响。根据同卵双生和异卵双胞胎研究的一致率,OCD 的遗传率约为 50%(4)。尽管传统遗传学研究已经突出了一些假定的染色体区域 ( 5 ),但尚未确定 OCD 的致病基因 ( 6 )。
最近,全基因组关联研究 (GWAS) ( 7 , 8 ) 已经确定了与 OCD 相关的单核苷酸多态性 (SNP) 在谷氨酸信号传导和突触功能中的作用。然而,基于 SNP 的 OCD 遗传力估计为 0.22 ( 7 ),这表明对疾病风险的剩余贡献不能用弱相关的常见 SNP ( 9)。基于流行病学研究的强迫症的深刻遗传性与 GWAS 估计的有限多基因风险之间的明显矛盾,即所谓的“缺失遗传性”,引发了“从头范式”的假设,这表明了潜在的作用强迫症发展过程中的罕见变异。最近,Cappi等人对亲子强迫症三重奏进行了两项全外显子组测序 (WES) 研究。( 10 , 11 ) 已经显示出令人信服的证据证明突变 (DNM) 在 OCD 中的作用,这进一步证明了(DNM)变异在精神疾病的遗传结构中具有重要作用的假设 ( 12 )。两项强迫症外显子组研究 ( 10 ,11 )(三人组的数量分别 = 20 和 184)估计约 22% 的 OCD 患者携带可估计疾病发生的破坏性编码 DNM。同时,他们强调了在强迫症的病理学中具有潜在作用的基因,例如CHD8和SCUBE1。
然而,大多数关于强迫症易感性的大规模研究都集中在通过靶向测序 ( 13 ) 或外显子组测序方法 ( 10 , 11 )将疾病风险归因于基因组的蛋白质编码区域。基因间、非编码 RNA (ncRNA) 和大结构变异 (SV) 区域 ( 14 , 15 ) 中明显的高渗透性致病变异也是已知的,这表明全基因组测序 (WGS) 检测到的突变类别和大小比WES,可以被认为是研究强迫症的首选基因组平台。此外,WGS 在其他神经精神障碍研究中的应用,例如自闭症谱系 (ASD) ( 14 –16 ) 和智力障碍 (ID) ( 17 ) 已经展示了其在了解基因组规模上 DNM 的总体负担及其对疾病的贡献方面的无与伦比的能力。
因此,在之前对 OCD 的外显子组研究之后,我们应用 WGS 和生物信息学分析来研究一组 OCD 亲子三重奏,以识别基因组规模的从头变异,包括单核苷酸变异 (SNV)、小插入和缺失 (indels ) 和 SV。在 53 个亲子家庭中,我们发现了强有力的证据表明在启动子相互作用的环中突变发生率很高。我们还观察到 DNM 优先发生在不耐受基因和影响染色质修饰的基因上。此外,我们鉴定了三种高置信度的染色质修饰物——SETD5(包含 SET 的结构域 5)、KDM3B(赖氨酸去甲基化酶 3B)和ASXL3(转录调节因子 3)——作为基于多条证据的强迫症候选风险基因。最后,我们的综合分析发现,受强迫症和图雷特障碍 (TD) DNM ( 18 – 20 )影响的基因具有相似的生物学功能,但会影响不同的大脑区域和细胞类型表达模式,这在一定程度上解释了两者之间复杂的共病关系。两种神经发育障碍。
我们对来自 53 个家庭的 160 个 DNA 样本进行了 WGS,其中 54 名强迫症患者及其未受严重精神病影响的父母(材料和方法以及表 S1)。质量控制后,来自 51 个三人组和一个四人组的 53 名先证者以及他们未受影响的父母留在了随后的分析中,其中一个三人组家庭被移除(参与者总数为 157 人)。平均而言,89.1% 的基因组区域在个体层面被 20 次或更多读数覆盖。平均测序深度为 29.92×(图 S1)。在 53 个亲子家庭中,我们最初确定了 4143 个新的 SNV 和短插入缺失。在以~96% 阳性率进行 Sanger 测序验证后(参见材料和方法以及表 S2),我们保留了 4062 个 DNM 列表(表 S2),突变率为 1.34 × 10 -8(95% 置信区间:1.26 × 10 -8到 1.41 × 10 -8)每个碱基对。每个先证者的 DNM 数量在基因组中从 51 到 117 不等(外显子组中从零到四个)。35 名先证者携带一个或多个外显子 DNM。在每个个体数DNMS的是0.93个SNV和0.13跨编码区的插入缺失,62.5对于个SNV,以及14.7整个基因组,类似于在精神疾病的其他家庭为基础的WES和WGS研究报告插入缺失(14 - 16 )。
接下来,我们计算了全球五种一般突变类型的相对突变率(特定类型的 DNM 数量除以研究中 DNM 的总数):外显子、内含子、非翻译区 (UTR)、ncRNA 和基因间。的结果类似先前在ASD大WGS研究(报道14),OCD先证者和Simons Simplex资料集(SSC)控件(之间的差异15在大多数类型)没有达到,除了非编码RNA的多重检验校正后统计学意义(P = 0.02)。此外,我们将破坏性 DNM 类别定义为导致从头功能丧失(LoF;移码插入缺失、停止增益或剪接位点突变)或致病错义[由 SIFT ( 21 ) 和 PolyPhen-2 (22 ) 具有破坏性影响,以及组合注释依赖耗尽 (CADD) ( 23 ) ≥ 20]的 PHRED 标度评分。然后,我们通过计算与同义突变率的相对外显子突变率来评估 OCD 对 SSC 对照的破坏性突变率( 15),因为使用同义突变作为内部对照应该能够抵抗由比较引起的潜在伪影。来自不同研究的数据。我们观察到显着更高的突变率导致 OCD 中的 DNM [ P = 0.02,优势比 (OR) = 2.32]。此外,鸟嘌呤-胞嘧啶含量(GC 含量)(Wilcoxon 检验P = 0.1)或基因长度(Wilcoxon 检验P = 0.9)在病例和对照中的 DNM 基因之间,表明观察到的 OCD 破坏性突变率的显着性不是由偏差引入的,例如 GC 含量或基因长度。
接着,我们研究了OCD突变是否在功能基因组区域发生更频繁地通过集成来自多个不同的数据源的基因组注释,包括PsychEncode(24),ENSEMBL(25),和流程图表观基因组(26)。在校正多次测试后,我们没有观察到 DNM 直接命中启动子、增强子或具有非编码区的全长基因的全局富集,这也类似于之前对精神分裂症 (SCZ) 三重奏的大型 WGS 研究 ( 14 )。然而,我们观察到我们的 OCD 突变在启动子锚定的染色质环中显着富集(经验P校正< 0.001 和 OR = 1.23;图 1A),它们是启动子相互作用的远端调节元件。为了排除通过研究设计和测序技术问题的潜在的偏见,我们收集了两个额外的控制数据(27,28),发现启动子锚定染色质循环控制DNM分布在不同的研究(图S2相似。科克伦测试P = 0.91)。我们将我们的 OCD 数据与三个对照数据进行了比较,通过二次抽样测试和留一染色体(LOCO)(材料和方法)估计 SE,并获得一致的富集,没有交叉研究异质性(二次抽样:荟萃分析或= 1.25,P < 2.2 × 10 -16,Cochran 检验P = 0.94;LOCO:荟萃分析 OR = 1.25,P< 2.2 × 10 -16,Cochran 检验P = 0.15)。
跨基因组区域的 DNM 比率。
(A和B)(A)不同功能基因组区域和(B)不同染色质状态内每个个体突变率的分布。的P值表示的对照或不同患者的其他疾病的意义和进行校正用于使用假发现率的多个测试。该X相当于由费舍尔的测试,误差条估计OR值轴代表95个%的置信区间。( C ) 原理图和综合基因组学查看器图显示了两个通过定量聚合酶链反应 (qPCR) 验证的 dSV。( D) 比较受相同四种 DNM 影响的基因的不耐受性。SSC 控制,Simons Simplex Collection 兄弟姐妹;pLI,基因对 LoF 突变不耐受的概率;dSV,从头 SV。
 在查看器中打开
在查看器中打开染色质状态是空间环境中多个表观基因组标记的组合,可以捕获描述不同类别的基因组元件的信息,例如启动子、增强子、转录、抑制和重复区域。因此,我们扩展了我们对 DNM 负担的分析,通过将突变映射到 15 种核心染色质状态 ( 26 )来研究它们在病例与对照中表观基因组标记的发生情况。我们观察到锌指基因和重复区域内发生显着更高的突变(染色质状态 8;经验P校正< 0.0001 和 OR = 9.65;图 1B),其特点是组蛋白标记H3K9me3、H4K20me3和H3K36me3频率高,其他标记频率相对较低。与上述类似,染色质状态之间的 DNM 分布在不同对照研究之间高度一致 [Pearson 相关系数 ( R ) > 0.96;如图。S2],并且这些研究的富集结果是一致的(子抽样:荟萃分析 OR = 7.24,P= 1.23 × 10 -10,Cochran 检验P = 0.87;LOCO:荟萃分析 OR = 7.46,P < 2.2 × 10 -16,Cochran 检验P = 0.07)。
除了识别点突变和插入缺失,我们还探讨了从头 SVs (dSVs) 是否在强迫症中起作用。在机器学习检测、手动检查和群体频率过滤(材料和方法)之后,我们确定了 GnomAD 群体中未出现的七种潜在 dSV。其中两个是定量聚合酶链式反应 (qPCR),根据样本可用性验证为真正的从头变体。其中一个被鉴定为患有早发性强迫症的男性患者(ID:WOC3_114_1_PT)的基因FBL的第二个内含子的杂合从头缺失(chr19:40331154-40331255)(表 S3)。该 dSV 跳过了大部分第二个内含子和外显子 3 5' 的两个碱基,并将一个碱基插入外显子 2 的 3',导致FBL 发生类似移码的改变(图 1C)。FBL 编码 Fibrillarin,这是一种核蛋白,可甲基化组蛋白 H2A (H2AQ104me) 的 glutamine-104 并调节染色质结构和活性 ( 29 )。另一名确诊的 dSV 是另一名男性患者(ID:WOC4_34),也患有早发性强迫症。dSV 是 703 个碱基对 (bp) 片段 (chr12:64800102–64800404) 的杂合缺失,它影响XPOT的第一个内含子的部分(图 1C和表 S3),预计对来自 ANNOVAR ( 30 )的基于基因的功能注释。
接下来,我们探讨了已识别的 DNM 的功能影响。由于基因对突变的耐受性不同,通过 LoF 不耐受 (pLI) ( 31 )的概率来衡量,因此我们研究了这种不耐受性是否在受不同突变类别影响的基因之间存在差异(图 1D)。我们观察到,在编码 DNM ( P = 0.003)、破坏性 DNM ( P = 0.03) 或错义 DNM ( P = 0.002)方面,OCD 相关基因明显比 SSC 对照更不耐受突变,但对于同义 DNM ( P = 0.25)。
此外,我们推断来自发育中的人类大脑的转录组数据将提高我们对强迫症病理生理学的理解,因为大脑皮层一直与强迫症有关(32)。我们检查了 Brainspan ( 33 )的表达数据,并通过加权基因共表达网络分析 (WGCNA) ( 34 )构建了产前和产后共表达网络,以访问三组基因列表:第 1 组包含改变蛋白质的 DNM(表 1),第 2 组包含启动子被 DNM 命中的基因,第 3 组是第 1 组和第 2 组的组合。
| 先证者身份证 | 位置 | 突变 | 基因 符号 | 突变类型 | 蛋白质 变化 | 筛 | 多酚2 HDIV | CADD | 分类 |
|---|---|---|---|---|---|---|---|---|---|
| WOC5_160_1 | 18:31324188 | 气相色谱 > G | ASXL3 | 移码 删除 | p.F1460Lfs*5 | 不适用 | 不适用 | 35 | 破坏性 |
| WOC3_114_1_PT | 6:29912028 | AG > AG | HLA-A | 移码 删除 | p.D251Tfs*45 | 不适用 | 不适用 | 32 | 破坏性 |
| WOC5_137_1 | 1:109242401 | CAAA>CAAA | PRPF38B | 移码 删除 | p.K324Efs*59 | 不适用 | 不适用 | 34 | 破坏性 |
| WOC5_26 | 2:114482963 | CA > C | SLC35F5 | 移码 删除 | p.L408Rfs*11 | 不适用 | 不适用 | 35 | 破坏性 |
| WOC5_160_1 | 19:16611793 | CT > CTT | C19orf44 | 移码 插入 | p.K65Efs*15 | 不适用 | 不适用 | 14.66 | 破坏性 |
| WOC5_6 | 19:2939254 | AGTGAGGGAATGACA CCACCCTTACCCAAG GAGGCA>A | ZNF77 | 剪接位点 突变 (移码 缺失) | p.A41Lfs*5 | 不适用 | 不适用 | 16.62 | 破坏性 |
| WOC5_160_1 | 3:49713641 | C > T | 亚太经合组织 | 废话 | p.Q199X | 不适用 | 不适用 | 35 | 破坏性 |
| WOC4_28 | 13:32937534 | T > G | BRCA2 | 废话 | p.L2732X | 不适用 | 不适用 | 39 | 破坏性 |
| WOC5_102_1 | 14:20779873 | G > A | CCNB1IP1 | 废话 | p.R224X | 不适用 | 不适用 | 37 | 破坏性 |
| WOC4_34 | 4:47427852 | C > G | GABRB1 | 废话 | p.Y414X | 不适用 | 不适用 | 27 | 破坏性 |
| WOC4_165_1 | 6:39286844 | C > T | KCNK16 | 废话 | p.W93X | 不适用 | 不适用 | 43 | 破坏性 |
| WOC4_170_1 | 22:25320164 | C > T | GSM1 | 废话 | p.R1008X | 不适用 | 不适用 | 46 | 破坏性 |
| WOC84 | 1:118616533 | G > A | SPAG17 | 错义 | p.R777C | D | D | 29.5 | 破坏性 |
| WOC4_170_1 | X:51640699 | C > T | 魔法1 | 错义 | p.R515C | D | D | 28.9 | 破坏性 |
| WOC5_157_1 | 5:137722015 | A > G | KDM3B | 错义 | p.E362G | D | D | 28.6 | Damaging |
| WOC5_096_1 | 17:19284412 | A > G | MAPK7 | Missense | p.Y158C | D | D | 27.5 | Damaging |
| WOC5_62 | 1:36479519 | C > T | AGO3 | Missense | p.R192W | D | D | 27.4 | Damaging |
| WOC5_178_1 | 3:142741802 | C > G | U2SURP | Missense | p.P376A | D | D | 25.9 | Damaging |
| WOC4_130_1_WY | 3:9476069 | C > T | SETD5 | Missense | p.R77C | D | D | 24.6 | Damaging |
| WOC5_093_1 | 17:18195985 | G > C | TOP3A | Missense | p.P324A | D | D | 25.2 | Damaging |
| WOL3_046_1_ZXY | 19:58438675 | C > T | ZNF418 | Missense | p.G207R | D | D | 21.7 | Damaging |
| WOC5_093_1 | 1:206648328 | C > T | IKBKE | Missense | p.R32C | D | D | 28.8 | Damaging |
| WOC5_099_1 | 4:142949945 | G > T | INPP4B | Missense | p.A922D | D | D | 25.7 | Damaging |
| WOC5_149_1 | 8:131072874 | G > A | ASAP1 | Missense | p.T1048M | D | D | 24.5 | Damaging |
| WOC5_17 | 2:71817395 | C > T | DYSF | Missense | p.A1152V | D | P | 27.9 | VUS |
| WOC5_164_1 | 12:96617430 | A > T | ELK3 | Missense | p.N29I | D | P | 25 | VUS |
| WOC5_4F | 13:76381720 | G > T | LMO7 | Missense | p.R201L | D | P | 23.2 | VUS |
| WOC5_097_1 | 3:38595773 | C > T | SCN5A | Missense | p.V1586M | D | P | 23.1 | VUS |
| WOC5_130_1 | 20:33594249 | T > C | TRPC4AP | Missense | p.T606A | T | P | 25.9 | VUS |
| WOC5_161_1 | X:123519751 | G > A | TENM1 | Missense | p.T1944I | D | B | 23.6 | VUS |
| WOC5_155_1 | 11:62416110 | A > G | INTS5 | Missense | p.V481A | D | B | 23.5 | VUS |
| WOC5_078_1_X | 15:41810235 | C > T | RPAP1 | Missense | p.S1314N | T | B | 22 | VUS |
| WOC5_60 | 16:68225447 | A > G | NFATC3 | Missense | p.T959A | D | B | 21.7 | VUS |
| WOC5_091_1 | 6:32123547 | G > A | PPT2 | Missense | p.M140I | D | B | 20.8 | VUS |
| WOC5_076_1_WSH | 14:91928489 | T > C | PPP4R3A | Missense | p.M451V | T | B | 20.4 | VUS |
| WOC5_15 | 17:40149144 | T > C | DNAJC7 | Missense | p.S38G | T | B | 19.65 | VUS |
| WOC5_4F | 3:102157378 | C > T | ZPLD1 | Missense | p.P32L | 吨 | 乙 | 18.89 | VUS |
| WOC5_094_1 | 19:47207624 | G > A | PRKD2 | 错义 | p.P74S | 吨 | 乙 | 16.23 | VUS |
| WOC5_138_1 | 19:36223863 | C > T | KMT2B | 错义 | p.P2138L | D | 磷 | 15.46 | VUS |
| WOC5_116_1 | 19:57036758 | G > A | ZNF471 | 错义 | p.S367N | D | 乙 | 15.06 | VUS |
| WOC5_152_1 | 12:113759149 | C > T | SLC8B1 | 错义 | p.R54H | 吨 | 乙 | 14.72 | VUS |
| WOC5_39 | 2:217012900 | C > T | XRCC5 | 错义 | p.T524I | 吨 | 乙 | 14.16 | VUS |
| WOC5_39 | 9:125551863 | A > C | OR5C1 | 错义 | p.I218L | 吨 | 乙 | 12.96 | VUS |
| WOC5_084_1_PS | 6:151671185 | T > A | AKAP12 | 错义 | p.D455E | 吨 | 乙 | 6.24 | VUS |
| WOC5_150_1 | 18:76753591 | G > C | SALL3 | 错义 | p.V534L | 吨 | 乙 | 0.008 | VUS |
| WOC5_091_1 | X:114468473 | CCCGCCGCCGCCGCCGCC> CCCGCCGCCGCCGCC | LRCH2 | 非移码 删除 | p.G44del | 不适用 | 不适用 | 16.51 | VUS |
表 1。本研究中鉴定的所有改变蛋白质的 DNM 列表。
VUS,意义不确定的变体。不适用,不可用。
我们首先观察到产前网络中的模块 M16 包含的 set1 基因明显多于预期(双边 Fisher 精确检验P = 0.00012)。根据 Metascape(图 2A和表 S4),M16 的功能丰富,包括组蛋白修饰、神经元投射形态发生和突触组织。在探索 M16 中 DNM 基因的连通性时,我们观察到三个 DNM 基因充当枢纽,即ASXL3 [度数 = 161,kME(模块内连通性的度量)= 0.78],SETD5(度数 = 45,kME = 0.75),和KDM3B(度数 = 246,kME = 0.87),在 M16 中,平均度数为 41。接下来,我们观察到出生后网络中的模块 M11 包含的 set1 基因明显多于预期(P = 0.007)。M11主要富集表观遗传调控相关功能,如共价染色质修饰、组蛋白赖氨酸甲基化和染色质重塑(图2B和表S4)。在研究 M11 中 OCD DNM 基因的连通性时,我们还发现了两个 OCD 基因,SETD5(度数 = 26,kME = 0.92)和KDM3B(度数 = 27,kME = 0.84),它们作为 M11 中的枢纽,平均度数9. 然而,我们没有在两个网络中观察到 set2 或 set3 基因的相似结果。
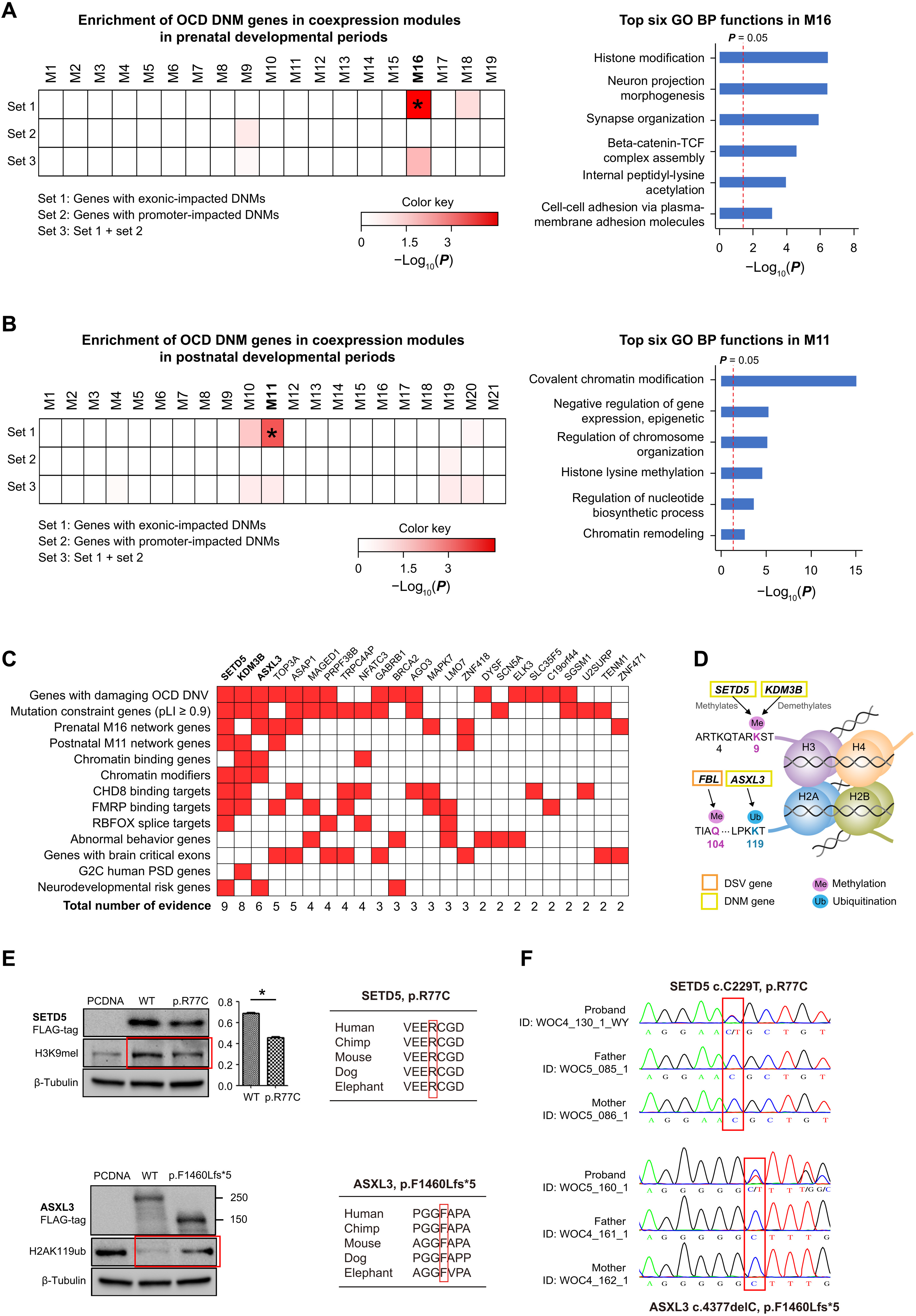
图 2。编码 DNM 的生物学意义。
(A和B)在(A)产前和(B)产后人类大脑(左)和基因本体论(GO)生物过程(BP)的共表达模块中富集受DNM影响的基因,根据相应模块的Metascape富集结果(右) )。热图表示富集程度的P值。( C )编码DNM影响基因的潜在致病性的综合分析。使用 13 个独立的证据来源对受 DNM 影响的基因进行排序。( D ) SETD5、KDM3B和ASXL3的染色质调节活性示意图。( E)蛋白质印迹结果(左)和发现突变位点的跨物种保护(右)。( F ) (E) 突变的 qPCR 验证。CHD8,染色质解旋酶 DNA 结合蛋白 8;FMRP,脆性 X 智力低下蛋白;RBFOX,RNA 结合 fox-1 同源物 1;G2C,认知程序的基因。
如前所述,我们对突变富集、dSV 定位和编码 DNM 共表达的分析始终涉及 OCD 中的染色质区域和染色质修饰相关途径。因此,我们假设与突出显示的途径相关的 DNM 基因子集可能与 OCD 更相关。因此,我们根据 13 个独立的证据来源(中位数 Jaccard 相似性指数 = 0.0244)对所有 45 个 DNM 基因(表 1)进行了优先排序(图 2C;补充材料和方法的详细信息)。我们发现三个基因——即SETD5、KDM3B和ASXL3 ——具有最多的支持证据 (≥6) 并且都是突变不耐受 (pLI ≥ 0.9) 的染色质修饰剂,它们充当网络中枢。SETD5起组蛋白甲基转移酶的作用并将组蛋白 H3 的Lys 9单甲基化,而KDM3B是一种组蛋白去甲基化酶,可明确地将组蛋白 H3 的Lys 9去甲基化。此外,ASXL3是多梳抑制性去泛素酶 (PR-DUB) 复合物的一部分,可将组蛋白 H2A 赖氨酸119去泛素化(图 2D)。此外,对于受患者 WOC3_114_1_PT 从头结构变异影响的基因FBL,先前的研究已经证实其能够甲基化组蛋白 H2A 谷氨酰胺-104 并调节核仁活性(图 2D)。
为了确认这些突变是否如预期那样影响基因在染色质修饰中的功能,我们选择了SETD5和ASXL3基因在人胚胎肾 (HEK) 293T 细胞中进行野生型 (WT) 与突变体实验以验证假设(材料和方法)。我们首先用 WT/突变体 SETD5 和 ASXL3 构建了表达质粒,与 FLAG-tag 融合,用于 HEK293T 细胞的过表达实验。接下来,我们用表达空 pcDNA3.1 载体(作为对照)、WT SETD5 或 ASXL3 和 p.R77C SETD5 或 p.F1460Lfs*5 ASXL3 的质粒转染 HEK293T 细胞。我们从每个细胞组收集蛋白质裂解物,并在 48 小时后通过蛋白质印迹检查组蛋白 H3K9me1 或 H2AK119ub 水平。我们发现与转染空 pcDNA3.1 载体或 WT SETD5 相比,p.R77C SETD5 突变体的转染不能保留组蛋白标记 H3K9 的单甲基化水平,表明 p.图 2E )。同时,我们观察到与 pcDNA 对照相比,转染 WT ASXL3 降低了 H2AK119 泛素化水平,证实了 WT ASXL3 的组蛋白去泛素化功能。相反,具有p.F1460Lfs*5的ASXL3突变体的细胞的H2AK119泛素化水平与pcDNA对照相似,表明该突变体缺乏正常的组蛋白去泛素化功能(图2E)。
强迫症被假设是由神经递质途径的改变引起的,例如谷氨酸系统、多巴胺系统和血清素系统。因此,我们假设在本研究中,例如确定为染色质改性剂SETD5,KDM3B,ASXL3和FBL其可用表观遗传修饰调节神经递质表达。为了研究强迫症中染色质修饰剂和神经递质之间的表达调控,我们使用了 Jaffe等人进行的一项研究的脑基因表达数据。( 35),其中包含从强迫症病例和非精神病对照分离的死后大脑的背外侧前额叶皮层的表达谱。我们首先计算了强迫症病例和对照组中三种染色质修饰剂和神经递质之间的皮尔逊R s。然后,我们通过计算强迫症患者和对照组之间的绝对共表达差异(|ΔCo-exp| = |R OCD – R对照|)来测量共表达调节(ΔCo-exp )(图 3A和表 S5)。我们发现KDM3B和多巴胺基因之间的整体共表达在OCD 病例和对照之间显着改变(Wilcoxon 检验P = 0.025)。相比之下,两者之间的共表达ASXL3和谷氨酸以及在ASXL3(Wilcoxon 检验P = 0.047)和血清素之间被轻微破坏(Wilcoxon 检验P = 0.045;图 3A)。
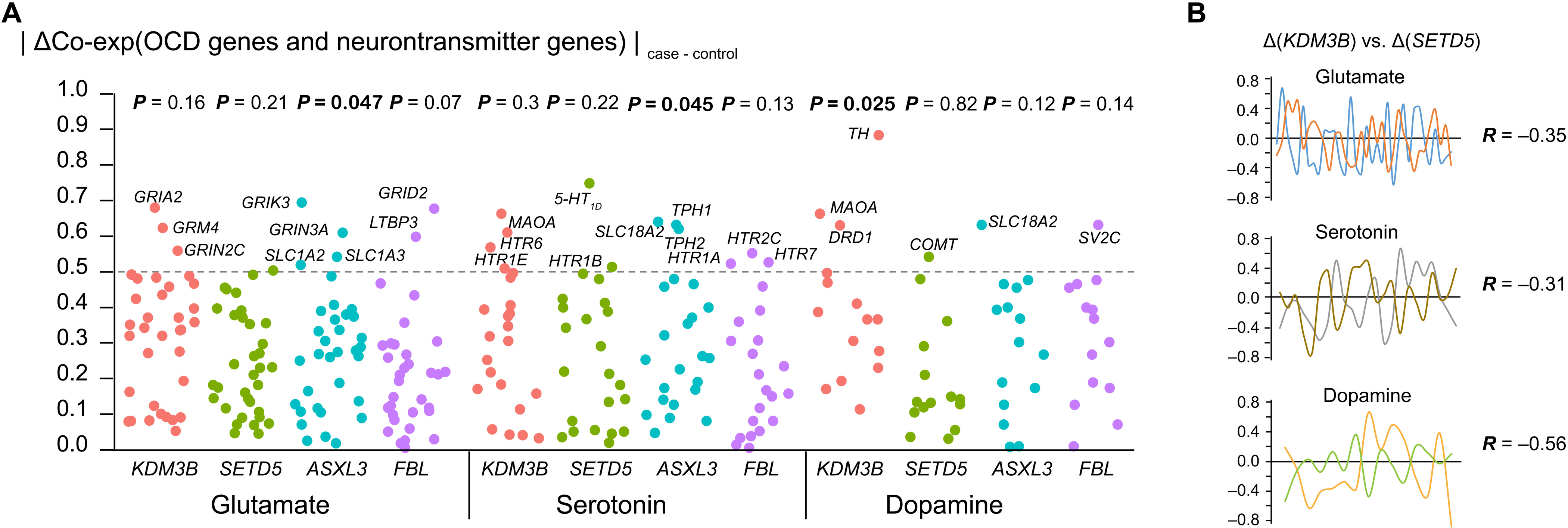
图 3。关键从头基因之间的共表达中断。
( A )强迫症患者前额叶皮层中FBL、SETD5、KDM3B和ASXL3与神经递质(谷氨酸、血清素和多巴胺)相关基因的共表达。每个点代表一个参与一个神经递质系统的基因,Y轴显示患者和对照之间共表达 (ΔCo-exp) 的绝对差异。使用 Fisher 检验计算P值并指示 ΔCo-exp 是否显着大于预期。( B ) 由KDM3B和SETD5引起的中断具有相反的趋势。对于每个神经递质系统,ΔR将SETD5和KDM3B的值相互作图,并计算它们的相关性。
通过检查对照组和强迫症病例之间共表达变化最大的神经递质,我们观察到参与酪氨酸转化为多巴胺的酪氨酸羟化酶 ( TH ) 基因受到KDM3B 的破坏最高(ΔKDM3B = 0.89,来自R = 0.22 控制到R = -0.66 强迫症,P = 0.0065;图 3A和表 S5)。还发现参与血清素和多巴胺代谢的MAOA基因在强迫症患者中被KDM3B 破坏(Δ KDM3B = 0.66,从R = -0.07 控制到R= 0.59 强迫症,P = 0.06)。之间的共表达SETD5和血清素基因HTR1D,也称为5-HT 1D,被显著改变(Δ SETD5 = 0.75从? =对照0.35?- [R = -0.4在OCD,P = 0.002)。FBL与调节神经和内分泌细胞分泌的多巴胺基因SV2C之间的共表达发生了显着变化(Δ FBL = 0.76,从对照中的R = -0.23 到OCD 中的R = 0.53)。
由于SETD5甲基化组蛋白标记H3K9,而KDM3B使组蛋白标记H3K9去甲基化(图 2D),我们假设SETD5和KDM3B可能以不同方式调节相同神经递质的表达。正如预期的那样,我们在谷氨酸、血清素和多巴胺系统中观察到ΔCo-exp SETD5和 ΔCo-exp KDM3之间的负R s 分别为R = -0.35、-0.31 和 - 0.56(图 3B)。与此相反,ASXL3,这促进组蛋白标记H2A119,不同的组蛋白标记的去泛素化,不与任一相关SETD5或任何神经递质调节系统中的KDM3B。
OCD 和 TD 表现出许多相似之处,包括遗传学、表型和流行病学 ( 36 )。因此,为了进一步了解 OCD 的复杂遗传学,我们通过分析罕见的 DNM(补充材料和方法)来比较和对比 OCD 和 TD。通过直接比较 OCD 和 TD 之间的重叠基因,结合当前研究和已发表的外显子组数据 ( 10 , 11 , 18 – 20 ),我们观察到这些疾病之间的基因重叠比预期的要多(所有外显子 DNM:P = 1.21 × 10 -38;LoF DNM:P = 9.19 × 10 -18;错义 DNM:P = 1.01 × 10 -34) (图 4A )。接下来,我们研究了 15 个疾病相关基因组(补充材料和方法)中 OCD 和 TD 基因的富集是否也相似。我们通过比较 log 10转化的富集 OR(图 4B)观察到两种疾病的富集之间存在强正相关(Pearson R = 0.54,P = 0.01 ),表明两者之间共享许多功能性生物学途径障碍。

图 4。受OCD和TDA编码突变影响的基因比较。
(一)维恩图显示了受 TD DNM 影响的基因(来自三项研究)和受强迫症 DNM 影响的基因之间的关系 [来自 Cappi等人的DNM 。( 10 , 11 ) 和当前的研究]。( B ) TD DNM 和受强迫症 DNM 影响的基因的基因集富集之间的相关性,这不是由功能基因集之间的相似性或依赖性引入的(平均 Jaccard 相似性指数 = 0.025)。每个点代表 15 个手动策划的基因组之一(补充材料和方法)。在X轴代表该基因组内的或TD的DNM基因富集,而?轴表示的OCD DNM-受影响的基因。Rs 和P值使用 Pearson 相关分析计算。(C和D)TD和OCD基因在(C)脑区和(D)脑细胞类型中的不同表达模式。红线表示标称P值阈值 (0.05)。TC,颞叶皮质;STR,纹状体;PFC,前额叶皮层;PC,顶叶皮层;OC,枕脑壁;MD,丘脑中背核;髋关节,海马;DTH,背侧丘脑;CB,小脑皮质;AMY,杏仁核。
由于强迫症中很大一部分 (75%) 的 DNM 基因不与 TD 共享,我们假设这种差异可能阐明可能参与强迫症发病机制的特定发育时间、大脑区域或神经元细胞。使用大脑发育期和大脑区域的BrainSpan 数据 ( 33 ),我们观察到 OCD 突变基因富集在背侧丘脑 [EWCE ( 37 ) P = 0.034],而 TD 突变基因富集在枕叶皮质 ( P = 0.039;图 4C )。根据 Dronc ( 38 ) 对成人脑细胞类型数据进行的一项研究,发现 OCD DNM 基因富含星形胶质细胞 ( P= 0.023),而发现 TD 突变基因在 GABA 能神经元中富集(P = 0.006;图 3D)。根据大脑发育时期没有观察到富集差异。这些结果表明,有明确、不同的发展模式将这两种疾病区分开来。
本研究出于对识别神经精神疾病发病机制背后的超罕见、潜在的高度外显性遗传变异的兴趣日益增长 ( 14 , 15 , 17),,我们首次通过WGS进行全基因组 DNM 分析描述了患有强迫症的家族。我们确定了 4147 个全基因组 DNM,其中 57 个影响先证者的外显子区域。通过探索强迫症先证者中 DNM 的全球负担,我们观察到在启动子锚定的染色质环和组蛋白标记频率高的区域发生的突变比例高于预期,例如 H3K9me3、H4K20me3 和 H3K36me3(染色质状态8)。在检查受 OCD 突变直接影响的基因的特性时,我们观察到 DNM 基因比 SSC 对照明显更不耐受突变,这与 DNM 可能是 OCD 的重要危险因素的假设一致 ( 10 , 11 )。
使用来自发育中的人类大脑多个区域的表达数据,我们发现在我们的强迫症全基因组筛查中鉴定的 DNM 基因分别在产前和产后发育中显示出高度相关的基因表达模式。产前阶段共表达网络包括一个单一的富集模块 M16,与组蛋白修饰和神经元和突触组织相关,而产后阶段包括一个单一的富集模块 M11,主要突出染色质修饰。这些来自两个连续阶段的丰富模块都将染色质调节确定为相关的顶级途径,强调了其在强迫症中的重要性。这些结果还表明,由于 M16 具有其他复杂的神经元功能,因此在产前发育过程中,额外的功能活动参与了强迫症的发展,
染色质修饰基因与各种神经发育障碍的风险相关 ( 39 , 40 )。在本研究中,我们观察到三个在 OCD 中具有最高循证排名的基因(SETD5、KDM3B和ASXL3)和一个被破坏性 dSV ( FBL )破坏的基因。这些基因主要是染色质修饰剂,并受到从头破坏性突变的影响,尽管它们对蛋白质改变变体具有极高的不耐受性。SETD5和KDM3B都调节H3K9组蛋白甲基化,这对异染色质维持至关重要(41)。这与我们的观察结果一致,即 OCD 患者在染色质区域(染色质状态 8)中含有更多的突变,其中包含高频率的 H3K9 组蛋白标记(42)。在低频中的突变SETD5已经在ASD(重牵连39)和ID(40)。SETD5 单倍体不足的小鼠模型表现出认知和记忆受损以及行为不灵活 ( 43 ),这表明可能与 OCD 临床表型相似。KDM3B是一组重要的组蛋白赖氨酸甲基化酶 (KMT) 和组蛋白赖氨酸去甲基化酶 (KDM) 的一部分,它们参与基因调控和表达。KDM3B从头和遗传的致病性变异可导致以 ID、身材矮小和面部畸形为特征的综合征 ( 44 )。所述ASXL3基因(OMIM?号:615485)与斑布里奇-Ropers综合征,发育障碍,其特征在于延迟精神运动发育和严重的ID(关联45)。该基因编码一种多梳组蛋白,它是 PR-DUB 复合物的一部分,具有去泛素化 H2AUb1 的功能(45)。因此,先前的遗传学研究已经通过不同的表观遗传调控机制确定了它们与各种其他精神障碍的重要联系。FBL通过介导组蛋白 H2A (H2AQ104me)的“Gln 105 ”甲基化而充当蛋白质甲基转移酶,这种修饰会损害 FACT 复合物的结合 ( 29 )。它在人脑中的 mRNA 和蛋白质中表达 ( 46 ),pLI 突变不耐受评分 > 0.95。尽管FBL与精神疾病之间没有直接联系,但之前的研究已将FBL 的SNP与表型联系起来,例如尼古丁代谢物比率(47),这表明它可能是研究具有成瘾行为的疾病的潜在候选者。
Cappi等人的外显子组研究。( 11 ) 将染色质重塑基因CHD8(编码染色质解旋酶 DNA 结合蛋白 8)鉴定为 OCD 和多种其他精神疾病的风险基因 ( 48 , 49 )。尽管由于复杂疾病的多基因性质和我们的样本量,我们在研究中没有在CHD8中发现 DNM,但已发现SETD5和KDM3B都是CHD8 的结合靶标( 49 )。因此,这证实了染色质调节参与强迫症发病机制的概念。
强迫症的病理生理学与皮质-纹状体-丘脑-皮质 (CSTC) 电路的异常以及该网络中谷氨酸、血清素和多巴胺系统的失调有关。最近,Pauls等人提出了一种整合了电路、神经化学和遗传/表观遗传元素的 OCD 模型。( 4),这表明具有遗传风险的强迫症患者可能容易受到环境因素的影响,这些环境因素可能通过表观遗传机制触发谷氨酸、血清素和多巴胺系统相关基因的表达修饰。我们的分析表明,在强迫症患者的前额叶皮层中,三个系统基因和染色质修饰剂之间的共表达模式发生了显着改变。这些结果表明,涉及SETD5、KDM3B、ASXL3和FBL 的染色质修饰可能是控制必要的神经认知功能的神经递质系统表达的上游调节剂。这种级联的任何部分的中断都可能导致异常的强迫表型。
尽管在当前研究中只有 53 个亲子家庭,但我们的研究结果中最一致的观察之一是染色质修饰功能的丰富,这表明 OCD 的表观遗传调控具有高度显着、有力的证据,并表明 OCD 存在潜在的表观基因组脆弱性。因此,尽管突触蛋白通常被认为与强迫症的发病机制有关(8),我们的研究结果强调了超出突触的其他贡献机制的存在。尽管需要进一步的大规模研究来证明 DNM 在 OCD 中的表观遗传调控的致病作用,但鉴于本研究中的中等样本量和统计学意义,我们的观察结果是可信的,考虑到 ASD 也有类似的发现。和 SCZ,以及CHD8在最近的强迫症外显子组研究中被确定为最重要的候选风险,并且也是染色质重塑基因。
最后,我们的研究结果提供了证据,表明强迫症与 TD 有很强的遗传病因,尽管在这两种神经精神疾病中,转录模式因大脑区域和神经元细胞类型而异。对于 TD 和 OCD 基因,在神经发育基因和产前期也观察到类似的富集,这与神经递质代谢 ( 50 ) 和症状的类似异常一致。我们的研究结果进一步支持这两种疾病都是发育障碍(11)。然而,强迫症基因在背侧丘脑中表现出独特的高表达,这与强迫症病理模型中的 CSTC 回路一致。TS和OCD之间的这种趋同和分歧可以从多基因风险的角度来解释。鉴于这两种疾病存在广泛的遗传和表型异质性,并且在本研究中仅检查了 DNM,我们的研究结果可能仅代表这两种疾病的收敛和/或发散方面的一小部分。尽管如此,我们的分析方法为理解 OCD 和 TD 之间潜在的合并症提供了重要的一步。
根据全血基因组 DNA 的可用性和表型信息的完整性,我们收集了 53 个不相关的亲子家庭,包括 160 个个体。亲子家庭由 52 个三人组(每个家庭有一个受强迫症影响的后代,总共 156 个样本)和一个有两个受强迫症影响的后代的家庭(总共四个样本)组成。表 S1 中提供了每个参与者的详细信息。所有受强迫症影响的患者均符合以下条件:(i)根据精神疾病诊断和统计手册第四版(DSM-IV)(51),患者被诊断为患有强迫症。) 标准。(ii) 年龄在 18 至 65 岁之间,并且 (iii) 耶鲁-布朗强迫症量表总分截止值为 16。如果患者 (i) 包括除 DSM-IV 标准以外的其他疾病,则将其排除在外强迫症,(ii) 有中度至重度的自杀意念,(iii) 是怀孕或哺乳期的女性。本研究排除了被诊断患有 SCZ、分裂情感障碍、ASD、未另行指定的普遍性发育障碍或 ID 的患者。通过使用 Mini International Neuropsychiatric Interview ( 52),并且排除了具有任何 DSM-IV Axis I 精神病学诊断的父母的家庭。经上海市精神卫生中心机构审查委员会批准,所有参与者均获得知情同意。
从全血或淋巴母细胞来源的细胞系中提取的基因组 DNA 通过 PicoGreen 和凝胶电泳评估质量,然后由 Novogene(Novogene Biosciences Inc.,北京,中国)测序。DNA 数量通过 Qubit 3.0 测量,至少 1 μg 未降解的基因组 DNA 用于基因组文库制备和 WGS。我们在 Illumina HiSeq 4000 测序平台(150 bp 双末端读数)上对之前从未测序过的所有三重样本进行了测序。使用处理流程,所有从头变体都被确定为高质量,然后使用 Integrative Genomics Viewer (IGV) 通过对齐读数的可视化手动检查 ( 53 )。使用 wANNOVAR ( 30)。我们的 WGS 数据的平均覆盖深度为 29.9 倍,平均对齐率为 99.87%。在一个三重奏被排除在质量控制之外后,剩下的 53 个三重奏被提交进行后续分析。
使用 Sentieon DNAseq ( 54 ) 处理管道,使用 BWA-mem ( 55 ) v0.7.15-r1140将读数与 GRCh37.63 人类基因组参考构建对齐,并通过 Sentieon Dedup 进行排序。然后,重复读取被 Sentieon LocusCollector 标记并删除。使用 Sentieon Realigner 和 QualCal 执行 indel 重新校准和基础质量评分重新校准。接下来,我们使用 SAMtools(版本 1.3.1)flagstat(56)和 Picard 的 WGA 指标(57)(版本 2.5.0)对 BAM 文件进行了质量检查。
使用两种算法的组合调用从头 SNV 和插入缺失,SAMtools bcftools(版本 1.3.1)(56)和贝叶斯框架 TrioDeNovo(58),每个家庭的默认设置。根据以下阈值过滤假定的从头变体:(i)基因型质量≥30;(ii) 先证者和父母双方的最小测序深度为 15 个读取点;(iii) 测绘质量≥30;(iv) 等位基因平衡 (AB) < 0.05 的父亲和母亲纯合子;(v) AB 介于 0.3 和 0.7 之间的先证者是杂合子;(vi) TrioDeNovo 的从头质量评分≥7;(vii) 与标注为节段重复的已知区域不重叠;(viii) 1000 基因组计划中的次要等位基因频率 ≤ 5.0 × 10 -3 ( 59)、ExAC ( 31 )、EVS ( 60 ) 和 gnomAD ( 61 ) 数据库;(ix) 在 50 bp 距离内聚集的变体已被删除。最后,在 silico BLAT 搜索 ( 62 ) 中使用来自家族的比对读数检测所有从头变体,然后使用 IGV 通过比对读数的可视化手动检查 ( 53 )。
变体由四个不同的注释组注释如下:(1)变体类型:每个变体使用 wANNOVAR(30)按突变类型分类,包括 SNV 和 indel(<50 bp),例如移码、停止增益(废话)、非同义SNV(错义)和同义SNV等(2)基因组位置注释:变体的基因组位置由wannovar(30)注释并分配以下优先级:编码、内含子、UTR、上游、下游、ncRNA和基因间。(3) 基因集:与大脑发育或神经精神疾病相关的基因列表选择如下: (3.1) CHD8靶基因定义为来自两个染色质免疫沉淀测序研究的列表的联合 ( 49 , 63)。(3.2) FMRP靶基因被定义为来自 Darnell等人的列表的联合。( 64 ) 和阿斯卡诺等人。(65)。(3.3) RBFOX剪接目标选自Weyn -Vanhentenryck 等人。(66)。(3.4) 从 Genes2Cognition 数据库 ( 67 )中提取人类突触后密度蛋白。(3.5) 受限基因在 ExAC 数据库中被定义为 pLI 评分 ≥ 0.9 ( 31 )。(3.6) 编码染色质修饰物的基因从 Chen等人下载。( 68)。(3.7) 神经发育障碍风险基因来自 Stessman等人。(69)。
已经使用四种类型的方法来检测 SV:Manta ( 70 )、cn.MOPS ( 71 )、DELLY ( 72 ) 和 LUMPY ( 73 )。我们应用了 SV2 ( 74) 将 50 bp 到 10 Mb 内的 SV 合并到每个家族的 VCF 文件中,并选择 FILTER 标记和 DENOVO_FILTER 标记中都带有“PASS”的 SV 作为候选 dSV。此外,我们对最终 dSV 执行不同的过滤条件如下:对于男性患者,我们筛选常染色体和 X 染色体上未受影响的父母中不存在或 Y 染色体上未受影响的父亲中不存在的纯合 SV。对于女性患者,我们筛选杂合/纯合 SV,在常染色体和 X 染色体上未受影响的父母中不存在这些 SV。由 SV2 检测到的潜在 dSV 通过两个程序进一步过滤。首先,我们与至少两名专家检查了相应三人组中每个 dSV 的 IGV,并删除了没有相应剂量变化的那些。其次,我们浏览了 GnomAD 数据库并删除了所有曾经出现过的 dSV(即,在非疾病汉族人群中,超过 50% 的 dSV 长度被来自 GnomAD 的同类型 SV 覆盖。最后,我们通过 qPCR 验证了候选 dSV 的存在。
为了计算全基因组和外显子组的突变率,我们首先从 UCSC 基因组浏览器下载了 hg19 的所有外显子和 UTR 区域的范围,并计算了整个编码区域中超过 20 倍覆盖率的碱基对总数基因组。然后,我们使用R 中的“ t.test ”函数估计了所有 53 名 OCD 患者在 95% 置信区间内的平均突变率。为了确定 OCD 患者和健康个体之间 DNM 分布的差异,我们纳入了来自 SSC 兄弟姐妹的 DNM(15) 用作控件。由于研究之间检测到的 DNM 数量略有不同,我们应用 Fisher 精确检验来评估 OR,该 OR 测量健康对照组与强迫症患者相比,DNM 的相对比率。然后,我们根据 Annovar ( 30 ) 注释划分所有编码突变:我们将移码插入缺失或起始位点/停止位点/剪接位点突变定义为 LoF 突变,将非同义 SNV 定义为错义突变。
我们比较了来自 An等人的OCD 患者和 SSC 兄弟姐妹之间的 CADD-phred 评分。( 15 ) 外显子和基因组突变。为了评估基因不耐受,我们收集了受强迫症患者不同类型编码 DNM(LoF、错义、破坏性和同义词)影响的基因的pLI ( 31 ) 评分。磷通过双边 Wilcoxon 秩和检验计算值。由于 CADD-phred 对应于突变严重程度的百分等级并且不遵循正态分布,我们应用累积分布曲线来可视化 CADD-phred 评分的分布和 Wilcoxon 秩和检验来比较预测的严重程度。为了评估 GC 偏差对不同基因组区域的影响,我们比较了 OCD 数据和 SSC 对照中 DNM 之间的 GC 含量和基因长度。GC含量和基因长度的数据来自GenBank。P值通过双边Wilcoxon秩和检验计算。
我们将强迫症患者的结果与从 PsyMuKB 数据库中获得的其他神经精神疾病(发育迟缓、ID、SCZ 和 ASD)的结果进行了比较 ( 46 )。由于在其中一些研究中无法获得单个个体的详细信息,我们对通过同义突变调整的突变计数应用了简单的 Fisher 检验。
为了研究突变的功能基因组分布,我们首先从 ENSEMBL ( 25 ) 和 PsychENCODE ( 24 ) 中获得了人类基因组功能区域的四个定义。): (i) 全长基因:我们从 biomaRt 中获得了所有编码基因的范围,条目为“start_position”和“end_position”。(ii) 启动子:对于 (i) 中获得的所有基因,我们找到了它们最长的转录本和相应的转录起始位点 (TSS)。我们将启动子区域定义为 TSS 上游 2-kb 和下游 1-kb。(iii) 启动子交互区域:我们从 PsychENCODE 资源页面下载启动子锚定循环文件,该文件是从人类前额叶皮层的 HI-C 数据中获得的。(iv) 增强子:从 PsychENCODE 资源页面,我们下载了使用前额叶皮层 HI-C 数据生成的增强子-基因交互文件,并提取了所有增强子区域。我们还从 Roadmap 项目(26), 由 chromHMM ( 75 )计算得出。
为了验证突变分布富集不是由随机效应驱动的,我们对阳性结果(启动子环和染色质状态 8)应用了排列测试。首先,我们随机选择了 53 个对照并重复了 Fisher 检验。此过程重复 10,000 次,并且使用 OR 的分布来生成OR = 1的P值。最后,我们删除了一条染色体上的所有突变,并对剩余的突变重复 Fisher 检验。对 22 条常染色体中的每一条重复该过程。
BrainSpan RNA 测序数据 ( 33 ) 应用于以下共表达网络分析。我们通过产前和产后样本将表达谱分为两个不同的时期集,包括所有大脑区域。对于每个子集,仅保留至少一半样本中每千碱基每百万映射读数 (RPKM) >0 且方差系数 >0.3 的基因用于 WGCNA ( 34 ) 分析。剩余的数据经过 log 10 转换。产前子集的软阈值设置为 12,产后子集设置为 16。最小模块大小设置为 20。我们通过基于 Pearson R的 WGCNA R 包中的 blockwiseModules 函数构建签名网络并将基因划分为树切割高度为 0.3 的模块。在检测到两个子集中的共表达模块后,我们通过 Fisher 精确检验测试了携带 OCD 编码突变的基因是否在某些模块中富集。然后将富集的模块用于基因本体富集和网络分析。
为了探索四个关键基因(SETD5、KDM3B、FBL 和ASXL3)和三个神经递质系统(谷氨酸、血清素和多巴胺)之间的潜在失调,我们首先计算了关键基因和前额叶皮层中所有其他基因之间的Pearson's R Jaffe等人的表达数据。( 35)。我们使用双边 Wilcoxon 秩和检验来检查染色质修饰剂和神经递质之间的整体共表达在强迫症患者和对照组之间是否显着。为了查看是否有任何单个神经递质基因显着失调,我们比较了|ΔCo-exp| 每个基因对|ΔCo-exp|的整体分布 相应的关键基因,并产生一个名义上的P值。
SETD5-FLAG 和 ASXL3-FLAG 表达质粒购自友泽生物科技有限公司(中国湖南),使用 Q5 定点诱变试剂盒(New England BioLabs)引入 SETD5 R77C 和 ASXL3 F1460Lfs*5 突变质粒)。所有质粒均通过 Sanger 测序确认。
HEK293T 细胞在冰上用 1× 裂解缓冲液裂解 30 分钟,然后以 12,000 rpm 的转速离心 15 分钟。通过在 4% 到 20% SDS-聚丙烯酰胺凝胶电泳凝胶上电泳分离细胞裂解物,然后转移到硝酸纤维素膜上。用含 5% 牛奶的 TBST(Tris 缓冲盐水,0.1% Tween? 20)封闭膜 1 小时,并在 4°C 下与相应的一抗孵育过夜:兔抗-FLAG 和抗-β-微管蛋白(Sigma -Aldrich)、兔抗 H3K9me1 (Abcam) 和兔抗 H2AK119ub (Cell Signaling Technology)。接下来,在室温 20° 至 25°C 下,与辣根过氧化物酶标记的山羊抗兔免疫球蛋白 G (Beyotime) 一起孵育 1 小时。然后使用 ImageJ 软件通过光密度分析进行免疫印迹的定量。
我们从 psyMuKB 数据库 ( 46 )下载了 OCD 和 TD 的公开可用的 DNM 数据。本数据集中共有两项强迫症研究 ( 10 , 11 ) 和三项 TD 研究 ( 18 – 20 )。记录了具有至少一个错义或 LoF 突变的所有基因。我们将我们的非同义突变基因与已发表的 OCD DNM 基因相结合,并将它们与所有以前的 TD DNM 基因进行比较。
为了分析 TD 和 OCD 基因的重叠,我们使用背景基因列表作为所有蛋白质编码基因进行了超几何测试。计算双尾检验的P值。为了分析 TD 和 OCD 基因的功能特征,我们首先从 psyMuKB ( 46 ) 中获得了 15 个基因组的列表,主要是关于中枢神经系统的。我们通过 Fisher 精确检验测试了所有这些基因集中的富集 TD/OCD 基因。然后,我们对来自两种疾病的基因的-log 10 (OR)进行了 Pearson 相关分析。为了分析 TD 和 OCD 基因的时空和细胞类型特异性表达模式,我们应用了 EWCE R 包(37),一个基于基因细胞类型特异性矩阵的引导富集工具,对两个数据集进行富集分析:(i)Brainspan(33)用于周期和区域分析,(ii)Dronc 数据(38)用于成人脑细胞类型分析。特异性在相应的论文中定义(37)。我们首先通过“generate.celltype.data”函数计算了两个表达数据集的特异性矩阵。接下来,通过“bootstrap.enrichment.test”函数测试所有 TD/OCD 基因的富集。我们将背景基因列表定义为在测试表达数据集中注释的所有基因。