染色质开放性是指真核生物染色质DNA和转录因子等蛋白能否结合的开放程度,这一特性反映了染色质转录活跃程度等信息。2013 年由美国斯坦福大学William Greenleaf 教授报道了ATAC-seq(Assay of Transposase Accessible Chro-matin with High-throughput sequencing),即染色质开放性检测,准确率与传统方法一致,且重复性更高,只需少量细胞/ 组织,就能完成高质量的测序,是目前研究染色质开放性的最优策略。
 技术原理 技术原理
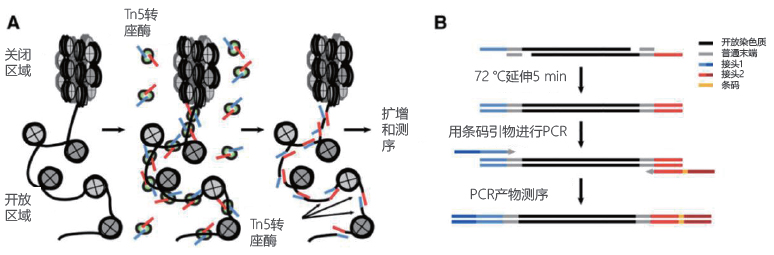
图1. ATAC-seq技术原理及实验流程[1]
 技术应用 技术应用
| |
1. 初步判断所研究的课题是否涉及表观遗传调控机制
2. 结合基序分析,识别参与基因调控的转录因子
3. 识别转录因子调控的靶基因和功能元件
4. 与超级增强子鉴定联合分析,明确活性超级增强子 的范围
5. 更好地了解药物或疾病的基因调控和细胞应答机制
6. 识别驱动细胞命运、疾病或应答相关的转录因子 |
|
 分析内容 分析内容
| |
1. 基因组比对
2. 基因组富集区域分析
2.1 富集区域鉴定
2.2 富集区域分布特征(Heatmap、metaProfile)
2.3 Peak相关基因GO分析
2.4 Peak相关基因KEGG分析
2.5 差异Peak分析
2.6 差异Peak相关基因GO,KEGG分析
3. 富集区域motif分析 |
|
 技术优势 技术优势
| |
1. 灵敏性高,低细胞起始量,每个样本50,000 个细胞即可
2. 实验步骤简化,实验重复性好,成功率高
3. 能同时揭示开放染色质的基因组位置、DNA 结合蛋白和转录结合位点的相互作用 |
|
 送样要求 送样要求
| |
样本类型:
1. 活细胞样本,50,000 个细胞每样本
2. 组织样本,质量≥ 100mg
样本物种:仅限人、大小鼠,其他物种需评估 |
|
 表观生物实测结果 表观生物实测结果
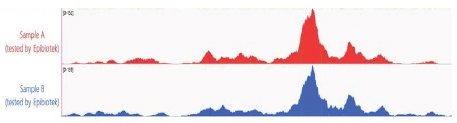
图1. ATAC-seq读长覆盖图,显示ATAC peaks
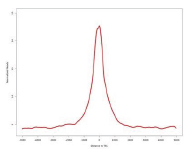 |
图2. A549细胞ATAC-seq Reads在TSS上的分布图
|
|
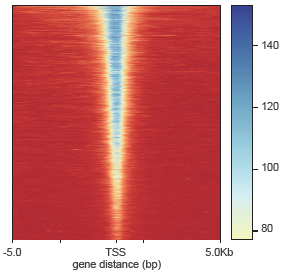
图3. TSS分布热图 |
 案例:PNAS: 肠上皮内淋巴细胞的微生物对增强子开放性的影响[1] 案例:PNAS: 肠上皮内淋巴细胞的微生物对增强子开放性的影响[1]
本项研究对小鼠肠道 αβ + 和 γ δ + 上皮内淋巴细胞进行了ATAC-seq,再通过鉴定普通增强子和超级增强子靶基因,揭示了被微生物影响的信号通路网络、代谢通路和增强子相关转录因子。这项研究的结果证明研究遗传修饰有助于鉴定宿主免疫细胞系中的微生物群落相关的功能特征。
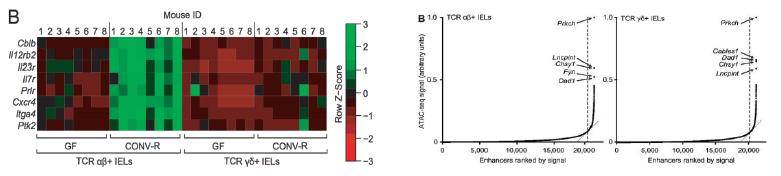 |
| ATAC-seq 信号样本间差异热图 |
|
ATAC-seq 信号可预测普通增强子和超级增强子 |
热图显示来自 JAK-STAT 元件和白细胞跨膜迁移信号传导通路的 ATAC-seq 信号差异,CONV-R TCR αβ + IELs 开放性增加与增强子相关。 |
|
指数曲线显示,最强的 ATAC-seq 信号发生在少数增强子上。虽然超级增强子只由 <5% 的 IEL 增强子组成,但 ATAC-seq 信号比普通增强子多。这个情况在其他不同组织的研究中也存在,ATAC-seq、靶向 p300 和 Mediator 的 ChIP-seq 有着类似的分布情况。 |
 参考文献 参考文献
[1] Semenkovich N P, Planer J D, Ahern P P, et al. Impact of the gut microbiota on enhancer accessibility in gut intraepithelial lymphocytes[J]. Proc Natl Acad Sci U S A, 2016, 113(51):14805~14810.
|






