
-

生物通官微
陪你抓住生命科技
跳动的脉搏
单细胞 RT-PCR 实验范例[创新技巧]
【字体: 大 中 小 】 时间:2003年12月26日 来源:
编辑推荐:
Martin Simon, Petra Albrecht & Helmut J. Schmidt, University of Kaiserslautern, Germany, Department of Ecology
Andrés Jarrin, Eppendorf AG
简介
本实验借助eppendorf cMaster RTplus PCR系统,首次通过检测mRNA的存在证实单细胞草履虫Paramecium primaurelia的两种表面抗原的同时表达。这个实验第一次在RNA水平上证实了表面抗原互斥表达(mutual exclusion expression)的一个例外。这个实验是通过两步法多重RT-PCR来实现的。
草履虫是单细胞的纤毛虫,其细胞表面包被了多种高分子量的,被称为固定抗原(immobilization antigens,iAg)的蛋白——这样命名的原因是由于某些针对这些表面蛋白的抗血清可以完全阻止草履虫的运动。各种草履虫都可以表达一系列的这种蛋白,在Paramecium tetraurelia 中发现了12种,在Paramecium primaurelia发现了3种。这些蛋白由不同的基因编码,但是都服从“互斥表达,mutual exclusion expression”的规律,就是说在同一个培养体系中所有的草履虫都表达同一个固定抗原(iAg),从而确定其血清型。通过改变其环境,例如温度,可以将草履虫从一种血清型变成另一种。
已知编码P. primaurelia Strain 156中固定抗原蛋白G和D的核酸序列,实验人员设计了两对引物,分别对应蛋白G和D,这样就可以通过RT-PCR的方法监测iAg的转录活性。
方法
mRNA的分离纯化和RT-PCR全过程都是在同一天完成的,所以中间没有间断或者是保存样品。所有的实验器皿都经过RNase无酶化处理并采用无RNase级别纯度的消化品。
单细胞P. primaurelia mRNA的分离纯化
单细胞在20ul 裂解buffer中反复吹打进行裂解,加入生物素标记的oligo dT探针,37度温育10分钟,使mRNA和Oligo dT充分杂交。裂解液经过streptavidin包被的小管并温育,生物素标记的Oligo-dT结合到streptavidin上,经过3次洗涤尽量除去DNA污染。
RT-PCR
一步法RT-PCR或者两步法RT-PCR都是直接在包被固定的管中进行反应。
实验中采用3对引物,分别是:Serotype G引物(产物约800bp),Serotype D(产物约500bp)和一对non-transcribedgenomic region对照引物(产物400bp),每个实验都双份平行同时进行。
One-step RT-PCR
| Components | Volume | Final concentration in the reaction |
| dNTP mix (each 10 mM) | 1.0 µl | 200 µM |
| Forward primer G (100 µM) | 1.0 µl | 2 µM |
| Reverse primer D (100 µM) | 1.0 µl | 2 µM |
| RTplus PCR buffer with 25mM Mg2+ | 5.0 µl | 1 x |
| RNase Inhibitor (0.5 µg/µl) | 0.5 µl | 5 ng/µl |
| cMaster RT enzyme (15 U/µl) | 0.5 µl | 0.15 U/µl |
| cMaster PCR enzyme mix (5 U/µl) | 0.5 µl | 0.05 U/µl |
| RNase free water | 40.5 µl |
Total volume 50.0 µl
Program parameters of the one-step RT-PCR
| Step | Temp. | Duration | |
| cDNA synthesis | 50 °C | 50 min | |
| Initial denaturation | 94 °C | 2 min | |
| Denaturation | 94 °C | 20 s | x 40 cycles |
| Annealing | 45 °C | 20 s | |
| Elongation | 68 °C | 30 s | |
| Cooling | 4 °C | Hold |
Two-step RT-PCR
Gene-specific primers were used for the cDNA synthesis.
Reverse transcription preparation
| Components | Volume | Final concentration in the reaction |
| dNTP mix (each 10 mM) | 2.0 µl | 1 mM |
| Reverse primer G (100 µM) | 1.0 µl | 5 µM |
| Reverse primer D (100 µM) | 1.0 µl | 5 µM |
| Left control primer (100 µM) | 1.0 µl | 5 µM |
| RNase free water | 5.0 µl |
Master Mix 1 10.0 µl
| RTplusPCR buffer with 25 mM Mg2+ | 4.0 µl | 2 x |
| RNase Inhibitor (0.5 µg/µl) | 0.5 µl | 12.5 ng/µl |
| cMaster RT enzyme (15 U/µl) | 2.0 µl | 1.5 U/µl |
| RNase free water | 3.5 µl |
Master Mix 2 10.0 µl
Program parameters of the reverse transcription
| Step | Temp. | Duration |
| Template RNA denaturation | 65 °C | 5 min |
| Fixation of the RNA | 0 °C | 5 min |
| Addition of Master mix 2 | (10 µl) | |
| Primer annealing | 45 °C | 10 min |
| cDNA synthesis | 50 °C | 50 min |
Preparation of the PCR
| Components | Volume | Final concentration in the reaction |
| dNTP mix (each 10 mM) | 2.0 µl | 400 µM |
| Forward primer D (100 µM) | 0.5 µl | 1 µM |
| Reverse primer D (100 µM) | 0.5 µl | 1 µM |
| Forward primer G (100 µM) | 0.5 µl | 1 µM |
| Reverse primer G (100 µM) | 0.5 µl | 1 µM |
| Right control primer (100 µM) | 0.5 µl | 1 µM |
| Left control primer (100 µM) | 0.5 µl | 1 µM |
| First-strand cDNA, see above | 6.0 µl | |
| Master Mix 3 | 11.0 µl |
| RTplusPCR buffer with 25 mM Mg2+ | 5.0 µl | 1 x |
| cMaster PCR enzyme mix (5 U/µl) | 0.5 µl | 0.05 U/µl |
| RNase free water | 33.5 µl | |
| Master Mix 4 | 39.0 µl |
结果
分别用独立的引物,对P. primaurelia单细胞不同的目标蛋白进行一步法RT-PCR扩增。结果显示采用eppendorf cMaster RTplusPCR system,血清型D和G都能够得到清晰的结果。扩增结果亮度的区别可以解释为代表草履虫体内mRNA转录本拷贝的区别。
采用两步法多重RT-PCR,可以看到在单细胞P. primaurelia中,蛋白G和D的转录本同时存在,其专一性可以有图2中证实:在cDNA中作为对照的非转录基因组片断没有扩增。
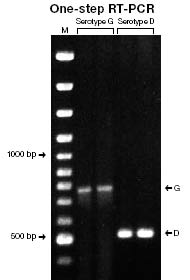
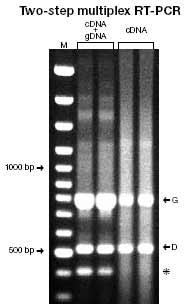
这些结果显示早前假设的单个草履虫细胞中这两个基因互斥表达至少有了一个例外。在RNA水平上的分析结果可以在蛋白水平上用免疫荧光染色的方法得到证实。这个实验结果表明,在极少量样品和低拷贝表达的情况下要求有同样灵敏度级别的试剂来检测,高灵敏度的eppendorf cMaster RTplusPCR system使得单细胞P. primaurelia的RT-PCR得以成功进行。采用其他的RT-PCR系统,包括AMV在内,都只能一种体形较大而且含有明显更多的RNA的草履虫P. tetraurelia中得到相应的结果,而 cMaster RTplus PCR system包含的经过改造的反转录酶由于有更高的灵敏度而得以成功在个体小而RNA少的P. primaurelia中得到结果。