|
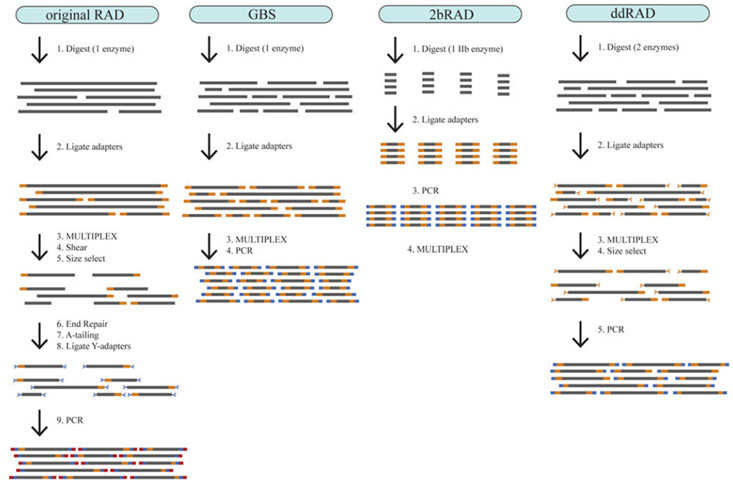
| 产品简介 简化基因组测序(Reduced-Representation Genome Sequencing,RRGS)是指利用限制性内切酶打断基因组DNA,对特定片段进行高通量测序获得海量遗传多态性标签序列来充分代表目标物种全基因组信息的测序策略。此方法实验步骤简单,成本低,而且可以不依赖参考基因组,就能获得全基因组范围内的遗传多态性标签,因而广泛应用于生态学,进化学和基因组学等领域。 主要的简化基因组技术比较 到目前为止,科学家已经开发出了多种简化基因组的技术。其中,使用得比较多的技术主要有四种:即经典的 RAD、经典的 GBS、2bRAD、 ddGBS(也就是ddRAD)。这四种方法的建库流程见下图。
下表比较了这四种方法的主要特点:
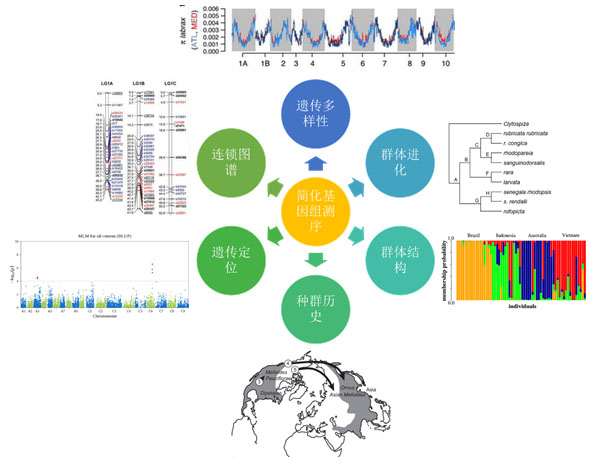
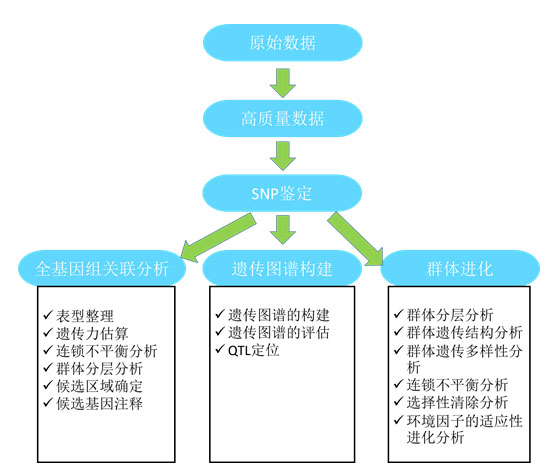
应用领域 简化基因组能获得全基因组范围内有代表性的遗传多样性位点,因而广泛应用于群体进化、群体结构、种群历史、遗传定位和连锁图谱的构建。
派森诺在简化基因组测序方面有丰富的经验,项目涵盖动植物20多个物种。
送样要求
常见问题 1. 简化基因组测序常用的指标有哪些? 主要有覆盖率和覆盖深度。覆盖率是指测序得到的碱基占全基因组的比例。覆盖深度是指单个碱基被测到的平均次数。覆盖率可以反映出分子标记是否有代表性,覆盖深度决定了分子标记的检测的准确度。 2. 有参和无参有什么差别? 有参考基因组的物种获得的SNP的准确性更高。 3. 分子标签的筛选有什么要求? 要求分子标签在全基因上均匀分布,在不同的个体之间有特异性等。同时,根据不同的研究目的,对分子标签的密度要求也有所不同。 |
|