7月20日,山东大学数学与交叉科学研究中心李国君教授团队在生物信息学顶级期刊Genome Research发表转录组重构重要研究成果“TransMeta simultaneously assembles multisample RNA-seq reads”。山东大学为论文第一完成单位,数学与交叉科学研究中心于婷博士与数学学院赵晓宇博士为论文共同第一作者,李国君教授为文章独立通讯作者。

RNA-seq测序技术为揭示和研究真核生物转录组的复杂结构提供了前所未有的机遇,基于RNA-seq数据精确重构转录本是转录组学开展后续分析的前提,为基因差异表达分析等下游研究起到铺垫作用,尤其是对包括癌症在内的复杂疾病的研究具有重大意义。然而如何从海量测序片段准确高效地重构出全长转录组,是目前面临的一个重大挑战。几乎所有转录组学研究都涉及多个样本的RNA测序,如何针对多样本的RNA测序数据创建一个一致的转录本集合也十分关键。目前,几乎所有的转录组组装算法都是针对单样本测序数据设计,专门针对多样本测序数据进行组装的工具却非常匮乏,而且其组装效果并不理想。
为此,李国君教授团队开发了一个全新的多样本转录组组装算法--TransMeta,实现了多个样本RAN-seq数据的同步精确组装,既可以为多样本测序生成一个一致的转录组,又可以同步地为每个独立样本生成一个特定的转录本集合。TransMeta算法引进了一个全新的图模型--向量加权剪接图模型(Vector Weighted Splicing Graph Model),区别于传统剪接图的标量加权,TransMeta算法首次提出了使用向量来对剪接图赋权的思想,其中向量的大小对应于要组装的样本个数,这合理地将转录组组装问题从一维的情况推广到了高维的情况,即从单样本组装推广到了多样本组装。基于向量加权剪接图模型,TransMeta算法着重考虑向量权之间的余弦相似度与双端测序信息,通过引进一个约束最优化问题实现了对剪接图的有效梳理,并结合一种基于动态规划的路径搜索策略来精确地重构转录本。
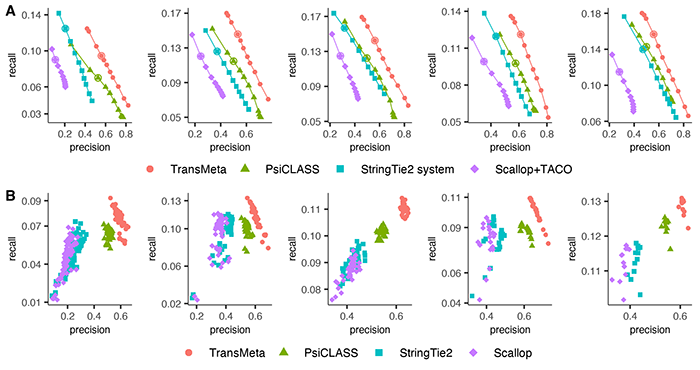
通过在多组包含不同样本个数的数据集(包括模拟数据与真实数据)上的测试,并与目前组装效果最好的组装算法,包括PsiCLASS,StringTie2,Scallop与TACO,进行比较, TransMeta算法在重构转录本的准确率与召回率上都有了明显的提高。在5组真实数据多样本组装层面的比较中,通过调整参数使得各个方法组装准确率相当时,TransMeta的召回率比表现次好的方法PsiCLASS高出了21%-57%。在单样本组装层面的比较中,TransMeta算法同样达到了最优,在包含73个样本的肝脏细胞测序数据上TransMeta的召回率比其他算法高出了19.6%-75.2%。
本项研究工作是转录组重构领域的一个重要突破,尤其是以向量来加权剪接图,并基于向量权设计组装算法,对相关领域的后续研究具有重要的推动作用。该项研究得到了国家自然科学基金重点项目的资助。

