
-

生物通官微
陪你抓住生命科技
跳动的脉搏
李轩研究组合作利用深度学习神经网络建立RNA的m6A修饰识别模型
【字体: 大 中 小 】 时间:2022年02月16日 来源:中国科学院分子植物科学卓越创新中心/植物生理生态研究所
编辑推荐:
2022年1月17日,中国科学院分子植物科学卓越创新中心李轩研究组、王佳伟研究组、中国科学院上海巴斯德研究所郝沛研究组合作,在国际学术期刊Genome Biology在线发表了题为“DENA: training an authentic neural network model using Nanopore sequencing data of Arabidopsis transcripts for detection and quantification of N6-methyladenosine on RNA”的研究成果
2022年1月17日,中国科学院分子植物科学卓越创新中心李轩研究组、王佳伟研究组、中国科学院上海巴斯德研究所郝沛研究组合作,在国际学术期刊Genome Biology在线发表了题为“DENA: training an authentic neural network model using Nanopore sequencing data of Arabidopsis transcripts for detection and quantification of N6-methyladenosine on RNA”的研究成果。 这项研究利用深度学习神经网络方法,训练构建了转录组RNA甲基化(m6A)修饰的识别量化模型DENA (Deeplearning Explore Nanopore m6A),并为从转录组直接测序研究RNA表观修饰的生物学功能提供了重要方法工具。
RNA转录后修饰是近年来研究RNA表观功能的新热点。RNA的N-6位甲基化(m6A)修饰,作为RNA最常见的修饰机制,参与了mRNA的剪接、运输、翻译、定位、及降解等一系列重要的分子过程。RNA的m6A修饰因受到多种机制调控而呈现显著动态变化,对其高精度定量检测一直是研究RNA表观功能的一个瓶颈。前人开发的MeRIP、PA-m6A-seq和miCLIP等各类实验方法,由于流程复杂、受RNA降解影响严重、及精度差等原因,在对RNA表观修饰研究中的应用受到限制。
Nanopore测序是目前唯一可以直接测定RNA序列的技术,为直接精准检测RNA的各种表观修饰提供了突破的可能性。RNA的Nanopore直接测序过程,当RNA碱基通过纳米孔时,其上的修饰结构会生成特异性的电流信号。理论上通过解析记录的电信号数据,可以识别碱基上的化学修饰结构。由于RNA修饰产生的电信号具有极高复杂度:以N-6位甲基化(m6A)修饰为例,对其信号识别和精确测量一直缺乏可靠的模型和算法。目前已经发现RNA上几百种不同复杂修饰结构,而每种修饰结构有不同的电信号特征,需要不同的识别模型和方法,所以建立可靠和高效率的RNA修饰识别模型和方法,是一项艰巨的挑战。
研究组针对RNA最普遍的N-6位甲基化(m6A)修饰结构,利用深度学习神经网络方法,建立转录组RNA甲基化(m6A)修饰的识别量化模型。研究组利用拟南芥材料,包括野生型和m6A修饰突变型,比较其mRNA的Nanopore直接测序数据,获得相同序列位点的m6A高修饰(野生型)和低修饰(m6A突变型)的信号特征。研究组国际上首次使用深度学习神经网络模型进行训练,建立了识别RNA甲基化(m6A)修饰结构的信号识别模型和分析工具(DENA)。研究的一些主要成果如下:
(1)首次从生物样品的RNA直接测序数据中,分离获得天然RNA的m6A修饰的信号特征,避免了人工合成RNA的测序数据信号噪音问题。神经网络模型训练则基于从3000多m6A修饰位点提取的上百万条测序数据特征,解决了人工合成RNA数据位点不足造成的训练复杂神经网络模型的困难。
(2)DENA是首个获得RNA的m6A修饰信号识别神经网络模型。对不同测试位点数据,获得的模型评估指标(AUC)在0.90 至 0.97之间, 精确度(accuracy)在0.83至0.93之间。DENA的开发基于转录组数据的分析要求,实现了分辨同一基因不同isoforms的m6A修饰功能。
(3)DENA不仅在拟南芥mRNA测序数据的m6A修饰位点预测有高精度,在对人细胞mRNA已知m6A修饰位点的检测和定量也获得了出色的结果(用SCARLET结果验证)。证明了DENA较好的鲁棒性,它不仅适用于拟南芥,也可用于其他生物物种的RNA修饰研究。
(4)利用DENA对野生型和三种m6A缺陷型拟南芥突变体的测序数据进行分析,首次建立了全转录组水平上、单碱基精度的拟南芥mRNA的m6A修饰图谱。
这项研究成果不仅为拟南芥和其他生物mRNA的m6A修饰研究提供了重要工具,也为开展RNA其他表观修饰的识别和分析,提供了研究思路和深度学习神经网络的识别模型模板。DENA已经在Github上开放并可免费获取(网址:https://github.com/weir12/DENA)。
中国科学院分子植物科学卓越创新中心博士研究生秦航、中国科学院巴斯德研究所博士研究生欧亮为本文的共同第一作者。中国科学院分子植物科学卓越创新中心李轩研究员和王佳伟研究员、中国科学院巴斯德研究所郝沛研究员为共同通讯作者。该研究工作获得了国家重点研发计划、国家自然科学基金和中科院先导项目的支持。
论文连接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-021-02598-3
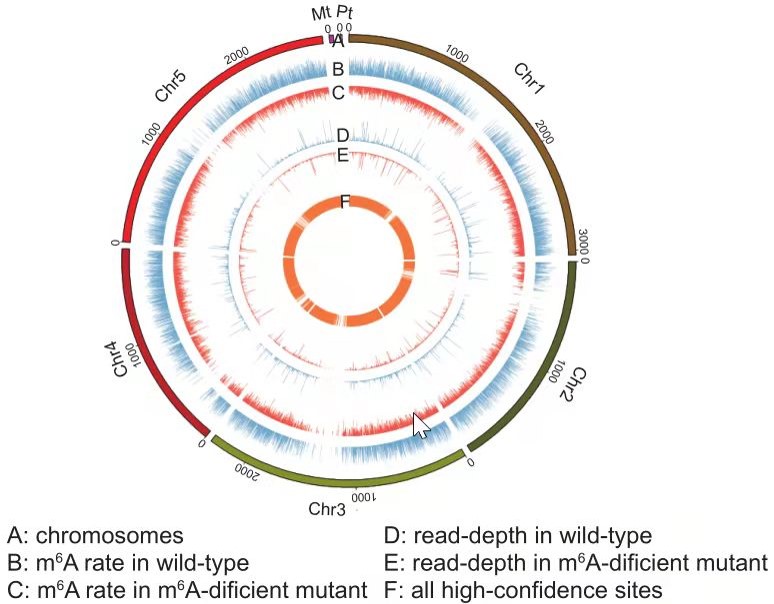
全转录组水平上、单碱基精度的拟南芥mRNA的m6A修饰图谱