
-

生物通官微
陪你抓住生命科技
跳动的脉搏
捕捉人类的古老起源
【字体: 大 中 小 】 时间:2023年12月25日 来源:Twist Bioscience
编辑推荐:
存储在DNA中的模式可以让我们了解古人类的生活、他们那个年代的群体动态,并有望告诉我们现代人类是如何形成的。然而,从这些饱经风霜的样本中提取这些信息却并非易事。
在我们这个世界里,DNA是看不见的永恒。DNA中的A、T、G、C不仅构成了地球上所有生命的基础,而且从生物体中脱落的DNA也将遍布我们周围的环境,在某些情况下,DNA的寿命比我们还要长。
作为一种生物材料,DNA特别稳定。若存储在阴凉干燥的环境中,比如洞穴深处的矿化骨内,DNA可以稳定保存数十万年1。因此,从出土的古人类遗骸中发现DNA也就不足为奇了,它呈现了一扇了解过去的窗口,研究人员可以通过它来研究人类的进化史。
古DNA的前景在于其携带信息的能力。存储在DNA中的模式可以让我们了解古人类的生活、他们那个年代的群体动态,并有望告诉我们现代人类是如何形成的。然而,从这些饱经风霜的样本中提取这些信息却并非易事。不仅在技术上存在挑战,而且提取DNA的过程是破坏性的,这意味着这些珍贵的文物必将遭到破坏,甚至彻底摧毁。因此,处理古DNA的过程容不得半点差错,即使是微小的失误也可能造成无法挽回的损失。面对如此高的风险,对研究人员来说关键在于拥有最合适的工具。
窥探过去的工具
1984年,加利福尼亚州大学伯克利分校的研究人员利用分子克隆技术产生了世界上第一段古DNA序列:线粒体DNA的两个短片段(< 300 bp),来自保存了约140年的斑驴(Equus quagga)肌肉组织标本2。这项研究不仅为马的进化史提供了有趣的信息,而且证明了一个概念,即使是年代久远的DNA也能提供有关过去的宝贵信息,从而建立了古基因组学(paleogenomics)这一新领域。
分子克隆也许让我们首次涉足古基因组学领域,但随着更先进的测序技术的采用,它很快就被淘汰了3,4。如今,研究古DNA的研究人员就像外科医生伸手去拿手术刀一样,往往会在新一代测序(NGS)的上游使用靶向富集组合,以帮助他们从古老样本中提取可靠的数据。
与早期技术(比如分子克隆和PCR)的局部性相比,NGS可实现大规模并行测序和广泛覆盖,因此单次运行即可对个体的整个基因组进行测序。NGS技术首次应用于古DNA分析是在2006年,当时研究人员对从一头28,000年前的长毛猛犸象身上提取的约13 Mb DNA和从一名尼安德特人身上提取的约1 Mb DNA进行了测序4。NGS提供的大量测序信息推动了古基因组学的蓬勃发展(详见图1)。
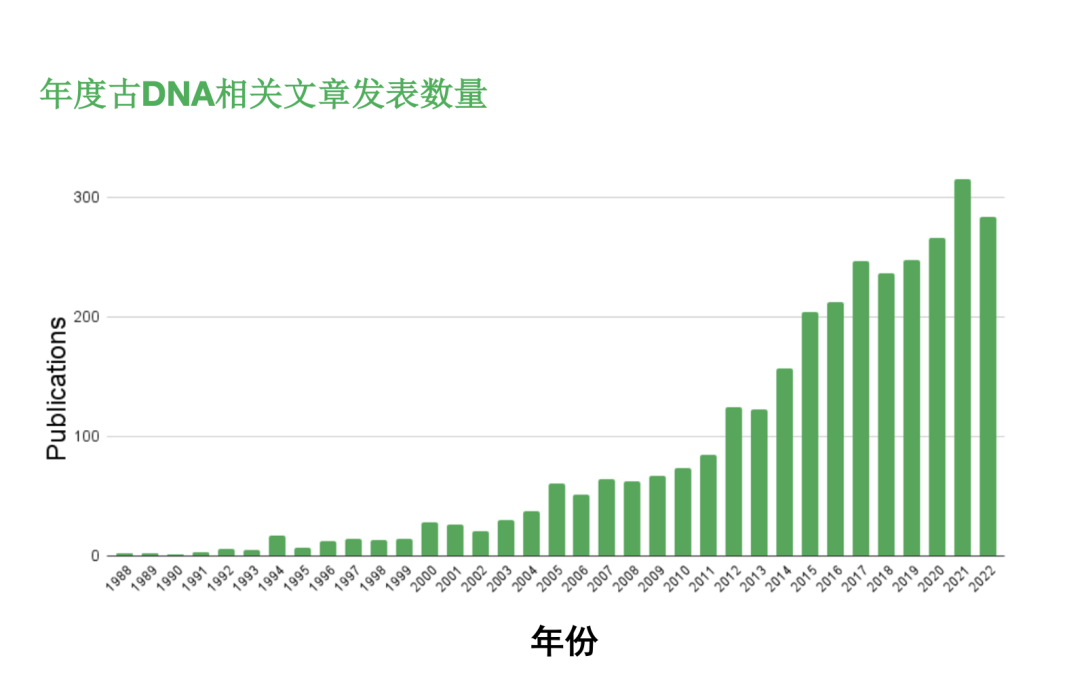
图1. 柱状图显示了NCBI PubMed数据库中包含“古DNA”一词的论文年度统计。
过去十五年来,NGS技术的普及加速了古DNA领域的研究。
然而,尽管NGS有诸多好处,但它也是一个成本极高的过程,特别是对古基因组研究来说。通常情况下,古DNA只占出土遗骸中总DNA的不到10%,其余90%(甚至更多)由细菌、真菌或现代人脱落的外源DNA组成4。如果不加区分地应用NGS技术,则会浪费大量资源对这些无法提供信息的污染DNA进行测序。这正是靶向富集NGS(以下简称靶向NGS)的优势所在。
古基因组学中的靶向NGS组合
有了靶向NGS,研究人员能够将测序资源集中在他们感兴趣的基因片段上。这通常是通过合成生物素化的寡核苷酸探针来实现的,这些探针与特定的目标序列互补。将这些寡核苷酸探针与样本DNA共同孵育后,它们会与互补的目标序列结合。之后加入链霉亲和素包被的磁珠来固定探针及其结合的任何序列。在洗掉样本中剩余的DNA后,剩下的就是感兴趣的结合片段。
通过去除过多的DNA,研究人员能够将测序资源集中在特定的基因组目标上,从而实现更深度的测序覆盖。这通常意味着以更少的资金投入获得更准确的测序结果。有关靶向富集的优势,请点击阅读我们之前发表的博文:一文了解 NGS靶向富集。
为了帮助您认识靶向富集在古DNA研究方面的强大潜力,我们做了一些粗略的数学计算。以标准的30倍覆盖度对现代人类基因组进行全基因组测序,需要约4亿条测序读数。然而,古DNA样本往往已被大量非人类DNA污染。而且,存在的古人类DNA通常已降解,仅有不到1%的序列能产生可用的数据。若利用广泛的非靶向策略使得感兴趣的变异达到所需的覆盖度,则会将总的测序读数推向数十亿条,这很快就会造成实验成本过高,且不切实际。有了富集技术,我们就能仅靶向感兴趣的变异区域,同时去除大部分污染DNA序列。因此,研究人员能够从古代样本中提取出成千上万个变异的宝贵信息,而每个样本只需2500万条读数。
自2015年以来,古基因组学领域在很大程度上依赖于一种被称为“1240k试剂”的特定靶向富集组合,它包含一组寡核苷酸探针,其捕获的DNA片段中含有124万个被认为可提供信息的多态性。截至2022年10月,世界上超过70%的全基因组古人类DNA数据都是用这个组合生成的5。
尽管1240k试剂是一种广泛使用的工具,但它也存在一些局限性,包括富集效果不一、等位基因捕获存在偏倚,以及由于缺乏商业化产品,合成目标探针组的成本太高5。总的来说,这些局限性使得人们很难对该领域内的数据集进行比较,同时也阻碍了大多数实验室开展古DNA的全基因组研究。为了减少这些问题,顶尖研究人员一直在努力改进古DNA靶向富集组合的设计,并推动其普及。
🧬 DNA衰变的类型
DNA随时间降解的方式有很多种。下面介绍了几种:
片段化:糖-磷酸骨架和嘌呤碱基之间的化学键很容易水解,导致DNA链上出现缺失位点。反过来,缺失位点又会导致链的断裂。这就是古DNA在提取时通常会产生较短片段的原因。
脱氨基:DNA衰变的另一种自然形式是胞嘧啶脱氨基后形成尿嘧啶。这是一个重要的变化,因为它会导致聚合酶在复制过程中发生碱基置换,造成C→T和G→A的置换。这两种情况都会干扰序列分析。
损伤:DNA上可能会发生许多不同的修饰,从而导致交联。这种修饰会阻碍酶发挥其功能,并阻止DNA链的复制。
Twist古DNA组合
2021年,Twist Bioscience推出了用于古DNA的商业化靶向富集组合,可富集1240k试剂覆盖的124万个多态性以及其他感兴趣的内容。哈佛大学的Nadin Rohland、Swapan Mallick、Matthew Mah、Robert Maier、Nick Patterson和David Reich着手将他们优化的Twist古DNA组合与1240k试剂及另一种商业化组合进行独立比较5。结果表明,Twist的组合实现了高度均一的覆盖,片段长度偏倚较小,SNP整体覆盖度提高了1.2至1.4倍,并且提供了高质量数据,使其能够与鸟枪法测序数据共同分析,而不会产生明显偏倚(这是该领域长期存在但尚未解决的问题)。
Twist古DNA组合的高性能使得哈佛大学实验室后来不再使用1240k试剂,转而使用经过优化的新组合,Twist试剂的使用次数已超过1万次5,6。作为一种商业化资源,Twist的组合还减轻了合成靶向富集探针的经济负担,为更广泛参与古基因组学研究打开了大门。
参考文献:
1. Allentoft, Morten E., et al. “The Half-Life of DNA in Bone: Measuring Decay Kinetics in 158 Dated Fossils.” Proceedings of the Royal Society B: Biological Sciences, vol. 279, no. 1748, 10 Oct. 2012, pp. 4724–4733, https://doi.org/10.1098/rspb.2012.1745.
2. Higuchi, Russell, et al. “DNA Sequences from the Quagga, an Extinct Member of the Horse Family.” Nature, vol. 312, no. 5991, Nov. 1984, pp. 282–284, ui.adsabs.harvard.edu/abs/1984Natur.312..282H, https://doi.org/10.1038/312282a0.
3. Danielewski, Mikołaj, et al. “Methodological Changes in the Field of Paleogenetics.” Genes, vol. 14, no. 1, 16 Jan. 2023, p. 234, https://doi.org/10.3390/genes14010234.
4. Shapiro, B., and M. Hofreiter. “A Paleogenomic Perspective on Evolution and Gene Function: New Insights from Ancient DNA.” Science, vol. 343, no. 6169, 23 Jan. 2014, pp. 1236573–1236573, science.sciencemag.org/content/343/6169/1236573.full, https://doi.org/10.1126/science.1236573.
5. Rohland, Nadin, et al. “Three Assays for In-Solution Enrichment of Ancient Human DNA at More than a Million SNPs.” Genome Research, vol. 32, no. 11-12, 1 Nov. 2022, pp. 2068–2078, https://doi.org/10.1101/gr.276728.122.
6. Liu, Yue-Chen, et al. “Ancient DNA Reveals Five Streams of Migration into Micronesia and Matrilocality in Early Pacific Seafarers.” Science, vol. 377, no. 6601, July 2022, pp. 72–79, https://doi.org/10.1126/science.abm6536.