
-

生物通官微
陪你抓住生命科技
跳动的脉搏
新的基因丢失鉴定方法:LOST&FOUND
【字体: 大 中 小 】 时间:2023年06月05日 来源:北京大学新闻网
编辑推荐:
近日,北京大学生物医学前沿创新中心(BIOPIC)、北京未来基因诊断高精尖创新中心(ICG)、北京大学生命科学学院生物信息中心(CBI)、蛋白质与植物基因研究国家重点实验室高歌课题组在期刊Molecular Biology and Evolution上发表论文,在前期工作基础上提出了新的基因丢失鉴定方法LOST&FOUND,并基于该方法对人类中的基因丢失及其影响进...
作为细胞中基本的功能单位,蛋白编码基因(Protein-coding genes,以下简称基因)可以通过转录-翻译过程指导合成对于生命活动至关重要的蛋白质,进而影响生命体的生理/病理性状。因此,基因的演化与生物体的表型演化息息相关,是介导物种特异性表型及其环境适应的重要动力之一。在演化过程中生物体可以获得新的基因来执行新的功能;因此,自上世纪后期至今,新基因(Gene Birth)已成为相关领域的研究热点。另一方面,万物皆有生有灭,与不断涌现的新基因相对,现有的蛋白编码基因也可能因突变等失去原有的功能并进而从基因组中丢失。然而,受限于可用数据等多方面因素,对基因丢失(Gene Loss)及其影响长期以来尚缺乏系统研究。
近日,北京大学生物医学前沿创新中心(BIOPIC)、北京未来基因诊断高精尖创新中心(ICG)、北京大学生命科学学院生物信息中心(CBI)、蛋白质与植物基因研究国家重点实验室高歌课题组在期刊Molecular Biology and Evolution上发表了题为“Genome-wide identification of gene loss events suggests loss relics as a potential source of functional lncRNAs in humans”的生物信息学论文,在前期工作基础上提出了新的基因丢失鉴定方法LOST&FOUND,并基于该方法对人类中的基因丢失及其影响进行了系统解析。
为精准鉴定基因丢失事件,高歌团队提出了新的基因丢失鉴定方法LOST&FOUND。LOST&FOUND的具体处理流程如图1所示。LOST&FOUND结合了全基因组比对以及多物种中的直系同源基因数据,并运用最大简约法等处理方法来对基因丢失进行鉴定识别。基于多物种直系同源基因关系的推断可使LOST&FOUND避免将参考物种中的基因获得事件错误识别为待研究物种中的基因丢失事件。而全基因组比对的使用则可充分考虑基因及其上下游区段的同源性,有利于LOST&FOUND识别由大片段删除等方式造成的基因丢失事件。

图1
基于该方法,团队在人类基因组中共鉴定到155个基因丢失事件。其中,有88个基因丢失事件在人类基因组内含有同源区段残骸(relics)。有趣的是,通过将基因丢失残骸与长非编码RNA区段进行比较,团队发现,在人类基因组中,有33个基因丢失事件的发生与长非编码RNA的起源相关,并将该部分通过基因丢失起源的长非编码RNA命名为derived lncRNA(图2 A-B)。
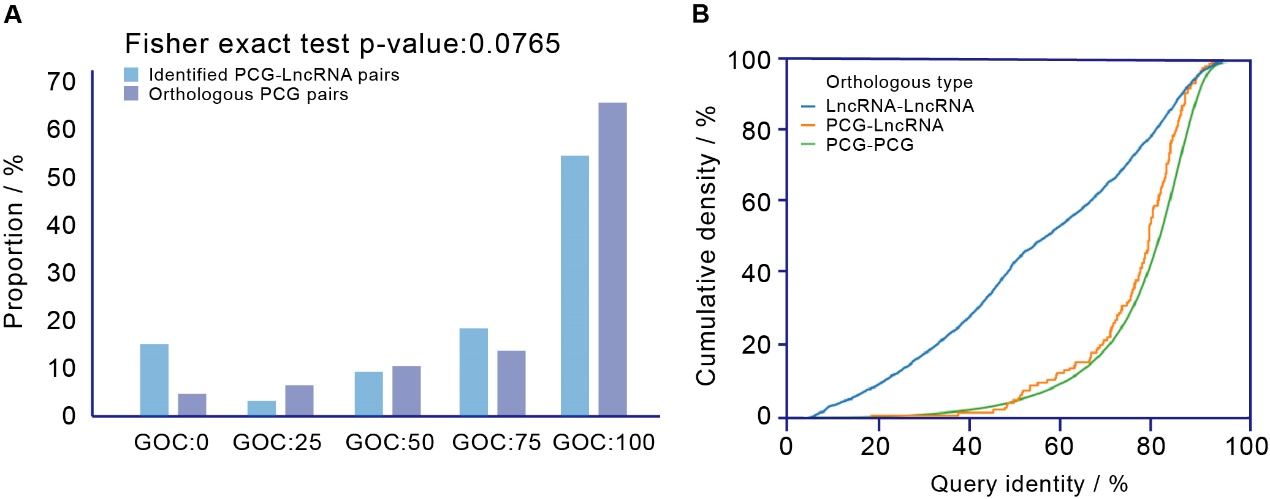
图2
综合多方面的注释,团队发现,derived lncRNA与其它长非编码RNA不同,其往往有着更高的表达量、更低的组织表达特异性、更长的转录本结构以及更强的序列保守性(图3 A-F)。基于GWAS、共表达、已有实验检索等功能性分析的结果则表明,这部分derived lncRNA与生长发育、免疫、生殖以及抑癌作用等过程的调控均有关系。同时,超过一半的derived lncRNA均受到了正选择的作用。
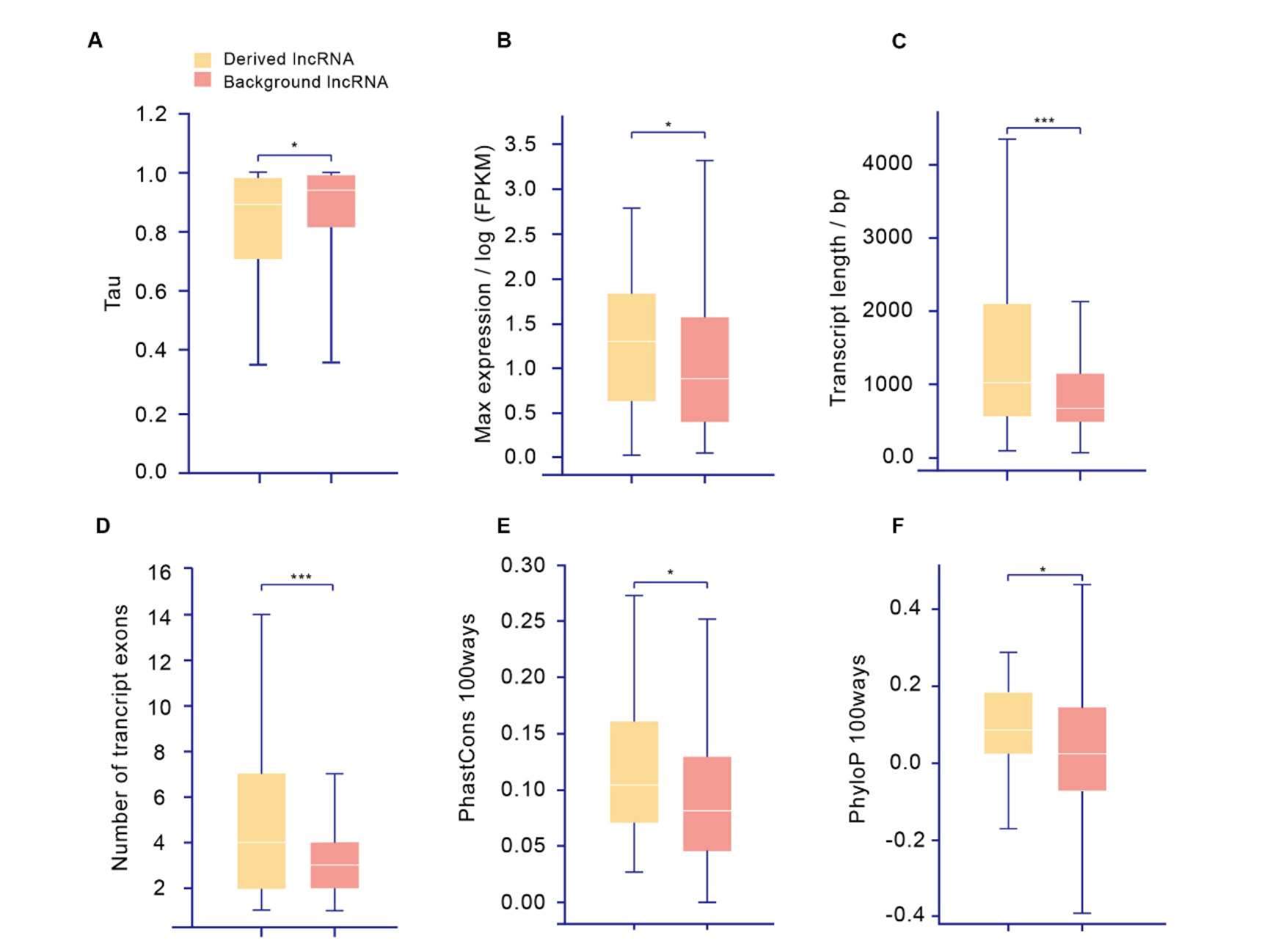
图3
高歌团队开发了新的基因丢失鉴定方法并通过该方法系统性研究了人类基因组中基因丢失的发生与影响。值得注意的是,高歌团队在人类基因组中发现了33个基因丢失事件与长非编码RNA的起源相关,且这部分长非编码RNA可能具有重要功能。结合前期已发表工作1-3,这些结果提示在多个物种中,演化过程中古老的蛋白编码基因有可能“再生”为新的长非编码RNA来发挥作用,在基因生-死、编码-非编码等看似对立的概念之间建立起了有趣的联系。
高歌的博士生温正扬为该论文第一作者,高歌为该论文通讯作者。该研究得到了蛋白质与植物基因研究国家重点实验室、北京未来基因诊断高精尖创新中心等的资助。计算分析工作于北京大学高性能计算校级公共平台和北京大学太平洋高性能计算平台完成。
参考文献:
1 Duret, L., Chureau, C., Samain, S., Weissenbach, J. & Avner, P. The Xist RNA gene evolved in eutherians by pseudogenization of a protein-coding gene. Science312, 1653-1655 (2006).
2 Zhao, Y. et al. Identification and analysis of unitary loss of long-established protein-coding genes in Poaceae shows evidences for biased gene loss and putatively functional transcription of relics. BMC Evol Biol15, 66 (2015).
3 Hezroni, H. et al. A subset of conserved mammalian long non-coding RNAs are fossils of ancestral protein-coding genes. Genome Biol.18, 162 (2017).