
-

生物通官微
陪你抓住生命科技
跳动的脉搏
【新工具】从长读长异构体测序中准确鉴定融合转录本
【字体: 大 中 小 】 时间:2024年03月22日 来源:基因有限公司
编辑推荐:
Broad研究所等人员开发了一个新的工具CTAT-LR-fusion(作为Trinity Cancer Transcriptome Analysis Toolkit的一部分),用于从bulk RNA-seq/scRNA-seq的长读长数据中准确识别融合转录本。
基因融合
基因融合在多种成人和儿童癌症中作为癌症驱动因素被发现。准确检测融合转录本对于癌症的临床诊断、预后评估以及指导治疗开发至关重要。目前大多数融合转录本检测方法采用Illumina RNA-seq,但受限于短读长序列的限制,只能识别基因融合转录本的一部分结构。而长读长异构体测序(Long read Isoform sequencing,LR-Iso-seq)的最新进展使得在bulk RNA-seq和单细胞转录组(scRNA-seq)中检测完整融合转录本成为可能。
文献速递
Broad研究所等人员开发了一个新的工具CTAT-LR-fusion(作为Trinity Cancer Transcriptome Analysis Toolkit (CTAT)的一部分),用于从bulk RNA-seq/scRNA-seq的长读长数据中准确识别融合转录本。用该工具分别对9种肿瘤细胞系、T细胞浸润黑色素瘤样本和三种高级别浆液性卵巢癌样本的长读长和短读长RNA测序数据进行分析,结果表明该工具在真实长读长RNA-seq数据的融合转录本检测中显示出超过其他工具的准确性,并且长读长测序在融合检测中显示出比短读长测序更高的灵敏度。

结论
1 CTAT-LR-fusion pipeline
为了进一步推进使用LR-Iso-seq的融合转录本检测,研究人员专门为LR-Iso-seq开发了CTAT-LR-fusion(含或不含Illumina RNA-seq数据)。CTAT-LR-fusion包括两个阶段:首先使用minimap快速识别候选的嵌合长读长,然后通过FusionInspector对候选融合基因对进行建模,并对融合转录本的表达量进行量化、基因融合注释和交互式可视化。
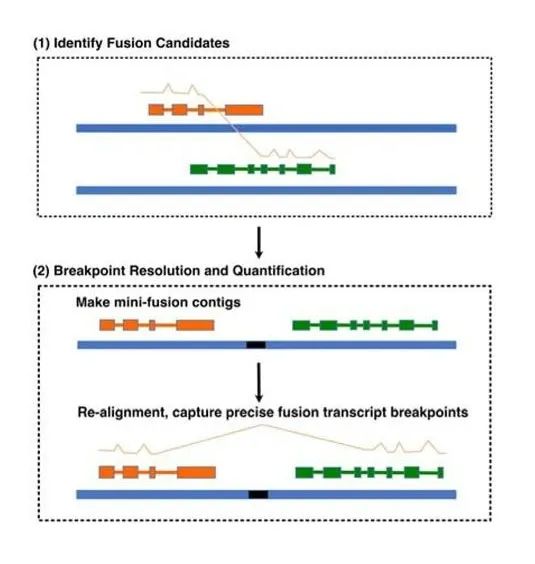
图1. CTAT-LR-fusion工作流程。
2 利用模拟的长读长数据来检验不同工具检测融合转录本的准确性
为更接近真实测序数据情况,这里依次使用了两组模拟数据:序列差异①从25%错误到5%错误(图2b,BadRead模拟)和②1%错误到0.1%错误或更好(图2c,PBSIM3模拟)的PacBio和ONT长读长数据,对比了CTAT-LR-fusion与其他长读长融合转录本检测工具(包括JAFFAL、LongGF、FusionSeeke和pbfusion)的准确性。
总的来说,在考虑融合基因的顺序和方向时,CTAT-LR-fusion和JAFFAL在这些模拟测试数据中表现最好,CTAT-LR-fusion在大多数错误率和测序技术的组合中表现最好,表现出高度准确的融合转录本断点检测,且正确地报告了融合基因对在相应的融合转录本中从5 '到3 '的融合顺序(图2a,b,c)。
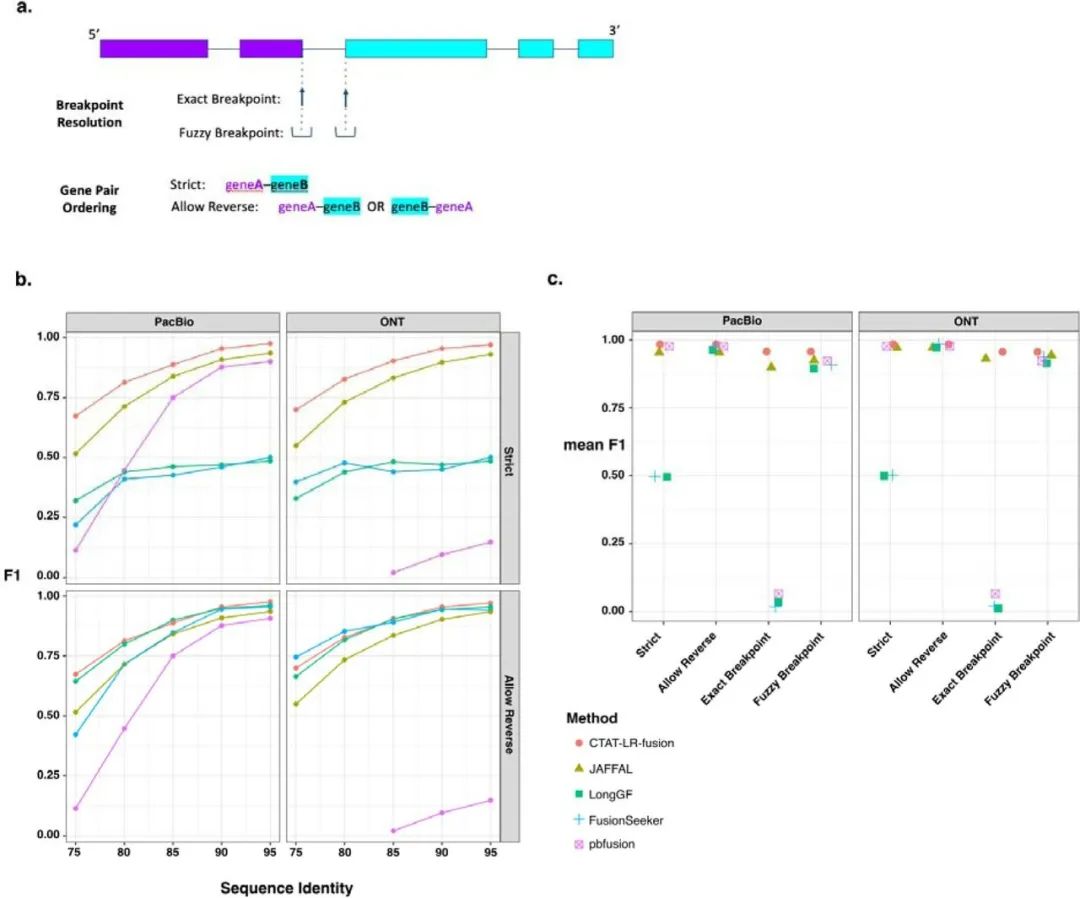
图2. 利用模拟的长读长数据来检验不同工具检测融合转录本的准确性。
3 参考RNA样本的长读长融合转录本检测
接下来,研究人员将商品化RNA样本(Seraseq Fusion RNA Mix v4,包含16个临床相关的融合转录本)以固定浓度混合到人类细胞系(GM24385)的总RNA中,使用MAS-ISO-seq方法进行长读长测序(3个重复;PacBio已基于此方法开发了商品化的Kinnex试剂盒,以提高测序通量)。并同时进行Illumina测序,用来评估CTAT-LR-fusion的分析能力以及长读长和短读长在融合转录本的检测能力。
结果表明所有16个融合转录本均通过CTAT-LR-fusion在三个PacBio MAS-ISO-seq重复中检测到(图3a)。但Illumina TruSeq RNA-seq中对应融合转录本的reads相对较少,并且三个重复中并没有检测到所有的融合转录本(图3a)。
在多种长读长融合转录本检测工具中,只有CTAT-LR-fusion和pbfusion (v0.4.0版本)能够识别三个PacBio MAS-ISO-seq数据中的16个融合转录本(图3b)。特别是对于前列腺癌的标志性融合TMPRSS2::ERG,LongGF和FusionSeeker都缺失了,而CTAT-LR-fusion则在三个PacBio MAS-ISO-seq数据中检测到45、98和104个支持TMPRSS2::ERG的长异构体reads。
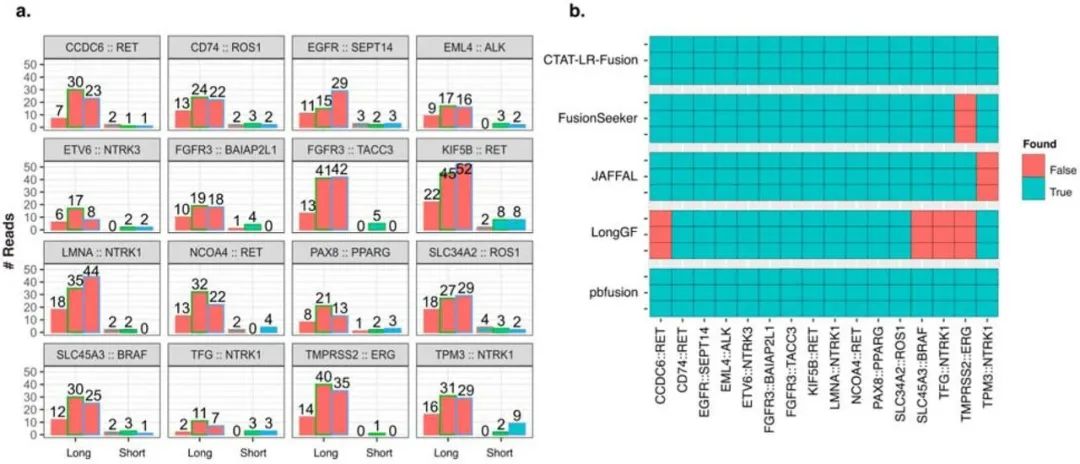
图3. SeraCare v4混合的参考样本的融合转录本检测。
4 9株肿瘤细胞系的长读长融合异构体检测
研究人员进一步利用来自乳腺癌(SKBR3、HCC1187、HCC1395)、前列腺癌(VCaP)、慢性骨髓性白血病(K562)、ALK+间变性大细胞淋巴瘤(KIJK)、T细胞淋巴瘤(MJ)、小细胞肺癌(DMS53)和尿路上皮性膀胱癌(RT112)等不同癌症类型的9种癌细胞系的转录组,探索了基于长读长的融合转录本检测。使用PacBio MAS-ISO-seq对每个细胞系进行bulk RNA-seq(每个样本约3-6M reads),并使用多种长读长融合转录本检测工具进行分析。
研究人员发现133个融合转录本被至少两种长读长融合转录本检测工具共同鉴定,其中在细胞系MJ中鉴定的融合转录本最少(3个),在VCaP中鉴定的最多(31个)(图4a)。在大多数(6/9)细胞系中发现了8种已知与癌症生物学相关的COSMIC融合(包括上述特征性融合转录本),并通过至少两种预测方法进行了鉴定,支持每种融合的reads数相似(图4b)。相比长读长测序数据识别到的133个融合转录本,短读长测序只检测出其中的79个(59%)。
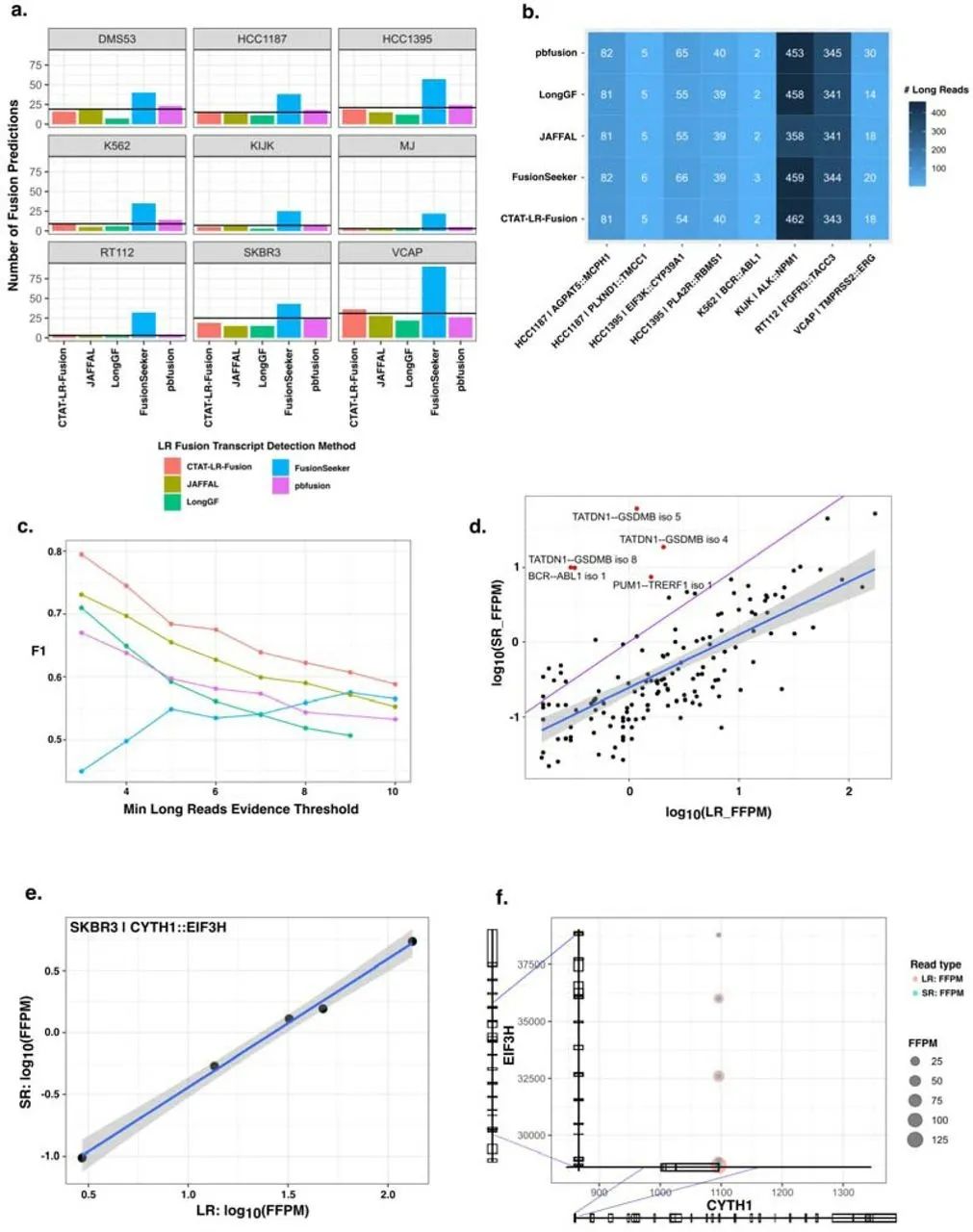
图4. 9株肿瘤细胞系MAS-ISO-seq融合转录本的检测。
5 肿瘤单细胞转录组的长读长融合异构体检测
为了检验CTAT-LR-fusion在单细胞长读长异构体测序数据中检测融合转录本的准确性,研究人员利用了两项近期发布的PacBio单细胞Iso-seq数据:T细胞浸润黑色素瘤肿瘤以及三种不同转移性高级别浆液性卵巢癌(HGSOC)。再加上对应的Illumina RNA-seq数据,进一步探索基于单细胞的长读长测序和短读长测序检测融合转录本的差异。
结合长读长、短读长融合转录本分析发现T细胞浸润黑色素瘤肿瘤数据中,NUTM2A-AS1::RP11-203L2.4存在于265个肿瘤细胞(38%)和仅3个正常细胞(0.05%)中(图5a)。另外,大约60%存在于肿瘤细胞的融合基因伴侣NUTM2A-AS1::RP11-203L2.4(在多种癌症类型中起作用)仅通过长读长证据被鉴定,另外20%仅通过短读长证据被鉴定,其余20%通过短读长和长读长证据同时被鉴定(图5b)。对于NUTM2A-AS1::RP11-203L2.4,长读长数据提供了8种不同的融合剪接异构体的证据,并明显可见完整的异构体结构(图5c)。短读长则提供了5种异构体的证据,且由于长度受限,只有融合转录本断点周围的部分异构体结构被解析(图5c)。
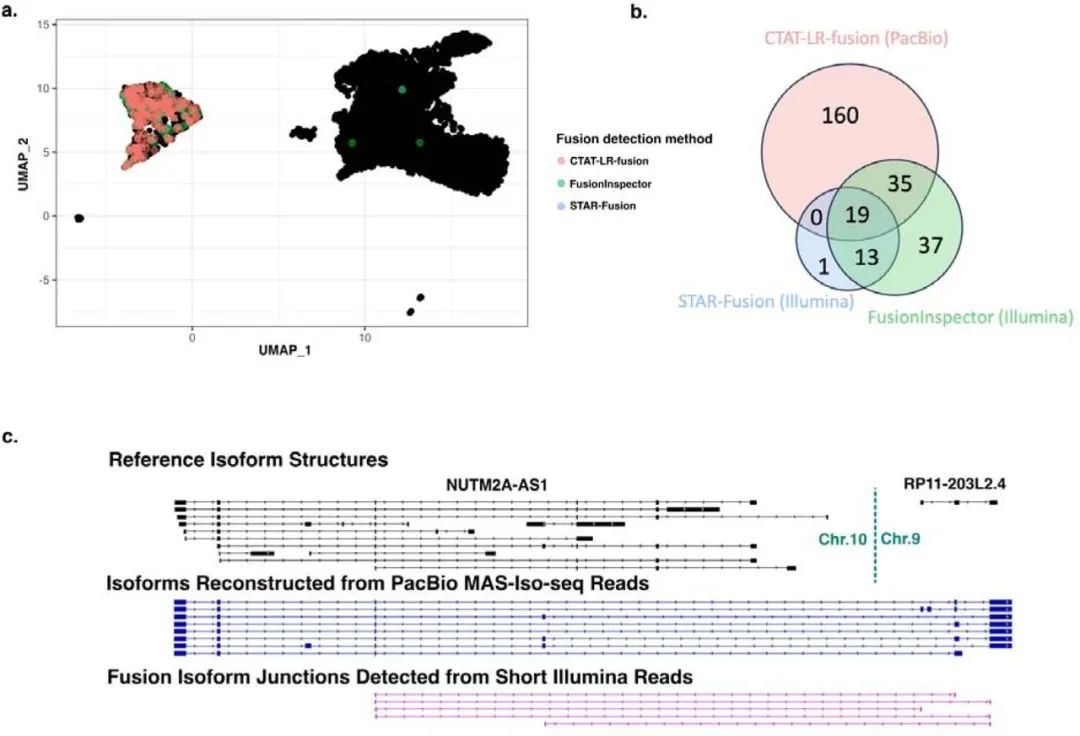
图5. 融合NUTM2A-AS1::RP11-203L2.4在T细胞浸润黑色素瘤样本中的检测。
在三种不同HGSOC患者样本中:
①患者-1肿瘤样本中研究人员仅鉴定出四种体细胞融合转录本:SMG7::CH507- 513H4.1(26个细胞),RAPGEF5-AGMO(6个细胞),NTN1-CDRT15P2(5个细胞)和GS1-279B7.2- GNG4(5个细胞)。其中三种类型的融合都只通过长读长才能发现,唯有RAPGEF5::AGMO一半(3/6)的细胞仅通过长读长测序检测到,1/6的细胞仅通过短读长测序检测到。
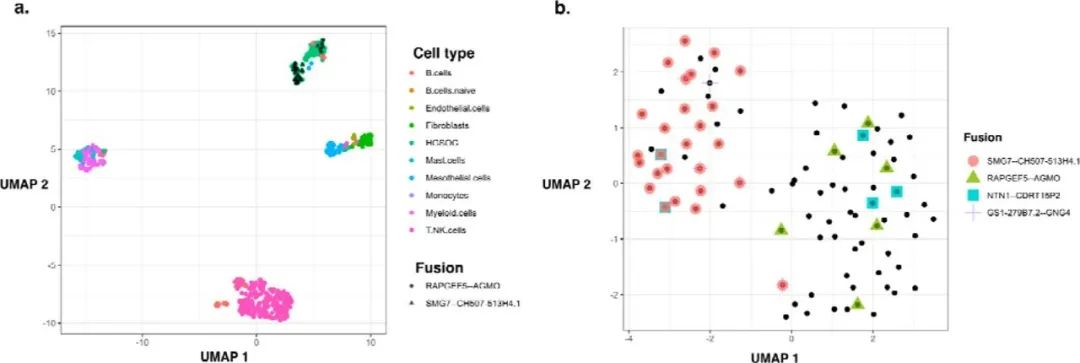
图6: 肿瘤细胞中融合表达的异质性。(A) HGSOC患者1所有细胞的UMAP图。(B)分析HGSOC患者1的HGSOC细胞的UMAP聚类发现,RAPGEF5::AGMO只在右簇中表达;SMG7::CH507- 513H4.1和GS1-279B7.2::GNG4的共表达且几乎完全在左侧簇中表达。融合NTN1::CDRT15P2在肿瘤细胞簇中均有表达,更可能是克隆表达。
②患者-2肿瘤样本中研究人员鉴定出16种不同的恶性细胞富集融合转录本,包括早期鉴定的IGF2BP2::TESPA1(176个肿瘤细胞,85%)和SPATS2::TRA2B(21个肿瘤细胞,10%),且几乎所有的SPATS2::TRA2B表达细胞(20/21)都共表达IGF2BP2::TESPA1。其中,患者2肿瘤样本中已知的肿瘤致癌基因,包括CBL::KMT2A(16个肿瘤细胞)和DEK::CASC17(11个肿瘤细胞),两者均仅通过长读长测序识别;另一种常见的融合PSMB7::SCAI(52个肿瘤细胞)也主要通过长reads检测到,具有四个融合剪接异构体。
③患者-3肿瘤样本中只有2个融合转录本在肿瘤细胞中富集:在16个细胞中发现了CBLC::CTC-232P5.1融合,另外在8个细胞中发现了SNRNP70::ZIK1。有趣的是,这些表达SNRNP70:: ZIK1的细胞都共同表达了CBLC::CTC- 232P5.1融合,这两种融合都涉及定位于chr19底部臂的基因(CBLC和SNRNP70在5Mb内的转录断点),并且可能源于相同的基因组重组事件。有证据表明,CBLC::CTC- 232P5.1融合存在5个融合转录本断点,表明至少有5个融合剪接同种异构体,其中融合SNRNP70::ZIK1仅通过长读长测序识别。
结论
融合转录本的正确检测和报告需要考虑融合基因在表达融合转录本的背景下的顺序和方向,以及融合转录本断点的准确报告,这通常涉及标准转录本剪接,将一个基因的外显子融合到融合伙伴的外显子上。因此长读长测序凭借其既长且准的特点在融合检测中显示出比短读长测序更高的灵敏度。
而该研究则是从数据分析角度适配长读长异构体测序数据,文中阐述了CTAT-LR-fusion工具的开发、应用和性能评估,展示了其在癌症研究和单细胞转录组分析中的潜力。总体而言,CTAT-LR-fusion在长读长和短读长数据的融合转录本检测中表现出色,尤其是在处理长读长数据时,能够提供更准确和全面的融合转录本信息。这些结果强调了长读长测序技术在癌症研究和单细胞分析中的重要性,以及CTAT-LR-fusion作为分析工具的有效性。
CTAT-LR-fusion可在https://github.com/TrinityCTAT/CTAT-LR-fusion/wiki获得。
原文链接:https://www.biorxiv.org/content/10.1101/2024.02.24.581862v1.full
PacBio Kinnex系列试剂盒
Kinnex试剂盒基于MAS-Seq方法,可将较小的DNA片段连接成较长的HiFi可用文库,提高测序通量,使长读长RNA-seq更具成本效益。
Kinnex单细胞RNA试剂盒以现有的 MAS-Seq for Single Cell 3' 试剂盒为基础,增加了对 10x Genomics 5' 试剂盒和文库复用的额外支持。可使测序通量提高16倍,获得基因表达及全长isoform信息,在单细胞水平上揭示RNA异构体多样性。
Kinnex全长RNA试剂盒可进行全长 RNA 测序,与典型的Iso-seq文库相比,其通量提高了8倍。可实现从5’端到3’端全长异构体测序,准确表征剪接位点,发现新基因和新异构体,鉴定融合基因,并获得基因及异构体read计数信息进而分析表达量。
— End —
基因有限公司作为PacBio公司的中国区合作伙伴,自2011年以来将PacBio第三代单分子实时测序技术引入国内,一直为国内用户提供专业的三代测序系统的安装培训,技术支持,应用培训与售后维护工作,赢得客户的一致好评与信任。基因有限公司将一如既往的支持越来越多的PacBio用户。
关于基因
基因有限公司成立于1992年,是一家提供生命科学科研仪器、试剂耗材和技术服务的综合服务商。基于“Gene Brightens Every Life • BioTech Connects the World”——“基因燃亮生命 • 生物技术连接世界”的愿景,专注于生命科学领域前沿技术的引进和推广,致力于推动该领域国内科研机构硬件水平及实验方案的革新与升级。同时,公司也一直致力于自主品牌的科研设备的研发与生产,拥有一系列通用性强、互补性高的自主品牌产品。
基因的服务网络遍及全国各地十多个大中城市,拥有包括仪器销售,试剂销售,市场与技术支持,维修,客服,物流等多个部门组成的完整服务体系。
我们希望通过不懈努力,为您的成功铺路搭桥,也为中国的生命科学事业赶超世界先进水平尽一己之力。欲了解更多信息,请访问www.genecompany.cn。
