
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于课程学习深度学习的宏蛋白质组学肽段鉴定增强方法
【字体: 大 中 小 】 时间:2025年10月09日 来源:Nature Communications 15.7
编辑推荐:
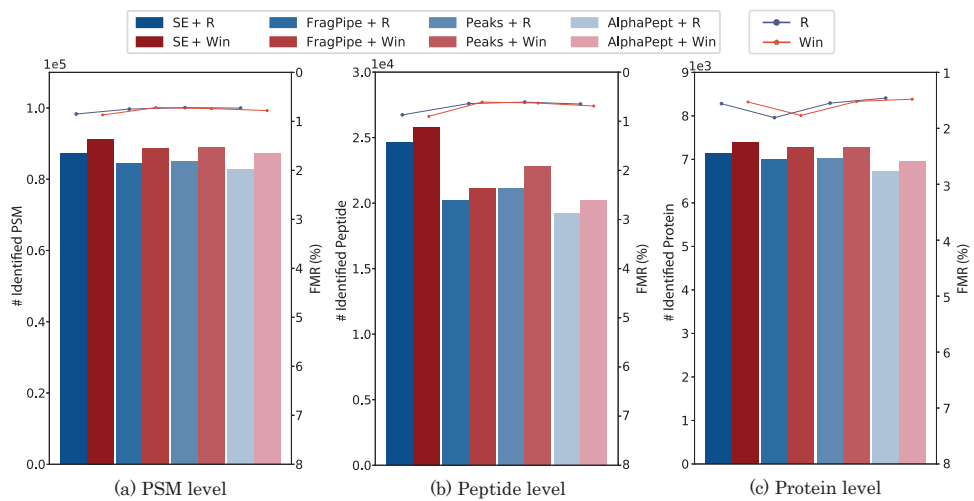
为解决宏蛋白质组学中因庞大且不完整的蛋白质数据库导致的肽段鉴定难题,研究人员开发了基于深度学习的PSM过滤工具WinnowNet。该方法采用课程学习策略,利用Transformer和CNN架构处理无序PSM数据,在同等错误发现率下比Percolator、MS2Rescore和DeepFilter等主流工具鉴定出更多真实肽段,并在人类肠道微生物组中发现更多与饮食健康相关的生物标志物,为个性化医疗提供有力支持。



 生物通微信公众号
生物通微信公众号
知名企业招聘