综述:单细胞组学领域的变革性进展:基础模型、多模态整合与计算生态系统的全面综述
《Journal of Translational Medicine》:Transformative advances in single-cell omics: a comprehensive review of foundation models, multimodal integration and computational ecosystems
【字体:
大
中
小
】
时间:2025年10月29日
来源:Journal of Translational Medicine 7.5
编辑推荐:
本综述系统梳理了单细胞多组学(single-cell multi-omics)领域三大革命性进展:基础模型(Foundation Models, scFMs)如scGPT和scPlantFormer在跨物种细胞注释和扰动建模中的卓越表现;多模态整合(Multimodal Integration)策略(如PathOmCLIP)如何协调转录组、表观基因组和空间成像数据;以及计算生态系统(Computational Ecosystems)如何通过标准化平台(如BioLLM)促进全球协作。文章指出了技术可变性、模型可解释性等挑战,并提出了连接计算洞察与精准医疗(Precision Medicine)的行动策略。
单细胞组学技术的出现彻底改变了在细胞水平研究生物系统的能力,为探索细胞异质性、发育路径和疾病机制提供了前所未有的见解。诸如单细胞RNA测序(scRNA-seq)、空间转录组学(ST)和表观基因组分析等技术产生了海量数据集,捕获了数百万个单细胞的分子状态。然而,这些进步也暴露了传统计算方法的局限性,这些方法通常为低维或单一模态数据设计,难以应对现代单细胞数据集的高维性、技术噪声和多模态特性。这种不匹配催生了基础模型——大型预训练神经网络,它们作为解码细胞复杂性的变革性工具应运而生。
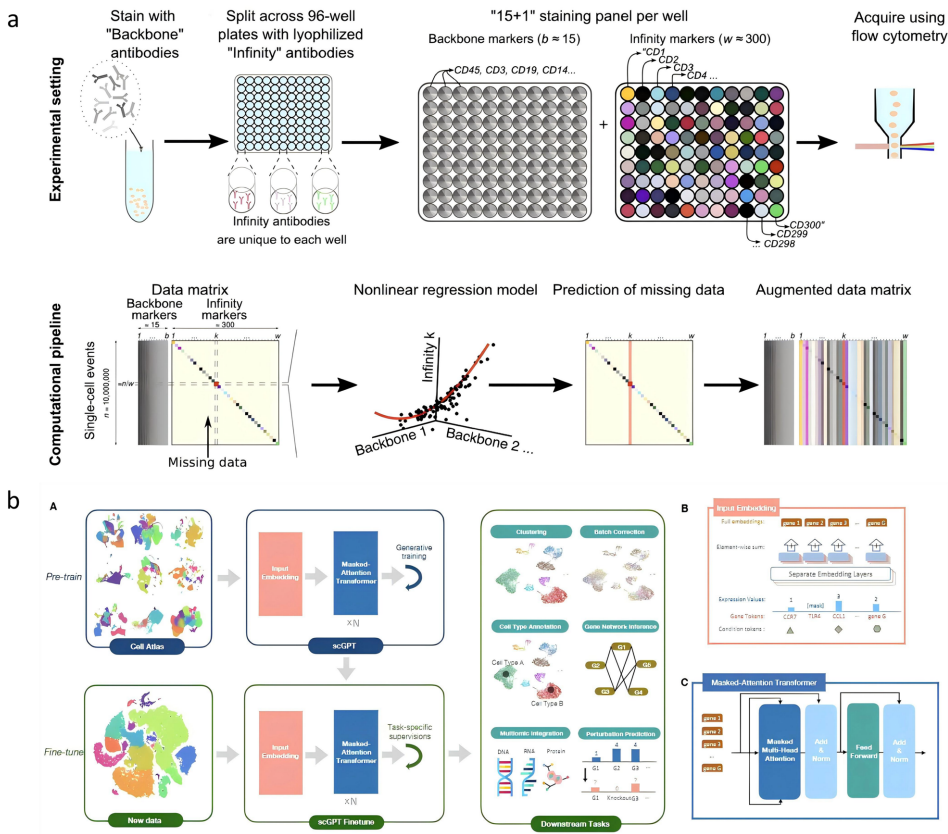
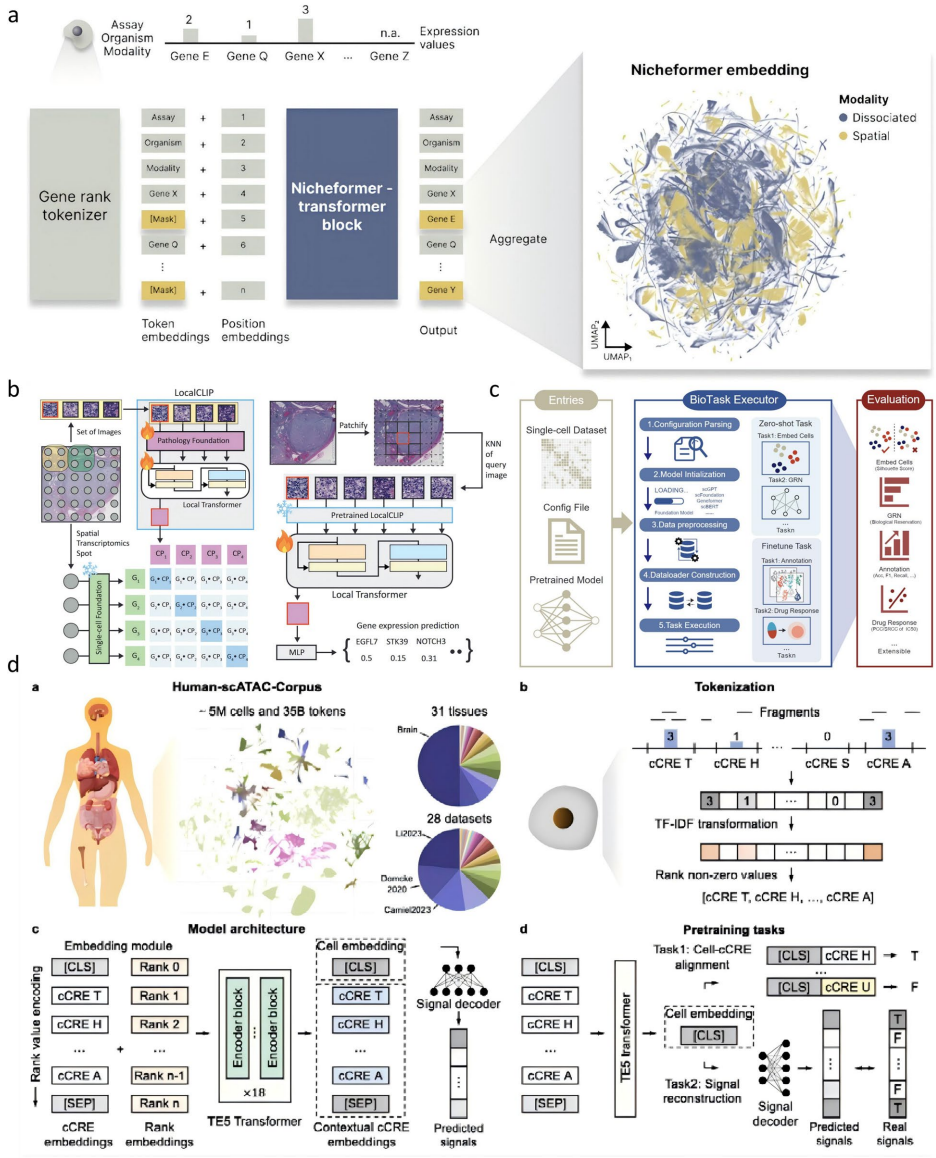
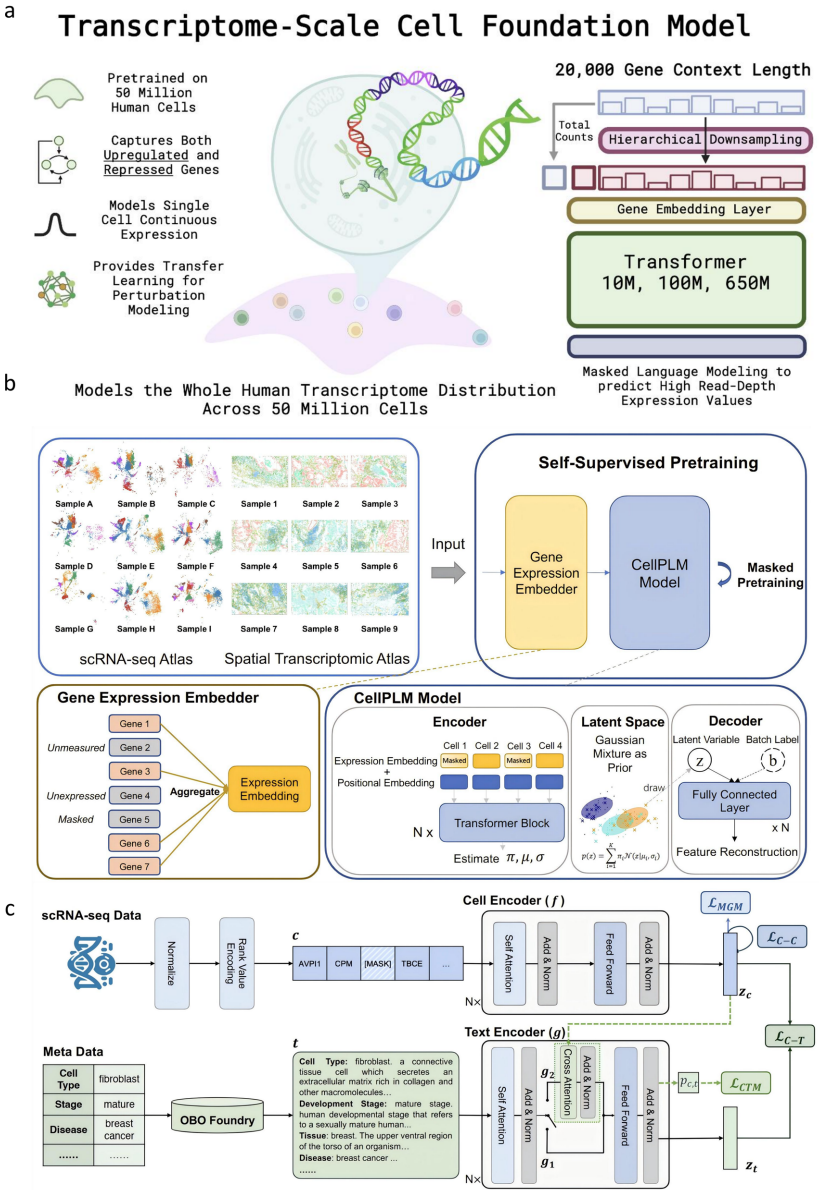
近期突破显示,基于Transformer的模型已成为该领域的主导架构。scGPT通过在超过3300万个细胞上进行预训练,为多组学任务设立了新基准,展示了其在跨任务泛化方面的卓越能力,例如零样本(zero-shot)细胞类型注释和扰动响应预测。与之相对,轻量级模型如scPlantFormer和CellPatch通过降低模型复杂度,在保持竞争力的同时提供了计算效率,后者甚至能将计算成本降低高达80%。混合架构也在兴起,例如scMonica融合了LSTM和Transformer模型,以捕捉生物数据中的时序动态。在规模扩展方面,Nicheformer在1.1亿个细胞上的训练创下了纪录,实现了强大的零样本能力。针对特定生物挑战,出现了如专注于表观基因组的EpiAgent和专注于扰动建模的CRADLE-VAE等专门的预训练框架。
多模态数据的整合已成为下一代单细胞分析的基石。在空间上下文建模方面,Nicheformer能够模拟组织内的细胞结构和相互作用。DECIPHER的解纠缠嵌入(disentangled embeddings)则进一步增强了空间分析能力。PathOmCLIP通过对比学习将组织学特征与多个肿瘤类型的转录组数据对齐,代表了多模态整合的重大创新。多模态框架正在迅速扩展,TMO-Net促进了跨癌症类型的多组学数据整合,而Vec3D则为三维分子景观可视化提供了可解释框架。GIST支持细胞结构的高分辨率3D分析。跨模态生成技术也在进步,例如Raman2RNA能够从显微镜图像预测基因表达谱。空间分析工具如BANKSY的统一细胞分型和Proximogram的疾病分类,正在改变我们基于空间分辨数据识别细胞类型和分类疾病的方式。为了应对整合挑战,StabMap的马赛克整合(mosaic integration)技术和sysVI的批次校正方法提供了解决方案。
对模型透明度的追求催生了用于分子调控因子识别的框架、Visible Neural Networks以及biolord的解纠缠表征等,旨在使模型预测更具可解释性和生物学意义。在计算效率方面,药物条件适配器(drug-conditional adapters)和启发式分块(heuristic patching)等创新优化了模型性能并最小化了资源需求。GPU加速技术显著减少了模型训练时间。集成计算生态系统的发展以ChatCell和CellWhisperer等平台为代表,它们实现了与单细胞数据的自然语言交互。此外,DISCO和CZ CELLxGENE等数据发现平台增强了单细胞数据集的可及性。BioLLM的通用接口等标准化努力正在建立共享和利用单细胞数据的通用框架。领域特定应用也在蓬勃发展,如在肝脏疾病、脑转移和植物生物学方面取得了显著进展。
尽管机器学习(ML)在单细胞组学中的应用前景广阔,但仍面临一系列持续挑战。数据质量和生物学相关性方面,实验方案缺乏标准化、平台特异性偏差以及生物样本的复杂性是主要障碍。过拟合和缺乏泛化性在临床研究中尤为普遍,小样本数据集和静态建模方法限制了模型的鲁棒性。机器学习模型的可解释性危机是一个核心限制,许多深度学习模型如同“黑盒”,在需要生成假设的医学研究中价值被削弱。机器学习应用中的“过度承诺与交付不足”现象也时有发生,例如无监督聚类方法可能产生误导性的结果。多模态数据(如流式细胞术与基因组学、转录组学)的整合对于理解复杂疾病机制至关重要,但仍处于早期阶段,面临着数据协调的挑战。最后,缺乏标准化的数据分析协议和模型基准测试框架阻碍了进展,影响了机器学习方法在临床研究中的可信度。
下一代深度学习架构有望整合分层注意力机制、几何深度学习和基于Transformer的表征,以解决细胞群体重叠的模糊性并检测超罕见表型。自监督对比学习框架有望通过解纠缠生物信号和实验伪影来革新未标注数据集的分析。虚拟细胞(virtual cell)系统和多尺度生物学模拟(multi-scale biology simulation)是一个关键前沿,通过将单细胞视为动态计算实体,可以模拟细胞行为和相互作用,并整合分子、细胞和组织水平模型。百亿亿次计算(exascale computing)和延迟优化工作流对于处理十亿级细胞数据集至关重要,需要稀疏感知算法和量化神经网络来实现实时分类。跨模态数据融合(cross-modal data fusion)和因果表征学习(causal representation learning)通过创建统一嵌入空间来协调流式细胞术、scRNA-seq和空间蛋白质组学数据,并结合因果发现算法来识别跨实验条件的不变细胞特征。临床级标准化(clinical-grade standardization)和符合法规的人工智能需要建立与IVDR/FDA-AI指南一致的严格验证框架。贝叶斯不确定性量化(Bayesian uncertainty quantification)和误差感知流程将在多个层次整合概率推理。最后,去中心化数据生态系统(decentralized data ecosystems)和富含元数据的存储库(metadata-rich repositories),如符合FAIR原则的知识图谱,有望促进大规模科学发现。
单细胞组学在基础模型、多模态整合和计算生态系统的推动下正在迅速发展,彻底改变了对细胞复杂性的研究。大规模预训练神经网络引入了单细胞分析,在多任务中实现了前所未有的精度。结合转录组学、表观基因组学、蛋白质组学和成像数据的创新多模态整合策略,使得构建全面的细胞图谱成为可能,从而更细致地理解细胞异质性和功能。尽管取得了这些进展,但在模型互操作性、标准化评估指标和数据协调方面仍然存在挑战,这强调需要持续的计算方法和数据标准创新。展望未来,因果推理和先进统计技术的整合将进一步提高单细胞分析的鲁棒性和临床适用性。随着计算能力的增长,百亿亿次计算平台和实时工作流的开发对于管理单细胞数据集日益增长的规模和复杂性至关重要。单细胞组学的这些变革性进展有望重塑我们对细胞生物学的理解,并推动精准医疗的创新,最终改善不同疾病患者的治疗结果。
 生物通微信公众号
生物通微信公众号
 生物通新浪微博
生物通新浪微博
今日动态 |
人才市场 |
新技术专栏 |
中国科学人 |
云展台 |
BioHot |
云讲堂直播 |
会展中心 |
特价专栏 |
技术快讯 |
免费试用
版权所有 生物通
Copyright© eBiotrade.com, All Rights Reserved
联系信箱:
粤ICP备09063491号


 生物通微信公众号
生物通微信公众号