
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于集合覆盖算法优化循环肿瘤DNA测序面板:结直肠癌早期检测的靶向基因筛选新策略
《Scientific Reports》:A set cover algorithm identifies minimal circulating tumour DNA sequencing targets for colorectal cancer detection
【字体: 大 中 小 】 时间:2025年11月21日 来源:Scientific Reports 3.9
编辑推荐:
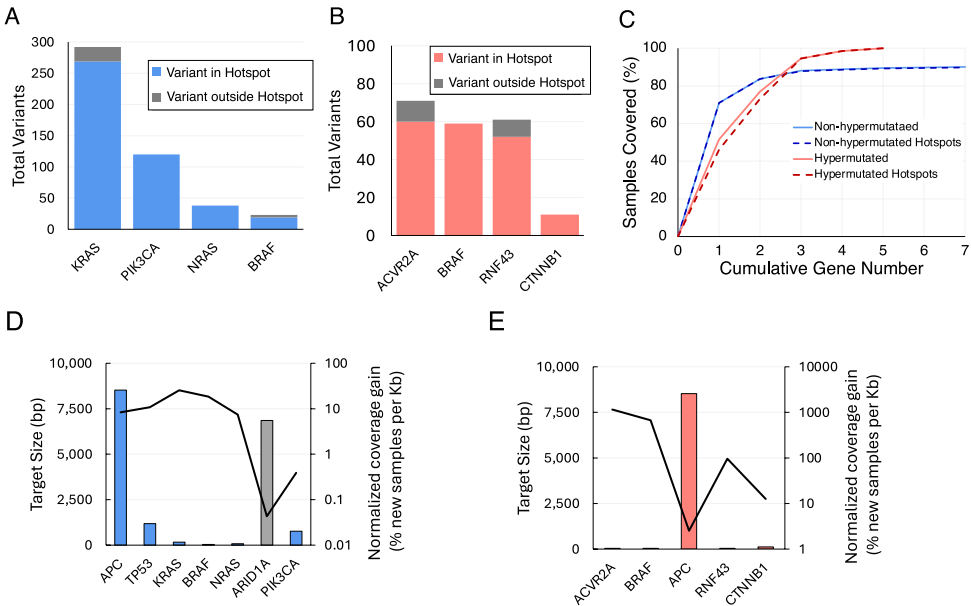
本研究针对循环肿瘤DNA(ctDNA)检测中因测序深度不足导致的早期癌症漏检难题,开发了OPTIC(Oncogene Panel Tester for Identifying Cancers)生物信息学流程。通过集合覆盖算法从2,940例结直肠癌(CRC)样本中筛选出APC、TP53、KRAS等9个核心基因的热点突变区域,构建仅10,975碱基的微型测序面板,覆盖96.3%的病例。该面板可实现超高深度测序(理论深度>72,000×),显著提升ctDNA检测灵敏度,为低成本、高精度的液体活检技术开发提供新范式。



 生物通微信公众号
生物通微信公众号
知名企业招聘