
-

生物通官微
陪你抓住生命科技
跳动的脉搏
新型隐孢子虫IOWA II株T2T基因组组装与注释:解析复杂亚端粒区域及跨研究兼容性基因标识
【字体: 大 中 小 】 时间:2025年06月20日 来源:Scientific Data 5.8
编辑推荐:
研究人员针对隐孢子虫(Cryptosporidium parvum)基因组组装难题,通过混合测序技术完成首个IOWA II株端粒到端粒(T2T)全基因组CpBGF,解析了8条染色体(9.26 Mb)及复杂亚端粒区域,采用与2004年参考基因组兼容的基因命名体系,整合Iso-Seq长读长转录组数据注释非编码RNA(ncRNA)和UTR边界。该研究为隐孢子虫病诊断、药物和疫苗开发提供关键资源,成果发表于《Scientific Data》。
隐孢子虫(Cryptosporidium parvum)是一种严重威胁人类和动物健康的肠道病原体,通过污染的水源和食物传播,尤其在免疫缺陷人群和婴幼儿中引发高死亡率。尽管其全球健康负担巨大,但基因组研究长期受限于亚端粒区域的复杂性和体外培养困难。现有基因组组装存在片段化、端粒缺失等问题,阻碍了致病机制研究和防控工具开发。
美国佐治亚大学等机构的研究团队利用混合测序策略,首次完成隐孢子虫IOWA II株CpBGF的端粒到端粒(T2T)基因组组装,获得9.26 Mb的8条染色体完整序列,解决了染色体1/7/8亚端粒重复区域的组装难题。通过保留与经典参考基因组(CpIOWA II)兼容的基因命名体系(如gp60基因ID从cgd6_1080调整为cpbgf_6001080),并整合PacBio Iso-Seq长读长转录组数据,新增766个长链非编码RNA(lncRNA)和87个小非编码RNA(sncRNA)注释,显著提升UTR边界准确性。该研究为隐孢子虫比较基因组学和功能研究建立新标准,相关成果发表于《Scientific Data》。
关键技术包括:(1)牛津纳米孔(ONT)长读长与Illumina短读长混合测序,采用NECAT和Flye双组装策略;(2)基于CpIOWA-ATCC基因组的Liftoff同源注释与BRAKER223从头预测结合;(3)利用4×108个子孢子提取的RNA进行PacBio Iso-Seq全长转录本分析;(4)通过OrthoFinder26验证基因保守性,建立跨研究基因ID映射表。
背景与摘要
研究揭示了隐孢子虫基因组中18S/28S rRNA基因、色氨酸合成酶β及MEDLE基因在染色体1/7/8亚端粒区的多拷贝现象,通过深度测序和生物信息学方法首次实现16个亚端粒区域和端粒的完整解析。
方法
采用酚-氯仿法从商业来源(Bunch Grass Farms)的子孢子中提取高分子量DNA,结合ONT MinION R9.4.1和MiSeq平台数据,经NextPolish18校正后获得N50达1.1 Mb的连续组装。RNA分析采用TAMA25算法,定义UTR需满足>90%转录本一致性。
数据记录
基因组数据存放于GenBank(ASM3523276v1),包含3925个蛋白编码基因,其中44个基因因染色体易位获得新ID。与CpIOWA-ATCC相比,CpBGF新增366个lncRNA注释,基因组功能区域覆盖率达88.61%。
技术验证
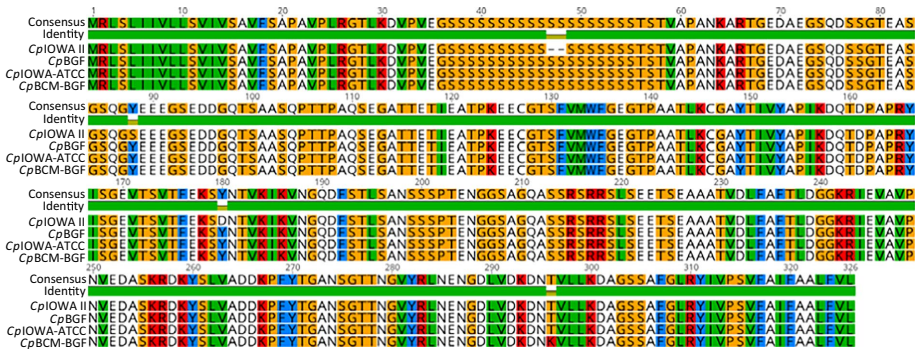
BUSCO评估显示96.2%的顶复门保守基因完整,Illumina读段比对率95%。所有预测蛋白以甲硫氨酸起始,较2004年参考基因组修正636个非标准起始密码子。GP60蛋白序列比对发现CpBCM-BGF存在第296位氨基酸变异(图1)。
比较分析
Dot plot显示CpBGF与CpIOWA II在染色体2/4/5存在易位(图2a),但与CpIOWA-ATCC/CpBCM-BGF保持共线性(图2b-c)。变异分析发现12个基因存在错义突变,如cgd7_5040的移码突变可能影响功能。

结论与意义
该研究建立的T2T基因组填补了隐孢子虫研究的三大空白:(1)首次完整解析亚端粒重复序列结构;(2)通过兼容性基因ID实现跨研究数据整合;(3)基于长读长转录组精确注释非编码调控元件。作为目前最完整的隐孢子虫参考基因组,CpBGF为病原体-宿主互作、耐药基因筛选及疫苗靶点发现提供不可替代的资源,其方法论对其它复杂基因组研究具有示范价值。
 生物通微信公众号
生物通微信公众号
知名企业招聘