
-

生物通官微
陪你抓住生命科技
跳动的脉搏
SeedSeg:基于图像分割的转基因种子计数工具加速T-DNA位点分离分析
【字体: 大 中 小 】 时间:2025年06月25日 来源:Plant Methods 4.7
编辑推荐:
为解决转基因植物研究中T-DNA插入位点分析耗时费力的问题,斯坦福大学团队开发了开源图像分析工具SeedSeg。该工具通过荧光/比色标记自动计数转基因与野生型种子,结合卡方检验判断T-DNA插入位点数,准确率高达99.78%。其创新性在于采用分水岭算法分离重叠种子,适配FastRed/RUBY等多种标记,为植物遗传工程研究提供高效解决方案。
在植物遗传学研究领域,转基因技术是探索基因功能和作物改良的核心手段。然而,一个长期困扰研究人员的难题是:农杆菌介导的转化常导致多拷贝T-DNA插入,这不仅可能引发转基因沉默现象,还会使后续遗传分析变得异常复杂。传统鉴定单拷贝插入株系的方法,如全基因组测序和Southern blot,不仅成本高昂,而手工计数荧光种子这种"原始"方法更是让研究人员在显微镜前数到眼花——据统计,约85%的拟南芥转化株含有多个T-DNA拷贝。
斯坦福大学Jennifer A. N. Brophy团队在《Plant Methods》发表的创新研究,带来了革命性的解决方案。他们开发的SeedSeg工具,只需将T2代种子倒在称量舟上拍摄荧光/明场图像,就能自动完成种子计数和遗传分离分析。这个看似简单的工具背后,隐藏着精妙的算法设计:通过动态调整径向阈值(radial threshold)适应不同种子大小,采用距离变换(Chamfer Distance transform)和分水岭算法(watershed algorithm)精准分离重叠种子,最后用卡方检验判断是否符合3:1的孟德尔分离比例。
关键技术包括:1)基于OpenCV的图像处理流程,分别处理荧光/明场图像;2)CIELAB色彩空间转换对比色标记(如RUBY)进行分析;3)参数自适应系统,默认径向阈值比为0.4时可实现零误差计数;4)集成Streamlit的交互式网页界面(https://seedseg-brophy.streamlit.app/),支持实时调整亮度/尺寸参数。测试中使用Leica THUNDER显微镜拍摄的拟南芥FastRed种子图像,准确率达99.78%,显著优于PlantCV等现有工具。
结果与讨论
精准计数性能
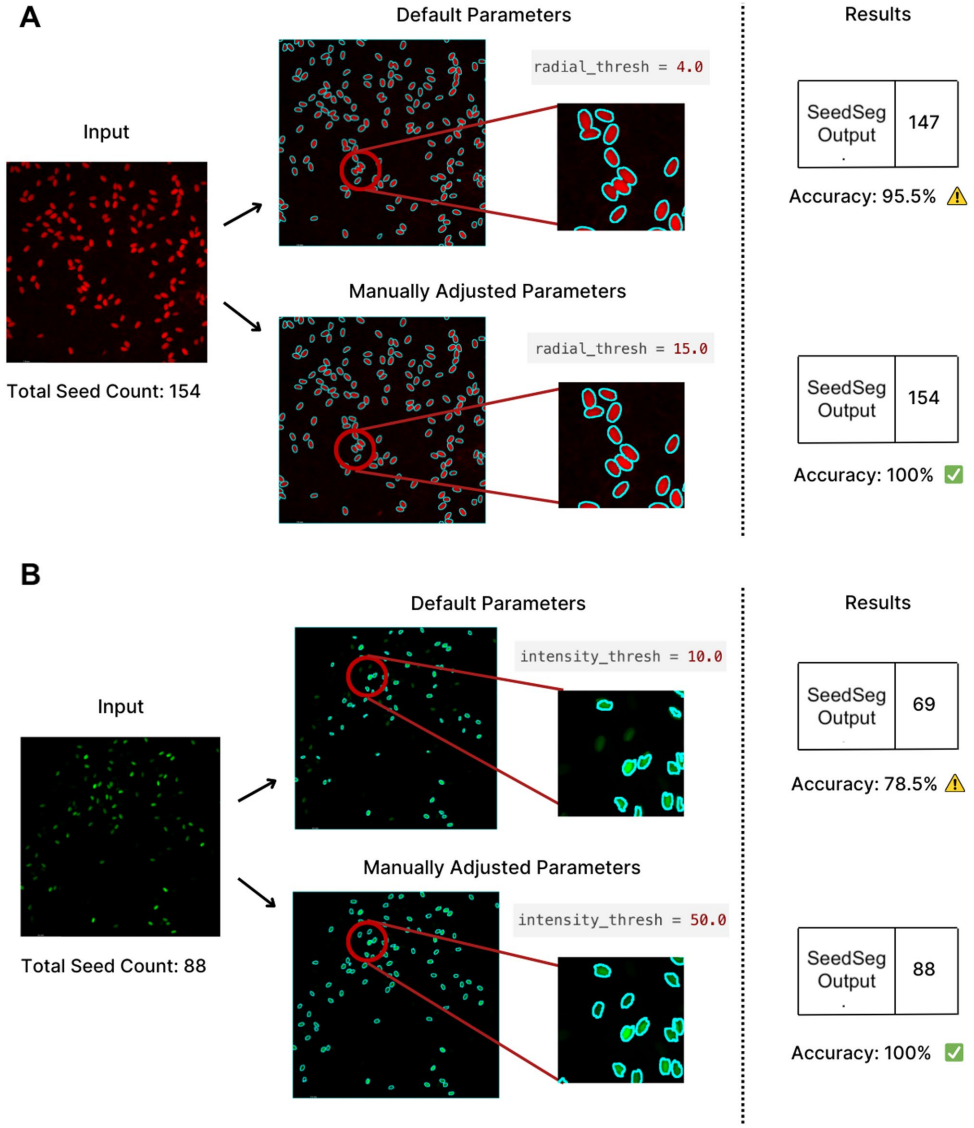
在基准测试中,SeedSeg对荧光图像的计数误差仅0.22%,明显低于PlantCV的4.53%。通过调整径向阈值比(0.45)和强度阈值(13.0),27/28的测试图像可实现零误差计数。如图2所示,增加径向阈值从4.0到15.0能有效解决种子重叠问题。
多标记兼容性
如图3所示,SeedSeg成功识别FastGreen荧光标记(ET GFP滤光片)和RUBY比色标记(基于b*值阈值)。对荠菜(Capsella rubella)种子的分析证明其跨物种适用性,只需调整曝光参数即可适配不同显微镜配置。

技术优势
相比PlantCV的简单二值化处理,SeedSeg通过距离变换和分水岭算法实现重叠种子分离。其创新性参数自适应系统能根据种子中值面积动态调整检测阈值,而交互式网页界面允许研究者直观验证分割结果(如图1所示的分步处理流程)。

这项研究的意义不仅在于技术突破,更开创了转基因株系筛选的新范式。通过将传统需要数天的手工计数缩短为几分钟的自动化分析,SeedSeg显著加速了单拷贝转基因株系的获得。其开源特性(GitHub仓库:https://github.com/santiaghini/seedseg)和跨平台兼容性,使其有望成为植物遗传学研究的标准工具。未来,该技术可进一步拓展至作物育种和大规模突变体库筛选,为粮食安全挑战提供新的技术支撑。
 生物通微信公众号
生物通微信公众号
知名企业招聘