
-

生物通官微
陪你抓住生命科技
跳动的脉搏
利用空气环境DNA测序技术监测农作物病原体与害虫:一种非靶向性监测新方法
【字体: 大 中 小 】 时间:2025年06月29日 来源:iScience 4.6
编辑推荐:
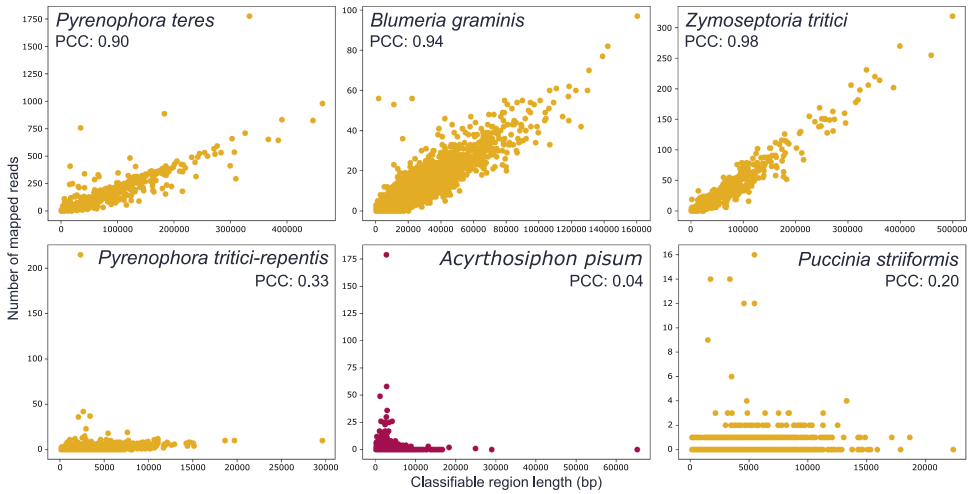
本研究针对传统农作物病虫害监测方法存在靶向性局限的问题,创新性地采用空气环境DNA(eDNA)宏基因组测序技术,实现了对多种作物病原体和害虫的非靶向监测。研究团队通过分析瑞典南部农田空气过滤器样本,证实空气中病原体DNA丰度与田间观测的作物损害程度显著相关(Pearson相关系数>0.9),同时揭示了参考基因组数据库质量对分类准确性的关键影响。该成果为建立大规模农业生物安全预警系统提供了新技术路径,发表于《iScience》期刊。
全球农作物每年因病虫害导致的产量损失高达20%-30%,传统监测方法依赖人工田间调查和靶向分子检测,难以实现大规模早期预警。尤其面对气候变化背景下病虫害传播格局的改变,农业系统亟需开发新型监测技术。瑞典于默奥大学Per Stenberg团队创新性地将空气环境DNA(eDNA)分析技术引入农业监测领域,通过高通量测序解析空气中游离的遗传物质,建立了覆盖真菌、昆虫、卵菌等多类生物的非靶向监测体系。
研究采用瑞典国防研究院(FOI)在斯科讷省建立的空气监测网络样本,选取2007年作物生长关键期的9周玻璃纤维滤膜(孔径0.2μm,累计过滤量>100万m3)。通过改良PowerWater试剂盒提取DNA后,使用Illumina HiSeq 2500平台进行150bp双端测序。数据分析采用Kraken2构建包含病原体近缘种的特异数据库(805GB),结合BBMap比对和NEAT模拟读数验证分类可靠性。
主要研究发现
空气eDNA实现多类病虫害检测
通过分析1.9×109条测序读数,在空气样本中检测到264种已知病原体/害虫所属的科级分类单元。其中真菌类(如子囊菌门)检出率最高(82%),线虫类最低(31%),反映不同生物类群的DNA释放效率差异。

数据库质量决定检测灵敏度
在13种当地已报道的基因组已知病原体中,11种被成功检出。但信号强度差异显著:大麦网斑病菌(Pyrenophora teres)读数达174,190条,而禾谷缢管蚜(Rhopalosiphum padi)仅5条。通过人工合成读数测试发现,参考基因组污染会导致严重误分类——条锈菌(Puccinia striiformis)读数被错误归类至螨类(Medioppia subpectinata)。
田间观测与DNA信号时空匹配
大麦白粉病(Blumeria graminis)的空气DNA信号早于田间病斑出现7-10天,预示eDNA可用于早期预警;而豌豆蚜(Acyrthosiphon pisum)信号滞后于虫口高峰,符合其有翅型扩散的生物学特性。
讨论与展望
该研究首次证实空气eDNA宏基因组测序可同步监测农作物真菌病、虫害等多元威胁,突破传统方法单次检测只能针对特定类群的技术瓶颈。通过整合现有空气质量监测网络(如欧洲EMEP计划),该技术有望以较低成本实现大陆尺度的农业生物安全监控。当前限制主要来自参考基因组覆盖度不足——研究中30%的田间观察到物种因缺乏高质量基因组而漏检。未来随着全球基因组计划推进(如Earth BioGenome Project),该技术灵敏度将显著提升。
研究团队建议建立两级监测体系:通过空气eDNA宏基因组扫描发现潜在威胁后,再采用qPCR等靶向技术进行田间验证。这种策略尤其适用于检疫性有害生物(如EPPO A1名录物种)的早期预警。该技术路线已获瑞典Formas基金会持续资助,正在北欧小麦茎锈病(Puccinia graminis)监测中开展应用示范。