
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于基因行为特征网络的富集分析揭示COVID-19重症特异性免疫疾病通路
【字体: 大 中 小 】 时间:2025年06月29日 来源:Bioinformatics 4.4
编辑推荐:
本研究针对基因网络分析中生物学意义解读的挑战,开发了基于基因行为特征的网络富集分析方法(GbNEA),通过整合基因表达水平、边权重和结构模式等网络特征,系统识别表型特异性基因网络中的功能通路。应用该方法分析日本COVID-19工作组1102例全血RNA-seq数据,发现无症状/重症样本网络富集于"系统性红斑狼疮"通路,轻症样本则与"炎症性肠病"、"原发性免疫缺陷"等通路相关,并鉴定出CXCL8、S100A9等关键枢纽基因。该研究为理解COVID-19严重程度的免疫机制提供了新视角。
在理解复杂疾病机制的研究中,基因网络分析已成为重要工具。然而,面对包含上万基因和大量连接的大规模网络,如何系统解读其生物学意义仍是重大挑战。传统方法如基因集富集分析(GSEA)主要关注差异表达基因,难以捕捉网络层面的分子互作特征。与此同时,COVID-19的临床异质性,特别是免疫系统在疾病严重程度中的作用机制仍不明确,亟需新的分析方法来揭示其背后的网络级调控特征。
日本COVID-19工作组的研究人员开发了基于基因行为特征的网络富集分析方法(GbNEA),该方法创新性地整合了基因表达水平、边权重(β?j)和边结构(Jaccard距离)等多维网络特征,通过统计量Wdj(t)(C,N)量化表型特异性网络差异。研究首先通过模拟实验验证了GbNEA在识别癌症相关通路中的优越性能,其准确率达0.96,显著优于传统方法。随后将其应用于1102例COVID-19全血转录组数据,按疾病严重程度分为无症状(n=71)、轻症(n=241)、重症(n=404)和极重症(n=303)四组,构建了588个基因(含294个免疫疾病相关基因)的表型特异性网络。
关键技术方法包括:(1)使用弹性网络(elastic net)估计有向基因网络,通过BIC准则选择正则化参数λ;(2)设计加权差异统计量Wdj(t)整合调控效应(rjC/rjN)和边结构差异(dJIj);(3)采用改进的Kolmogorov-Smirnov检验计算通路富集分数;(4)基于日本COVID-19任务组提供的全血RNA-seq数据,分析不同严重程度样本的免疫疾病通路富集特征。
研究结果部分显示:
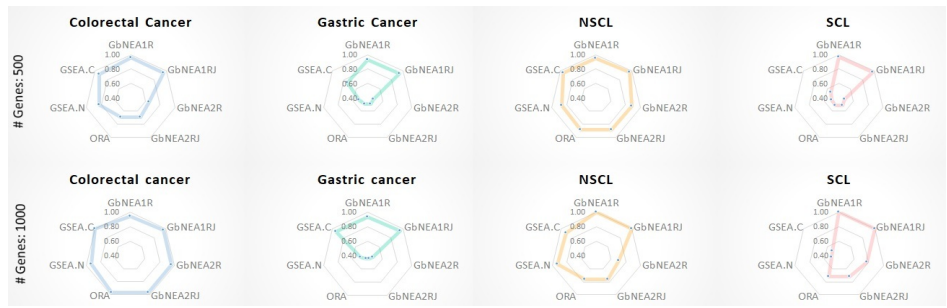

讨论部分指出,GbNEA通过整合多维网络特征,克服了传统方法仅关注基因表达水平的局限,为复杂疾病的机制研究提供了新范式。在COVID-19应用中,不仅验证了已知生物标志物(如CXCL8、S100A9)的重要性,还揭示了严重程度特异的免疫通路差异,特别是无症状/重症样本与SLE通路的关联,以及轻症样本中IBD、PID通路的富集,这些发现为理解COVID-19的免疫异质性提供了新视角。研究局限性包括计算效率有待优化,以及需要实验验证网络预测结果。未来可将该方法扩展至其他疾病领域,如癌症研究,并整合多组学数据进一步提升网络推断的可靠性。
 生物通微信公众号
生物通微信公众号
知名企业招聘