
-

生物通官微
陪你抓住生命科技
跳动的脉搏
长读长测序揭示太平洋牡蛎基因组结构变异图谱及其在生长与抗病性状中的生物技术潜力
【字体: 大 中 小 】 时间:2025年06月29日 来源:ICES Journal of Marine Science 3.1
编辑推荐:
本研究针对太平洋牡蛎(Crassostrea gigas)基因组结构变异(SV)检测的技术瓶颈,通过整合PacBio长读长测序与五种比对算法(LRA/Minimap2/NGMLR/Vulcan/WinnowMap),首次构建了染色体水平的SV图谱。研究发现193355-219501个SV位点覆盖基因组31.2%-35.2%,定位到骨形态发生蛋白(BMP)和超氧化物歧化酶(SOD)等关键基因,为海洋生物技术中的基因组编辑和替代亲本技术开发提供新思路。
海洋生物资源开发正面临遗传改良的技术瓶颈,尤其在具有重要经济价值的太平洋牡蛎(Crassostrea gigas)育种领域。传统短读长测序技术难以捕捉>50bp的结构变异(SV),而这类变异恰恰与生长速度、抗病性等关键农艺性状密切相关。澳大利亚国立大学土著基因组学中心与韩国中央大学的研究团队在《ICES Journal of Marine Science》发表的研究,通过创新性地整合长读长测序与多算法比对策略,绘制了迄今最完整的牡蛎SV图谱。
研究采用两项关键技术:①基于PacBio长读长测序数据,交叉使用C. gigas1(Qi et al. 2021)和C. gigas2(Penaloza et al. 2021)两个染色体水平基因组作为参考;②运用五种比对工具(LRA/Minimap2/NGMLR/Vulcan/WinnowMap)和两种SV检测软件(cuteSV/Sniffles2),通过SURVIVOR合并分析确保结果可靠性。样本来源于NCBI数据库收录的太平洋牡蛎基因组数据(SRA登录号PRJNA598006和PRJEB35351)。
【全基因组比对与SV特征】
通过MUMmer软件进行全基因组比对,发现11263-11520个SV事件。其中插入(INS)和缺失(DEL)占主导(97.8%-98.2%),平均SV长度达953-1050bp。特别值得注意的是,Minimap2在计算效率与SV检出率间展现出最佳平衡,而NGMLR虽检出率低但能识别独特SV类型。
【关键基因关联分析】
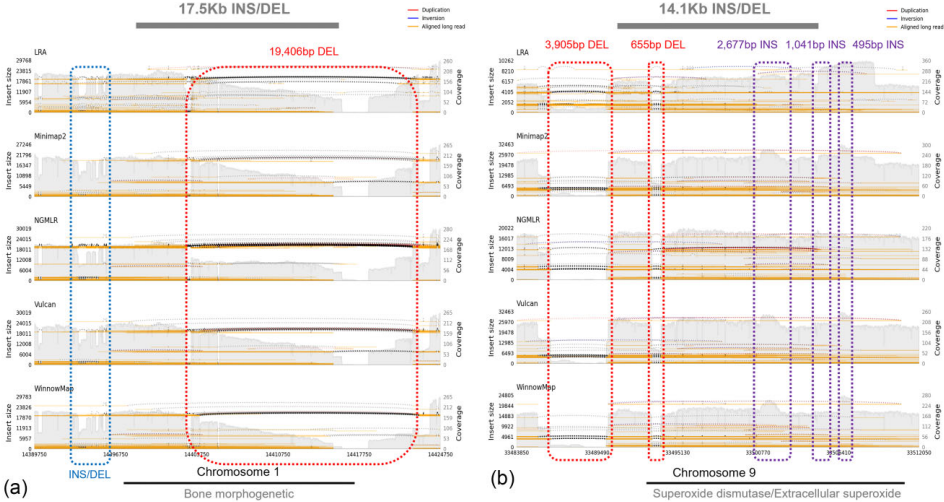
【超大变异体发现】
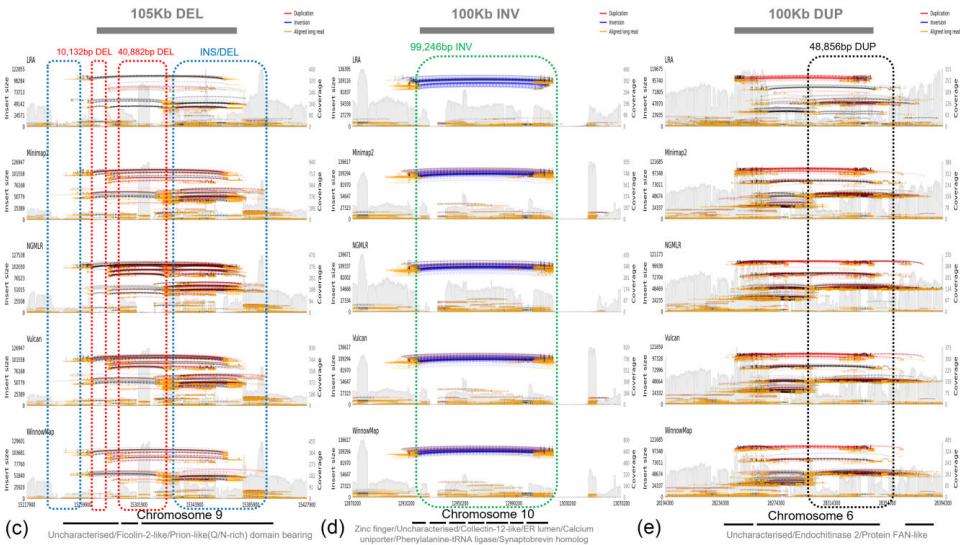
讨论部分强调了三项突破:①建立长读长SV检测的金标准流程,推荐400bp作为最优合并阈值;②揭示SV在牡蛎基因组中占比高达35%,远超短读长研究预期;③定位的BMP和SOD基因SV为分子育种提供靶点。该研究不仅推动贝类基因组进化认知,更为开发基因组编辑(如CRISPR)和替代亲本技术奠定基础,对实现水产养殖可持续发展具有重要战略意义。
 生物通微信公众号
生物通微信公众号
知名企业招聘