
-

生物通官微
陪你抓住生命科技
跳动的脉搏
高精准长读长HiFi测序:解锁小麦抗病基因密码,引领育种新革命
【字体: 大 中 小 】 时间:2025年06月18日 来源:基因有限公司
编辑推荐:
PacBio HiFi测序能生成长度达10–25 kb、准确率高达99.9%的Reads,特别适用于复杂结构区段的解析,极大提升了结构变异(SV)、拷贝数变异(CNV)与嵌合融合结构的识别能力,为植物复杂基因组解析和性状基因克隆提供了革命性解决方案。
测序技术的不断革新正深刻改变着生命科学的研究格局。以Illumina为代表的二代测序(NGS)长期主导基因组研究,依赖其高通量、低成本的特点,在动植物全基因组重测序、转录组表达谱分析等方面取得显著成效。然而,二代测序固有的短读长(通常为150-300 bp)限制,使其在处理高重复、结构变异丰富的复杂基因组(如小麦、甘蔗、油菜)时面临重大挑战,尤其是在抗病基因如NLR(nucleotide-binding leucine-rich repeat)类基因聚集的区域,易导致组装碎片化、错配以及关键功能基因的遗漏。
PacBio HiFi测序能生成长度达10–25 kb、准确率高达99.9%的Reads,特别适用于复杂结构区段的解析,极大提升了结构变异(SV)、拷贝数变异(CNV)与嵌合融合结构的识别能力,为植物复杂基因组解析和性状基因克隆提供了革命性解决方案。
在植物育种领域,特别是抗病育种中,精确、高效地鉴定与克隆抗病基因至关重要。植物免疫系统中的NLR类受体蛋白,常在基因组中呈簇状分布、拷贝多样、结构复杂,这些区域往往是短读长测序无法有效覆盖的“难点区域”。HiFi测序凭借其卓越的解析力,已成为当今植物功能基因组学和抗病育种研究中不可或缺的技术核心。下面将通过两项基于HiFi测序的最新研究,详细阐述其在小麦抗病基因发掘中的实际应用与优势。
由中国科学院遗传与发育生物学研究所的刘志勇研究员领导的团队及其合作者于2025年6月9日在《Nature Genetics》上发表了两篇研究论文,揭示了从小麦野生近缘种野生二粒小麦中发现的白粉病、条锈病新抗病基因模式。
TdCNL1/TdCNL5基因对赋予白粉病抗性

Zhu等人基于HiFi测序技术成功解析了野生二粒小麦IW170中一个对白粉病具有抗性的关键位点MLIW170/PM26。他们发现该位点由一对遗传连锁的非典型NLR类基因对TdCNL1和TdCNL5构成,该基因对共同作用,赋予小麦对白粉病的广谱抗性。
研究首先利用PacBio HiFi测序技术对IW170样本进行测序,生成141.4 Gb高质量HiFi Reads,约14×全基因组覆盖。利用Hifiasm进行de novo组装后,获得3,208条contig,N50达9.4 Mb,为基因克隆提供了坚实基础。在比对Zavitan、Svevo、CS等参考基因组后,识别出一个4.07 Mb的候选区域,包含8个NLR类基因和2个PGAM样基因。图位克隆和PacBio HiFi序列分析表明,TdCNL1编码一个含有非典型卷曲螺旋结构域的NLR蛋白(CNL),该蛋白与一个新的钾依赖性钠钙交换体整合结构域融合,而TdCNL5编码一个典型的CNL蛋白。
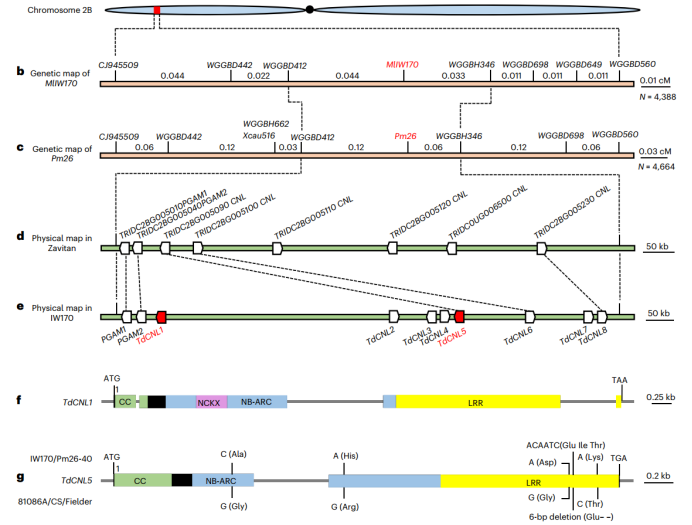
图1. MLIW170/PM26的图位克隆
后续通过EMS诱变、VIGS基因沉默实验证明,TdCNL1和TdCNL5协同作用是抗性所必需的,任何单独失活都会导致感病。而转基因实验验证显示,仅导入TdCNL1即可获得抗性增强,但与TdCNL5共转效果更优。这揭示出非典型-典型NLR组合的新型抗性机制。
HiFi测序在该研究中的贡献不止于基因定位,更在于高分辨率地揭示了该区域的结构变异(SV)、存在缺失(PAV)与拷贝数多态性(CNV),并在该区段中识别出非典型融合结构的CNL基因。这种对抗性机制的新认知正是得益于HiFi测序对复杂区域的深度解析能力。
参考文献:https://doi.org/10.1038/s41588-025-02208-z
TdNLR1/TdNLR2基因对赋予条锈病抗性

同样,Hu等人利用HiFi测序结合图位克隆和基因功能验证方法,在野生二粒小麦TD121中鉴定并克隆出控制条锈病抗性的YrTD121位点。该位点由头对头排列的TdNLR1和TdNLR2两个NLR类基因组成,二者功能互补,共同赋予作物高水平抗性。
研究利用PacBio HiFi测序技术对TD121样本进行全基因组测序,生成超过116 Gb数据,通过组装获得总长度达11.09 Gb,contig N50达3.06Mb的高质量序列。通过比对,YrTD121抗性位点被锚定于1BS染色体的一个约1.1 Mb区间(图2b–d)。该区域成功组装出3个非重叠contig(总长达1.18 Mb),包含7个抗病相关候选基因(图2e),其中两个为NLR类:TdNLR1与TdNLR2,呈头对头排列。
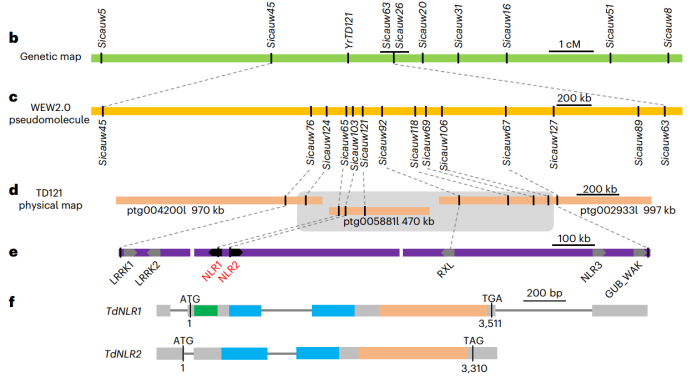
图2. YrTD121的图位克隆。
功能分析显示,TdNLR1编码典型CC-NLR蛋白,具备自我激活与细胞死亡诱导能力;而TdNLR2则编码非典型NLR,虽不具备融合域,但其功能缺失将严重削弱TdNLR1的抗性表达。通过EMS突变体、VIGS沉默实验、CRISPR-Cas9敲除实验及转基因补偿验证,研究全面确立了该基因对在抗条锈病过程中的功能模式。值得强调的是,该NLR基因对的空间排列方式(头对头)与功能互作机制,为植物免疫受体进化提供了新模型,也表明HiFi测序能在多样化的基因结构解析中发挥关键作用。
参考文献:https://doi.org/10.1038/s41588-025-02207-0
赋能育种
野生二粒小麦是现代面包小麦的祖先,通过长期适应复杂环境积累了丰富的遗传变异。然而,野生二粒小麦中的MlIW170/Pm26和 YrTD121位点仅在少数野生二粒小麦种群中存在,并未参与面包小麦的驯化或进化过程。研究人员通过将野生二粒小麦与高产面包小麦品种杂交,并采用分子标记辅助选择进行连续回交,成功培育出携带MlIW170/Pm26和YrTD121的高产抗病种质资源。这些发现为培育广谱多抗小麦品种提供了重要的抗病基因资源和理论依据。
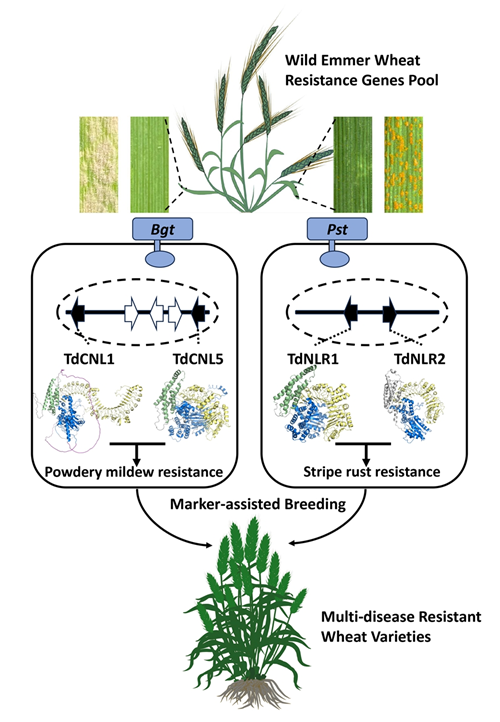
图3. 野生二粒小麦抗白粉病基因Pm26和抗条锈病基因YrTD121的克隆及其育种应用(图片来源:IGDB)
HiFi测序推动精准育种迈向新纪元
上述刘志勇团队的两项研究充分展示了HiFi测序在植物复杂基因组结构解析、抗病基因克隆与功能鉴定中的巨大潜力和独特价值。值得一提的是,这并不是该团队首次利用HiFi测序发现小麦抗性基因,2024年在发表于《Nature Communications》的“A membrane associated tandem kinase from wild emmer wheat confers broad-spectrum resistance to powdery mildew”中,他们通过图位克隆、HiFi测序、诱变和转基因实验分析了野生二粒小麦中编码跨膜结构域串联激酶(WTK7-TM)的广谱白粉病抗性基因Pm36。而2025年3月,刘志勇研究员领衔的植物免疫团队及合作者,在《Science》发表了一篇题为 “A wheat tandem kinase and NLR pair confers resistance to multiple fungal pathogens” 的研究论文,揭示了植物免疫系统中串联激酶与非典型NLR蛋白协同抵御病原菌入侵的全新免疫机制。这些发现不仅丰富了我们对植物免疫系统运作的理解,还为开发新的广谱抗病小麦品种提供了宝贵的资源和理论支持。
随着测序成本持续下降与生物信息分析工具不断优化,HiFi测序将在泛基因组构建、种质资源库挖掘、精准分子设计育种中发挥越来越关键的作用。其与多组学数据整合(转录组、甲基组、表型组)及人工智能辅助分析相结合,或将加速作物育种从经验驱动走向数据驱动,为保障全球粮食安全、培育新型抗逆高产作物提供坚实的技术支撑与理论基础。