
-

生物通官微
陪你抓住生命科技
跳动的脉搏
非洲人畜界面抗菌素耐药性传播的系统评价:基因组流行病学框架的构建与应用
【字体: 大 中 小 】 时间:2025年07月01日 来源:npj Antimicrobials and Resistance
编辑推荐:
本研究系统评价了非洲地区人畜界面抗菌素耐药性(AMR)传播的34项研究(18,604份样本),揭示了当前AMR传播推断存在的定义模糊、研究设计局限(64.71%基于大肠杆菌)、数据分辨率不足等问题,并提出整合基因组流行病学的高分辨率框架,为优化AMR国家行动计划提供关键证据。
抗菌素耐药性(AMR)已成为全球公共卫生危机,世界卫生组织非洲区域2019年因AMR相关死亡高达105万例。尽管AMR的进化机制研究已较深入,但其在人类、动物和环境间的传播规律仍不明确,尤其在非洲等低收入国家,人畜密切接触与薄弱的监测体系加剧了防控难度。当前AMR干预策略多聚焦于减少抗生素选择压力(如抗生素管理),却缺乏针对传播链的精准证据支持。这种"重进化、轻传播"的研究失衡,使得AMR控制效果大打折扣。
为破解这一困局,由Frank Chilanga领衔的国际研究团队在《npj Antimicrobials and Resistance》发表系统评价,首次全面剖析非洲人畜界面AMR传播推断的证据质量。研究人员检索77年间3576篇文献,最终纳入34项研究(2013-2024年),涵盖埃及、乌干达等14个非洲国家的18,604份样本,通过PRISMA标准进行质量评估,并创新性提出基因组流行病学框架。
研究采用多数据库系统检索(PubMed/Scopus/Web of Science等),运用随机效应模型进行meta分析,关键方法包括:1) 基于STROBE声明设计质量评估工具;2) 采用R语言RMeta包计算样本利用率的加权比例;3) 通过QGIS绘制地理分布图;4) 整合全基因组测序(WGS)数据与表型耐药谱分析。
研究结果揭示四大核心发现
1. 证据质量存在显著缺陷
纳入研究全部为横断面设计,仅11项采用随机抽样,23项依赖非概率抽样。虽然47%研究使用WGS(如核心基因组SNP分析),但样本代表性不足——中位仅16.58%采集样本用于传播推断(图4)。
2. 大肠杆菌主导传播推断
79.41%研究以革兰阴性菌为基础,其中64.71%聚焦大肠杆菌,而金黄色葡萄球菌仅占14.71%。这种病原体单一性可能低估了AMR传播的复杂性。
3. 传播定义缺乏标准化
88.24%研究声称发现人畜传播,但判断标准混乱:50%WGS研究使用SNP阈值(如核心基因组MLST),另有研究依赖脉冲场凝胶电泳(PFGE)或表型共现。仅10项尝试确定传播方向,其中3项依据毒力基因(如人源scn/sak基因在山羊分离株中的检出)。
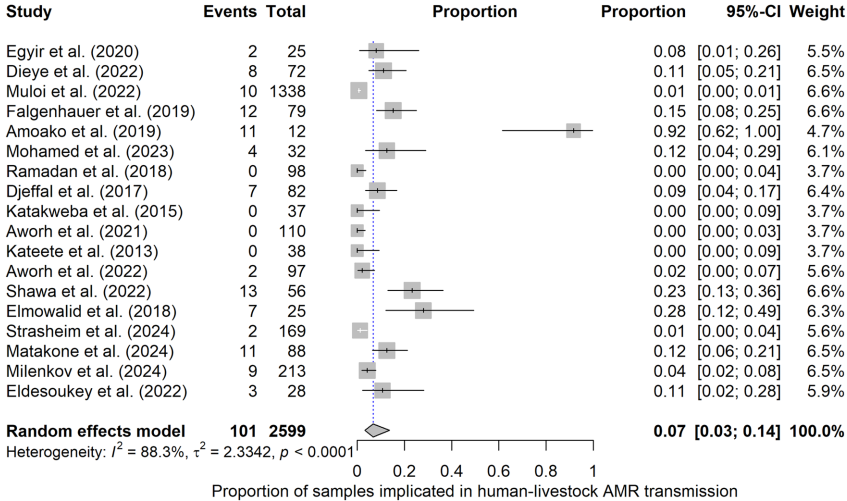
4. 定量数据严重缺失
通过18项研究的meta分析估算,仅7%(95%CI:3-14%)个体明确涉及传播事件(图5)。无一研究提供传播频率的定量指标或不确定性度量。
创新框架破解传播推断困境
作者提出五步递进式解决方案:1) 流行病学情境定义(优化样本量与时空设计);2) 哨兵菌选择(拓展至革兰阳性菌);3) 表型耐药检测(推荐MIC法替代纸片法);4) 基因分型(WGS为主,长读长测序辅助质粒追踪);5) 推断优化(开发概率模型量化传播置信度)。该框架特别强调需建立物种特异性SNP阈值,如针对ST612-CC8型MRSA克隆的遗传距离标准。
这项研究首次系统揭示非洲AMR传播研究的证据缺口,为《全球AMR行动纲领》在低收入国家的实施提供方法论革新。通过将基因组流行病学与传统流行病学深度融合,不仅推动AMR研究从"定性推测"迈向"定量推断",更为靶向干预(如重点防控大肠杆菌ST69克隆的跨种传播)奠定了科学基础。未来需在病原多样性、纵向设计和成本效益分析等方面持续突破,才能真正实现"One Health"视角下的AMR综合治理。
 生物通微信公众号
生物通微信公众号
知名企业招聘