
-

生物通官微
陪你抓住生命科技
跳动的脉搏
罗宾逊克鲁索岛刺龙虾Jasus frontalis的首个核与线粒体基因组图谱:为可持续渔业管理提供基因组学基础
【字体: 大 中 小 】 时间:2025年07月02日 来源:BMC Genomics 3.5
编辑推荐:
本研究针对智利胡安·费尔南德斯群岛特有经济物种Jasus frontalis的基因组资源匮乏问题,通过低深度测序技术首次解析其核基因组大小(1.42-1.65 Gbp)、线粒体基因组结构(15,514 bp)及转座元件组成(41-64%),并基于13个线粒体PCGs建立Achelata下目系统发育框架,为这一过度捕捞物种的保育策略和渔业管理提供了关键分子标记。论文创新性采用k-mer分析和DnaPipeTE等生物信息学工具,填补了东南太平洋重要渔业资源的基因组空白。
在东南太平洋的胡安·费尔南德斯群岛,一种名为Jasus frontalis的刺龙虾正面临严峻的生存挑战。这种最大体长可达48厘米的经济物种支撑着当地利润丰厚但不可持续的手工渔业,近年捕获量持续下降的迹象表明种群可能已过度开发。更令人担忧的是,尽管该物种生活史研究相对充分,但基因组资源极度匮乏——此前仅有的分子数据来自线粒体cox1基因片段和简化基因组测序(DaRTseq),严重制约了科学管理和保护策略的制定。
针对这一现状,由克莱姆森大学J. Antonio Baeza领衔的国际研究团队在《BMC Genomics》发表了一项开创性研究。研究人员采用Illumina HiSeq X Plus平台获得31,202,775条双端测序 reads,通过创新的生物信息学分析方法,首次绘制了这种海洋经济物种的基因组图谱。研究团队开发的技术路线极具特色:运用k-mer频谱分析估算核基因组大小,采用GetOrganelle从头组装线粒体基因组,利用DnaPipeTE解析转座元件组成,最后基于13个线粒体蛋白编码基因(PCGs)构建了Achelata下目的系统发育框架。
关键技术方法包括:(1)使用KMC3和GenomeScope2进行k-mer频谱分析,设置13种k-mer长度(18-54 bp)估算基因组参数;(2)采用Trinity组装和RepeatMasker注释的DnaPipeTE流程分析核基因组重复序列;(3)以J. edwardsii线粒体为种子,通过GetOrganelle完成15,514 bp线粒体基因组组装;(4)使用MITOS2和RNAfold分别进行基因注释和tRNA二级结构预测;(5)基于13个PCGs的氨基酸序列,采用IQ-TREE进行最大似然法系统发育分析。
基因组特征分析揭示:

线粒体基因组解析发现:

系统发育分析确立:
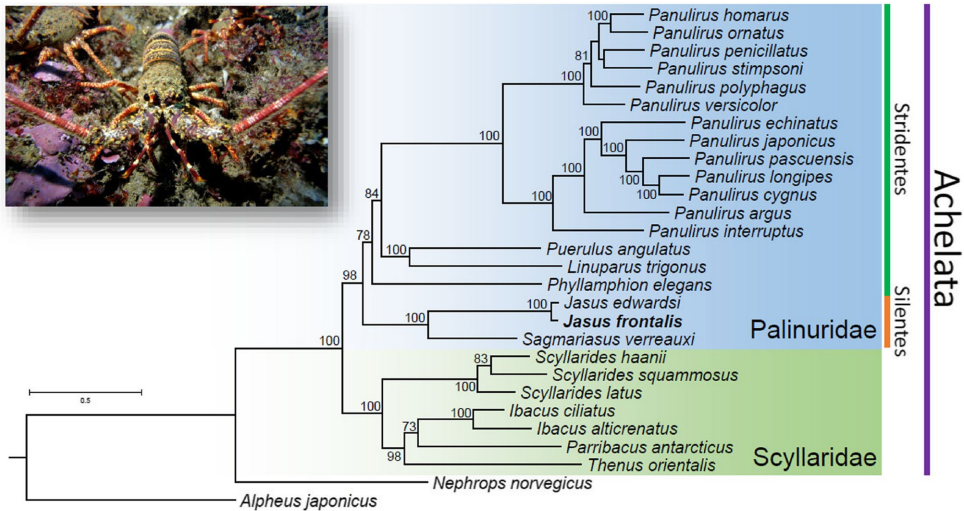
这项研究的意义远超常规基因组调查。首先,1.65 Gbp的基因组下限估计为后续染色体级别组装奠定基础;线粒体基因组可作为环境DNA(eDNA)监测的参考序列;转座元件图谱则为理解龙虾对环境适应的分子机制提供新视角。尤为重要的是,研究揭示了k-mer分析在大型基因组中的局限性,为未来海洋无脊椎动物基因组研究提供了方法学警示。这些发现不仅助力J. frontalis的种群管理和市场溯源,更丰富了我们对Achelata下目基因组演化的认知,为海洋生物多样性保护提供了分子工具包。
 生物通微信公众号
生物通微信公众号
知名企业招聘