
-

生物通官微
陪你抓住生命科技
跳动的脉搏
高分辨率小鼠脑立体定位图谱:单细胞水平的全脑三维空间定位新工具
【字体: 大 中 小 】 时间:2025年07月04日 来源:Nature 50
编辑推荐:
本研究团队通过连续微光学切片断层扫描技术,构建了首个具有1-μm各向同性分辨率的Nissl染色小鼠脑图谱STAM,解决了传统二维图谱分辨率不足、三维重建不精确的难题。该研究定义了916个脑区三维形貌,开发了支持任意角度切片生成、脑片配准和神经环路映射的信息学平台,为单细胞水平脑图谱研究提供了重要工具,相关成果发表于《Nature》。
在神经科学研究中,精确的脑结构定位犹如绘制"大脑地图",是理解神经环路和功能的基础。然而传统的小鼠脑立体定位图谱存在三大痛点:二维平面间隔过大(通常数百微米)、不同方向切片来自不同样本导致结构不一致、缺乏单细胞水平分辨率。这些问题严重制约了空间转录组和单神经元形态等前沿研究的空间定位需求。
为解决这一难题,华中科技大学的研究团队通过改进的Nissl染色方法和微光学切片断层扫描(MOST)技术,获得了0.35×0.35×1 μm3分辨率的全脑三维数据集。经过处理后构建出各向同性1-μm分辨率的立体定位地形图谱STAM,包含916个脑区结构的三维形貌,首次实现了从任意角度生成1-μm分辨率的脑切片图像。相关研究成果发表在《Nature》杂志,标志着脑图谱研究进入单细胞分辨率新时代。
关键技术包括:(1)改进的Nissl染色与MOST技术获取全脑数据集;(2)多模态图像配准整合免疫组化、原位杂交等数据;(3)基于颅内和颅骨标志点的空间坐标系统;(4)三维表面重建算法构建脑区模型;(5)开发支持脑片配准、神经环路映射的在线平台。使用1只C57BL/6J小鼠获取基础数据集,22只转基因小鼠提供特定神经元分布数据。
研究团队获得的MOST-Nissl数据集尺寸达11,400×9,000×14,000像素,通过观察细胞层状模式变化确定皮质区域边界,如视网膜区域与初级体感区的分界。连续1-μm轴向步进可精确追踪脑结构的出现与消失,如隔核三角区从隔核内侧开始出现至背侧穹窿腹侧消失的全过程。

整合多种标记数据(如Camk2-Cre神经元分布)辅助划分脑区,计算皮质深度细胞构筑特征。海马结构参考已有文献通过非线性配准引入,平均Dice评分>0.8。最终构建包含700个冠状面、256个矢状面和367个水平面的可视化平台,每个平面投影厚度20 μm。
通过表面重建算法保留精细解剖特征,如丘脑纤维穿透孔、嗅结节三层结构(含Calleja岛)。体感区不仅可视化三维形态,还将第二体感区细分为六层。
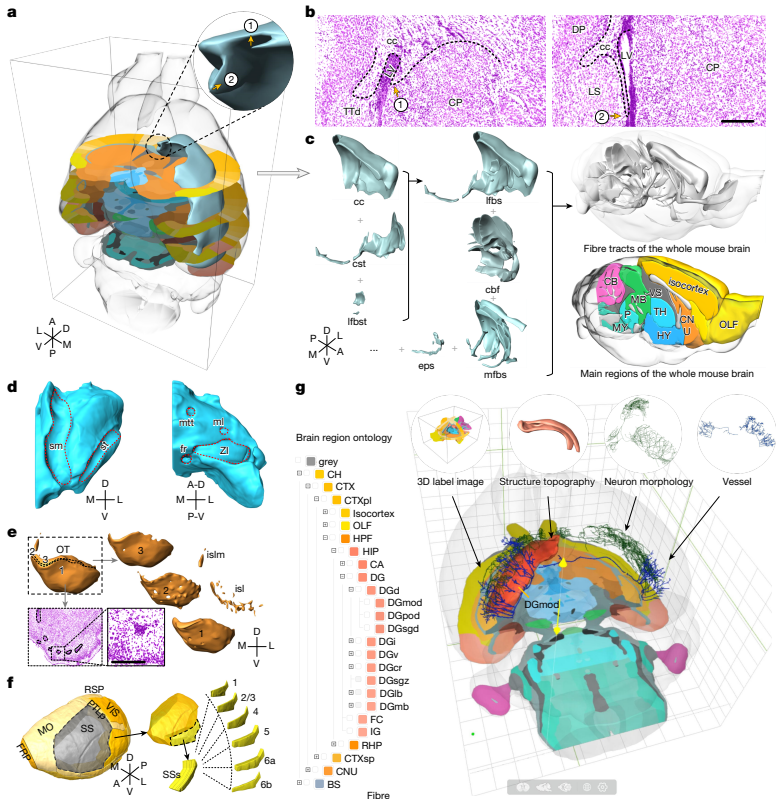
选取前连合(AC)、齿状回颗粒层(DGsg)等8个解剖结构的18个几何特征点作为颅内基准标记,通过fMOST技术获取含颅骨的全头数据集确定颅骨基准标记(前囟、人字点),建立颅内外空间对应关系。
区别于传统图谱仅提供三个标准平面,STAM可生成任意切割角度的2D图谱平面。如特定角度的切片可完整显示内侧缰核贯穿纤维束形态,偏转5.6°即导致多个皮质区域从斜切面消失。

整合1,644个单神经元形态数据,分析CA3spi(海马CA3区锥体层中间域)与CA3sod(海马CA3区始层背侧域)神经元投射差异:前者主要投射至同侧和对侧海马,后者则更多投射至内侧隔核等深部结构。
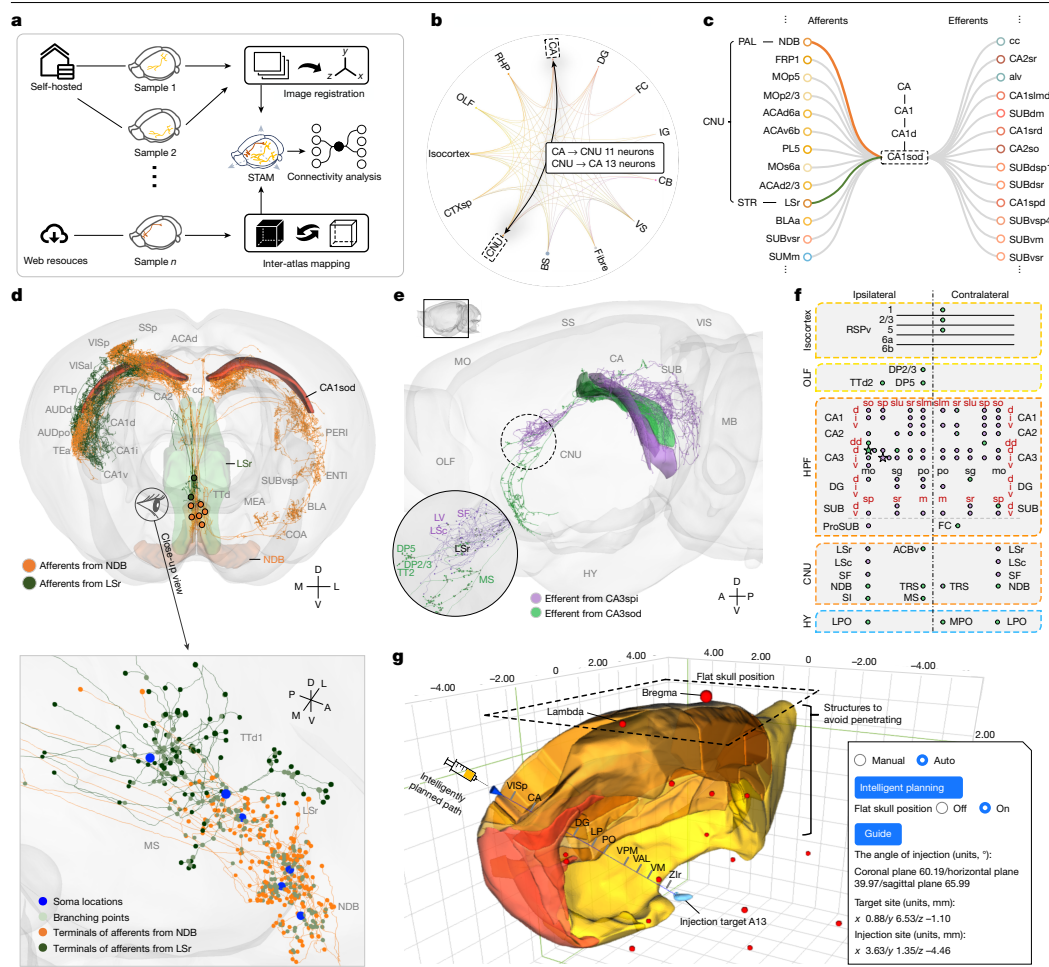
这项研究构建的STAM图谱具有三大突破性意义:首先,1-μm各向同性分辨率首次实现单细胞水平全脑空间定位;其次,多模态数据整合与任意角度切片功能克服了传统图谱的视角局限;最后,开发的在线平台(https://atlas.brainsmatics.cn/STAM/)支持脑片配准、手术规划等实际应用。研究不仅为脑科学大数据分析提供空间框架,未来还可整合阿尔茨海默病淀粉样斑块等病理特征,推动神经退行性疾病研究。正如作者所言,这项历时十年的工作"标志着脑图谱百年演进史上的新里程碑"。