
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于多组学整合分析揭示IRF1/GBP5/PARP9作为炎症性肠病诊断标志物与治疗靶点的研究
【字体: 大 中 小 】 时间:2025年07月05日 来源:Journal of Translational Medicine 6.1
编辑推荐:
本研究针对炎症性肠病(IBD)复杂发病机制中关键分子标志物缺乏的临床挑战,通过整合生物信息学、机器学习与孟德尔随机化(SMR)等多维技术,首次系统鉴定了IRF1、GBP5和PARP9作为IBD的潜在诊断标志物,并证实IRF1基因变异与IBD风险存在因果关联。研究创新性地构建"基因-免疫细胞/转录因子"调控网络,为IBD精准诊疗提供新靶点,发表于《Journal of Translational Medicine》。
炎症性肠病(IBD)作为一种慢性复发性肠道炎症性疾病,其复杂的分子机制使得临床缓解率始终难以提升。当前诊断主要依赖侵入性的内镜检查,且约30%患者存在诊断延迟。更棘手的是,现有治疗对部分患者无效,凸显对疾病分子机制的深入理解和新型生物标志物挖掘的迫切需求。在此背景下,天津医科大学团队在《Journal of Translational Medicine》发表的研究,通过多组学整合策略揭开了IBD发病的新机制。
研究采用四步技术路线:首先从GEO数据库获取4个RNA-seq数据集(GSE75214/GSE36807/GSE179285/GSE47908)构建训练/验证队列;其次通过加权基因共表达网络分析(WGCNA)和差异表达基因(DEG)筛选获得1816个IBD相关基因;进而采用蛋白互作网络(PPI)和12种机器学习算法(Random Forest/SVM等)鉴定核心基因;最后整合GTExv8血液eQTL数据和FinnGen GWAS数据,运用孟德尔随机化(SMR)和贝叶斯共定位分析验证基因与IBD的因果关系。
A total of 1816 differential genes associated with IBD were identified
通过比较133例IBD与22例对照的转录组,发现1816个差异基因(log2FC>0.585,FDR<0.05),主要富集于炎症反应和免疫调节通路。

WGCNA identified key modules on DEGs
MElightyellow模块与IBD相关性最强(R>0.5),其1094个基因显著参与干扰素信号通路。

Based on the integration and screening of PPI network analysis and machine learning
通过PPI网络和机器学习集成(RF+NaiveBayes算法AUC=0.855),最终锁定STAT1/PARP9/IRF1/GBP5/OAS2/TRIM22六个核心基因。

IRF1, GBP5, and PARP9 are diagnostic biomarkers for IBD
ROC曲线显示IRF1/GBP5/PARP9诊断效能优异(训练集AUC>0.95),三基因联合模型AUC达0.994。外部验证证实这些基因在IBD患者中显著上调(p<0.001)。

CIBERSORT evaluated IBD immune cell tags
M1巨噬细胞浸润与IRF1/GBP5呈正相关,而浆细胞呈负相关,揭示核心基因通过调节特定免疫亚群参与IBD发病。
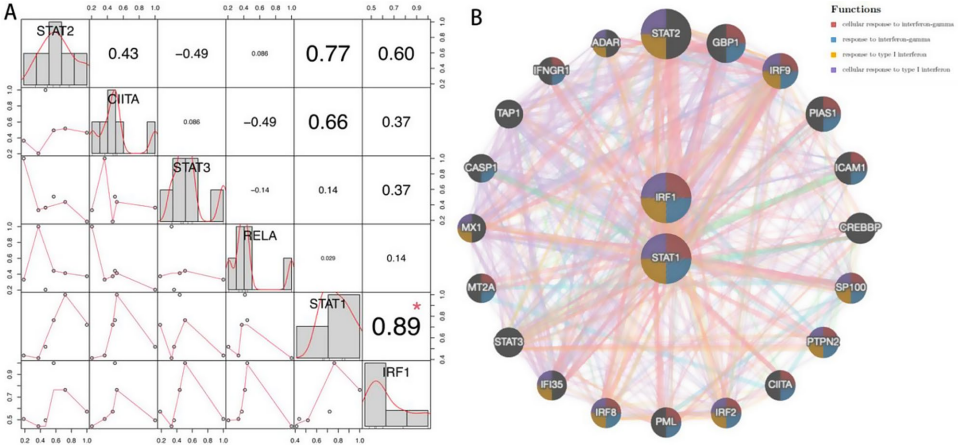
Elevated IRF1 in IBD patients has strong diagnostic value
SMR分析首次证实IRF1基因变异与IBD风险存在因果关联(b_SMR=1.5029,p=2.59E-09),HEIDI检验排除连锁不平衡干扰(p=0.351)。

该研究通过创新性的多组学整合策略,突破传统研究的三大局限:在方法学层面,将WGCNA模块分析与机器学习预测相结合,提升标志物筛选精度;在机制层面,首次通过SMR证实IRF1对IBD的遗传驱动作用;在转化医学层面,发现M1巨噬细胞-IRF1轴可作为治疗靶点。研究者预测靶向STAT3/RELA的小分子药物(如布地奈德、塞来昔布)可能通过调控IRF1网络发挥疗效,为开发新型IBD治疗方案提供理论依据。尽管存在样本来源单一等局限,但研究建立的"生物标志物-免疫调控-遗传基础"多维证据链,为IBD精准诊疗体系的构建奠定重要基础。
 生物通微信公众号
生物通微信公众号
知名企业招聘