
-

生物通官微
陪你抓住生命科技
跳动的脉搏
短读长宏基因组分类工具在废水处理微生物群落中的性能极限测试
【字体: 大 中 小 】 时间:2025年07月06日 来源:Scientific Reports 3.8
编辑推荐:
本研究针对废水处理系统中微生物群落分类的准确性难题,通过构建模拟群落(mock community),系统评估了Kaiju、Kraken2、RiboFrame和kMetaShot四种分类工具在短读长(150 bp)数据下的表现。研究发现Kaiju在属/种水平分类精度最高,但所有工具均存在显著误分类风险,尤其真核与细菌序列的交叉错误可能误导关键微生物功能研究。该成果为废水处理微生物组研究提供了方法学基准,发表于《Scientific Reports》。
工业化进程在推动社会发展的同时,也带来了严峻的水污染挑战。活性污泥(AS)和好氧颗粒污泥(AGS)作为主流生物废水处理技术,其核心在于复杂微生物群落的协同作用。然而,这些系统中微生物组成与功能的精确解析长期受限于宏基因组分类工具的准确性瓶颈——现有工具多在人类微生物组中验证,对富含稀有物种和环境特异性微生物的废水体系适用性存疑。
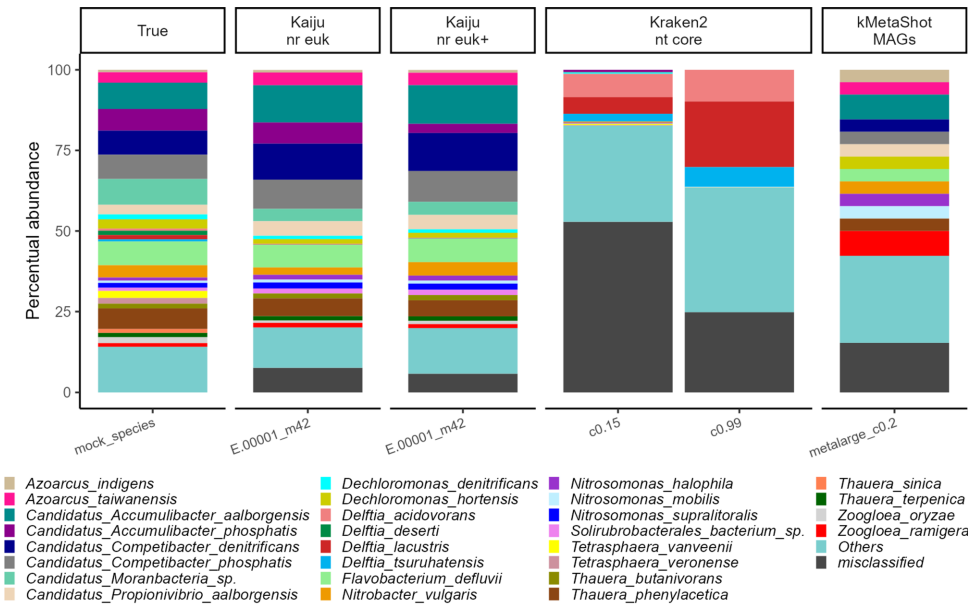
佛罗伦萨大学的研究团队在《Scientific Reports》发表了一项开创性研究,通过精心设计的模拟群落(包含16种细菌、3种真核生物和T4噬菌体),首次系统评估了短读长宏基因组分类工具在废水微生物组中的性能边界。研究发现:蛋白质水平分类工具Kaiju(nr euk+数据库)在属/种分类准确率(75%)和群落结构还原度上表现最优,但其仍存在将人类序列误判为疟原虫(Plasmodium ovale)等严重错误;而基于k-mer的Kraken2(nt core数据库)虽能检出全部目标菌属,但假阳性率高达25%。令人意外的是,专为宏基因组组装基因组(MAGs)设计的kMetaShot在MAGs层面实现零误判,但灵敏度仅30%,凸显组装质量对分类效果的决定性影响。
研究采用多维度技术路线:1)使用InSilicoSeq模拟Illumina NovaSeq 150 bp双端读长数据;2)通过BBDuk进行质量控制;3)采用MEGAHIT三种模式(default/metalarge/custom)组装contigs;4)利用MetaBat2构建MAGs;5)交叉比较四种分类工具(Kaiju、Kraken2、RiboFrame、kMetaShot)在reads/contigs/MAGs三个层面的表现;6)引入EukDetect和Bowtie2评估真核序列识别效能。
Mock processing stats
质量控制后92.6%的读长进入分析,各工具分类效率差异显著:Kaiju分类率76-94%(取决于最小覆盖阈值m),Kraken2(nt core)在置信阈值0.05时分类51%读长,而SILVA数据库下不足2%。RiboFrame内存占用最低(20 GB),但仅能分析16S rRNA片段。
Comparison at genus-level classification

Comparison at species-level classifications
Classification performances of phage T4 and lower metazoan
真核特异性工具EukDetect灵敏度不足(仅检出23条Diploscapter读长),而Kaiju(nr euk+)虽能识别线虫序列,但错误地将细菌读长归类为Steinernema(昆虫病原线虫)。T4噬菌体分类中,Kraken2(置信度0.05)检出率67%,显著优于Kaiju(38%)。
Homo sapiens reads misclassifications as bacteria and decontamination test

这项研究揭示了当前宏基因组分类工具在环境样本中的三大局限:1)数据库覆盖不全导致Candidatus菌属漏检;2)真核与原核序列交叉污染(如人类→疟原虫);3)短读长组装质量制约MAGs分类效果。作者建议:1)优先选用Kaiju(m≥30参数);2)对临床样本采用Kraken2(置信度0.05-0.3);3)必须进行人类序列去污染。该基准研究为废水处理微生物组的精准解析建立了方法学标准,尤其对资源回收(如聚羟基烷酸酯PHA生产)的菌群调控具有重要指导价值。
 生物通微信公众号
生物通微信公众号
知名企业招聘