
-

生物通官微
陪你抓住生命科技
跳动的脉搏
全球海洋双链RNA噬菌体新科"Paraxenoviridae"的发现及其生态演化意义
【字体: 大 中 小 】 时间:2025年07月06日 来源:The ISME Journal 10.8
编辑推荐:
本研究通过双链RNA(dsRNA)测序技术(FLDS)从北大平洋和东印度洋样本中重建了5个完整二分体RNA病毒基因组,结合系统发育分析将其归类为Durnavirales目(Pisuviricota门),揭示了"Paraxenoviridae"(暂定名)的衣壳蛋白(CP)结构变异与细菌宿主适应性。该发现扩展了海洋RNA噬菌体多样性认知,证实dsRNA噬菌体在全球海洋的广泛分布,为病毒-宿主互作及生态功能研究提供新视角。
海洋微生物生态系统中,RNA病毒的多样性长期被低估。传统研究受限于技术手段,对双链RNA(dsRNA)病毒尤其是感染细菌的RNA噬菌体认知严重不足。尽管近年宏转录组学揭示了包括"候选Paraxenoviricota门"在内的新病毒类群,但其基因组结构、宿主类型及进化地位仍不明确。例如,早期研究基于有限RdRP序列将Paraxenoviricota列为独立门类,而后续分析却质疑其分类位置。这些争议凸显了获取完整病毒基因组的重要性——只有通过全基因组解析,才能准确推断病毒复制策略、宿主范围及生态功能。
日本海洋研究开发机构(JAMSTEC)团队利用自主开发的FLDS技术(片段化连接双链RNA测序),对北大平洋和东印度洋表层水体微生物群落的dsRNA病毒组进行深度测序。通过CLC Genomics Workbench组装和保守末端序列聚类,重构了5个二分体RNA病毒完整基因组。研究结合RdRP系统发育树(IQ-TREE和FastTree)、AlphaFold2蛋白结构预测、HHpred结构域搜索及DALI结构比对,系统分析了病毒进化关系与功能蛋白特征。此外,通过全球海洋数据集(TARA Oceans、IMG/VR、LucaProt)的RdRP/CP序列挖掘,评估了该类病毒的生物地理分布。
1. 新型二分体基因组特征
从海洋样本中鉴定出5个完整二分体RNA病毒基因组(命名为GT1-GT5),每个基因组含RNA1(5.3–6.8 kb)和RNA2(3.5–6.6 kb)两个节段(图1)。RNA1均编码RNA依赖性RNA聚合酶(RdRP),RNA2编码衣壳蛋白(CP)或跨膜结构域蛋白。所有开放阅读框(ORF)上游均存在细菌核糖体结合位点(SD motif),强烈提示其以细菌为宿主。GT1-GT3的RNA1节段包含长达1.5 kb的5'非翻译区(UTR),其保守二级结构可能参与翻译调控(图2)。


2. 系统发育重定位
基于48个Paraxenovirus RdRP核心序列的系统发育分析表明,该类群属于Durnavirales目(Duplopiviricetes纲,Pisuviricota门),与未命名病毒家族f.0117_base-cysto形成姊妹群(图3)。这一结果纠正了此前将其列为独立门类(Paraxenoviricota)的观点。AlphaFold2预测的RdRP结构进一步证实其与Durnavirales成员(如Picobirnavirus)的相似性,但存在独特插入:HTH基序中60个氨基酸延伸环,可能参与dsRNA出口通道调控(图4)。


3. 衣壳蛋白结构创新
GT2和GT4的CP结构模型显示:壳区(S域)与Picobirnavirus CP同源,但投影区(P域)显著延伸(图5A)。GT2的CP含1,376个氨基酸,P域长达7 nm(Picobirnavirus的3倍),形成复杂的拓扑结构(图5B)。尽管序列差异大,DALI比对确认其S域与兔源Picobirnavirus CP(PDB:2VF1)结构相似(Z=2.4)。二聚体界面保守性提示两者采用类似的衣壳组装机制(图5C)。
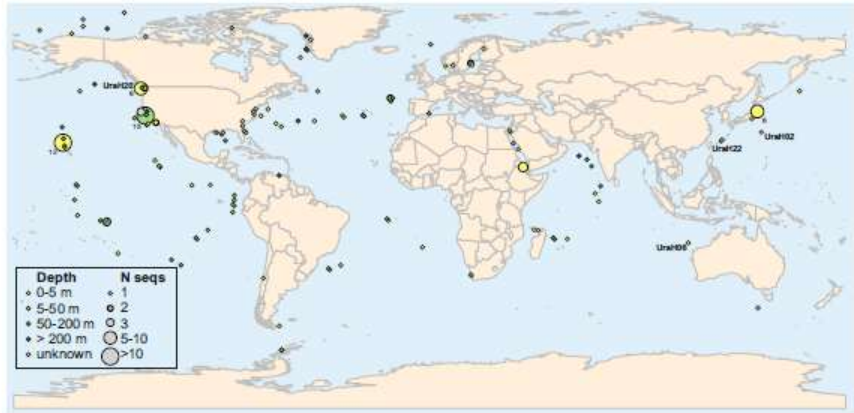
4. 全球海洋分布
该类病毒在4个海洋RNA病毒组中占比2–18%,与Picobirnaviridae、Totiviridae等已知dsRNA病毒共存(图6A)。全球数据集(658个RdRP/800个CP序列)证实其广泛分布于全球表层至中层海水,在极地至热带海域均有检出(图6B),表明其对海洋微生物生态的潜在影响。

本研究通过FLDS技术克服了分段RNA病毒基因组组装的挑战,首次揭示了海洋dsRNA噬菌体新科"Paraxenoviridae"的完整基因组特征。关键结论包括:
该研究不仅将dsRNA噬菌体的已知多样性扩展至新科水平,也为探索病毒-宿主共进化及海洋病毒生态功能提供了全新模型。相关成果发表于《The ISME Journal》,标志着海洋RNA病毒研究从片段化序列向完整功能基因组解析的重要跨越。
 生物通微信公众号
生物通微信公众号
知名企业招聘