
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于模拟小规模杂交奶牛群体的等位基因品种来源精准鉴定算法开发与应用
【字体: 大 中 小 】 时间:2025年07月13日 来源:Genetics Selection Evolution 3.6
编辑推荐:
针对低收入和中等收入国家(LMICs)奶牛杂交育种中缺乏系谱记录的问题,Berihu Welderufael等研究人员开发了一种不依赖系谱的等位基因品种来源(BOA)鉴定算法。通过模拟五轮杂交选育程序,采用多核心长度单倍型比对方法,实现了平均95.76%的正确分配率,核心分配准确度达0.99。该研究为LMICs开展基因组选择提供了重要技术支撑。
在低收入和中等收入国家(LMICs),小规模农户的奶牛养殖面临着生产力低下与适应性难以兼顾的困境。通过将高产的外来(exotic)品种与耐逆性强的本地(local)品种杂交,虽能短期提升产奶量,但长期缺乏系统的育种规划,导致种群出现无序混合。更棘手的是,这些地区普遍缺乏完整的系谱记录,使得传统育种方法难以实施。非洲奶牛遗传增益(ADGG)等项目试图通过引入优质遗传物质改善现状,却因环境差异和育种基础设施不足而收效有限。在此背景下,精准鉴定杂交个体中等位基因的品种来源(Breed Origin of Alleles, BOA)成为突破育种瓶颈的关键——这不仅能评估外来种质的有效性,更能为基因组选择提供重要依据。
爱丁堡大学罗斯林研究所(The Roslin Institute, University of Edinburgh)的Berihu Welderufael等研究人员在《Genetics Selection Evolution》发表研究,开发了一种不依赖系谱的BOA鉴定算法。研究团队采用AlphaSimR软件模拟了包含2500头牛的基因组数据,设定1条含1000个SNP的常染色体,通过马尔可夫聚结模拟器(MaCS)生成古代牛品种分化为外来和本地品种的遗传变异。随后模拟了5轮杂交选育过程:每轮选择1000头本地母牛与25头外来公牛配种,产生2000头杂交后代。利用AlphaPhase软件对基因型数据进行单倍型分型,设置10种核心长度(100-280个SNP)和偏移/非偏移两种模式,共构建200种分析场景。通过比较杂交个体单倍型与纯种单倍型库的最佳匹配度,实现等位基因品种来源的精准鉴定。
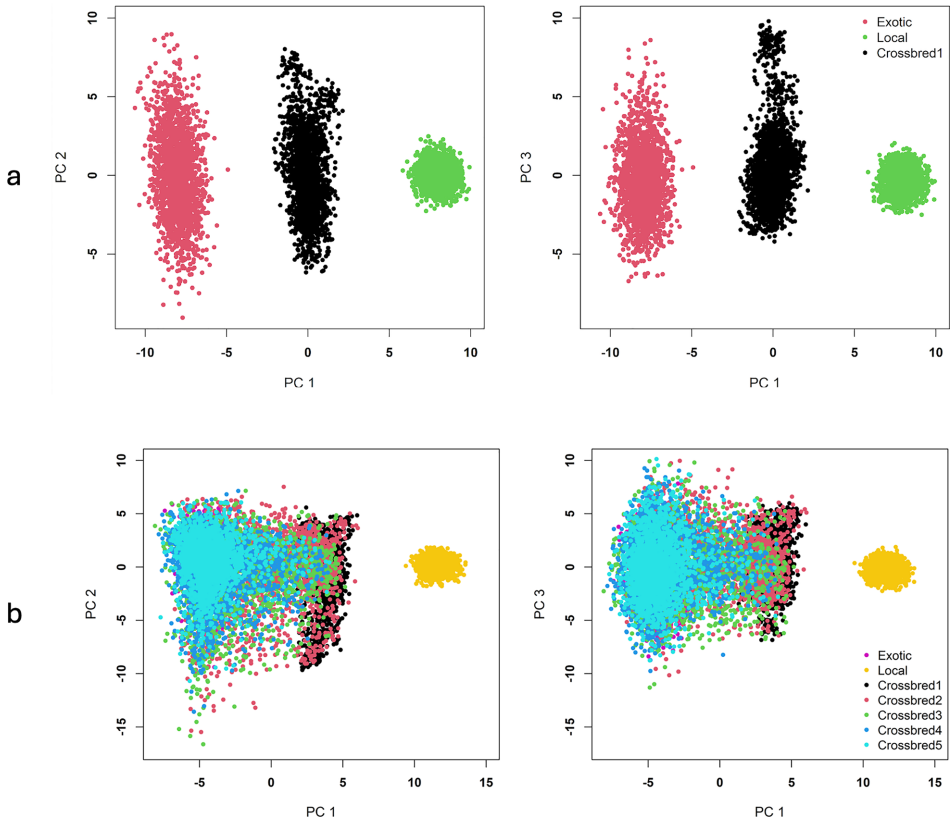
主成分分析(PCA)显示第一代杂交牛位于外来与本地品种之间,随着杂交代次增加,逐渐向外来品种聚类。群体分化指数FST为0.09,表明基础群体存在适度遗传分化。
核心长度为200个SNP时分配效率最优,五代杂交牛的平均分配成功率达96%。其中第一代杂交牛有29/1000个SNP未能分配,到第五代降至23/1000。

核心分配模式下平均正确率95.76%(错误率1.39%),共识分配模式下正确率93.21%(错误率仅0.46%)。阈值设定显示:匹配阈值50-60%时,分配效率与准确度达到最佳平衡。
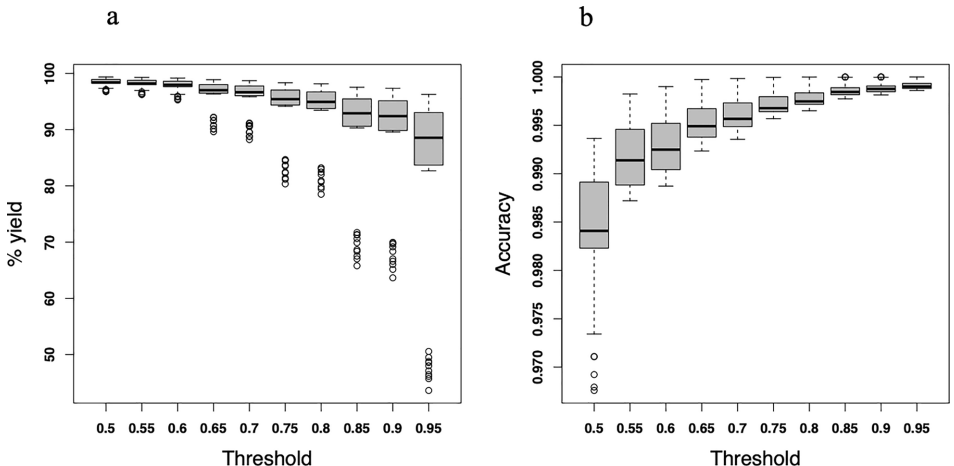
该研究建立的算法在模拟环境中展现出卓越性能,核心分配准确度0.99、共识分配准确度达1.00,显著优于既往报道的STRUCTURE(96%)和GENECLASS(85%)等方法。这种方法的价值在于:首先,突破了LMICs缺乏系谱记录的制约,仅需基因组数据即可追溯等位基因来源;其次,多核心长度与共识策略的应用,有效降低了单倍型比对错误的影响;更重要的是,为开展品种特异性效应估计提供了技术基础,这将显著提升基因组选择的准确性。研究者特别指出,虽然算法在模拟数据中表现优异,但在实际应用中仍需考虑非洲牛群复杂的杂交历史和多品种混合特性。未来研究可进一步整合基因组历史信息,以更全面地反映种群遗传背景。这项成果为LMICs实施可持续的奶牛遗传改良提供了重要工具,对实现联合国可持续发展目标中的"零饥饿"具有积极意义。
 生物通微信公众号
生物通微信公众号
知名企业招聘