
-

生物通官微
陪你抓住生命科技
跳动的脉搏
CATH-ddG:基于CATH同源超家族的蛋白质-蛋白质相互作用突变效应稳健预测新方法
【字体: 大 中 小 】 时间:2025年07月16日 来源:Bioinformatics 4.4
编辑推荐:
本研究针对蛋白质-蛋白质相互作用(PPI)突变效应预测中存在的泛化能力差异和数据泄露问题,开发了CATH-ddG框架。通过整合CATH同源超家族分类、几何深度学习编码器和FoldX能量约束,实现了对ΔΔG变化的精准预测。在HER2抗体和SARS-CoV-2 RBD-ACE2结合优化案例中验证了其优越性能,为药物设计和疾病机制研究提供了新工具。
在生命科学领域,蛋白质相互作用网络的扰动与疾病发生发展密切相关。然而,传统实验方法如表面等离子共振(SPR)耗时费力,而现有计算模型在预测突变对结合自由能(ΔΔG)影响时,面临"简单突变"与"困难突变"预测性能差异大、数据泄露严重等挑战。特别是当测试样本与训练样本的TM-score<0.6时,现有深度学习模型的预测准确性显著下降。
针对这一难题,中南大学计算机科学与工程学院的研究团队开发了CATH-ddG预测系统。该系统创新性地引入CATH同源超家族分类标准构建防泄露数据集,通过几何编码器捕捉突变微环境特征,并整合FoldX能量项作为物理约束。相关成果发表在生物信息学顶级期刊《Bioinformatics》上。
研究采用了三项关键技术:1)基于CATH v4.4数据库构建非泄露训练/测试集,确保序列相似度<30%;2)开发空间-序列几何编码器,采用256残基的k近邻图(k1=20,k2=7,k3=2)表征突变位点微环境;3)设计混合噪声策略(主干原子δ=0.15?,侧链δ=0.25?)进行数据增强。
【主要结果】
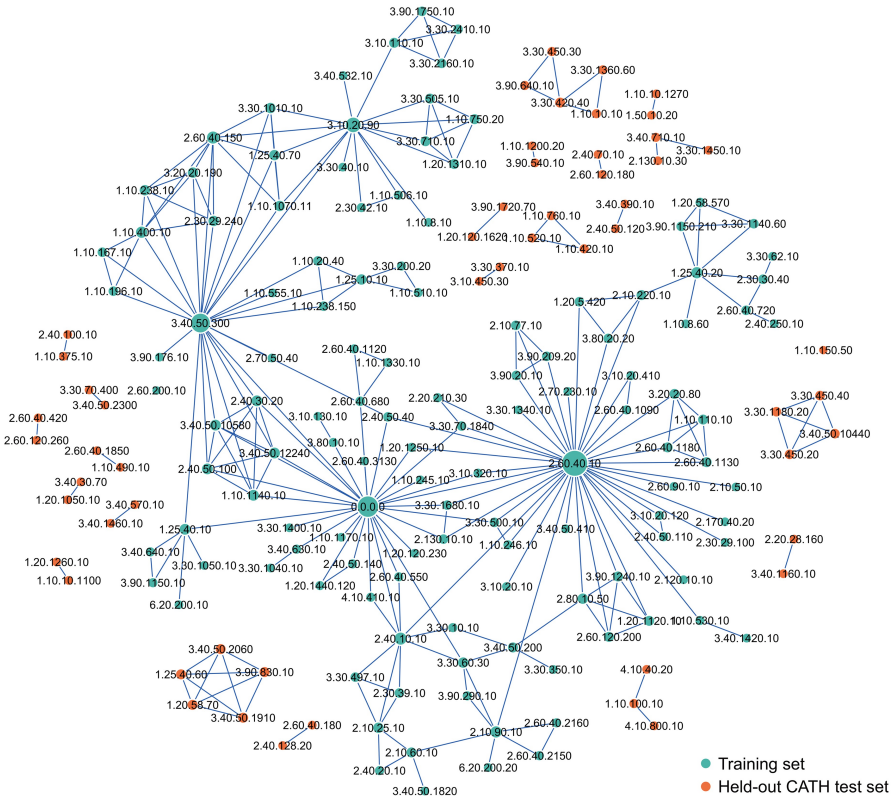
数据拆分验证:通过CATH超家族划分的测试集包含54个超家族、813个突变,显著降低了蛋白质水平和复合物水平的数据泄露。如图1所示,免疫球蛋白样超家族2.60.40.10是SKEMPI v2.0中最连接的超家族。
模型架构创新:如图2所示,CATH-ddG采用三重任务框架:(1)CATH超家族多标签分类的自我监督学习;(2)基于ProteinMPNN优化的空间-序列几何编码器;(3)FoldX能量项指导的轻量级预测模块。

性能验证:在HER2抗体测试集上,Pearson相关系数达0.607,比DiffAffinity提高2.88%;在SARS-CoV-2 RBD-ACE2测试集上,PearsonR为0.579,显著优于FoldX(0.385)。如图3所示,模型对多突变引起的构象变化(RMSD>1.5?)具有更好适应性。

消融实验:如图4所示,移除ESM2适配器使PearsonR下降15.58%,证实共进化信息的关键作用;而移除FoldX能量项导致SpearmanR降低11.15%,说明物理约束对突变效应排序的重要性。

这项研究通过整合多源生物信息,建立了蛋白质相互作用突变效应预测的新范式。其创新性体现在:1)首次引入CATH超家族分类解决数据泄露问题;2)开发混合噪声策略增强模型鲁棒性;3)实现ESM2语言模型与几何特征的有机融合。在419个HER2抗体变体和285个SARS-CoV-2 RBD突变的验证表明,该方法可为抗体药物优化和病毒进化研究提供可靠的计算工具。未来通过结合AlphaFold3等结构预测技术,有望进一步拓展其在蛋白质设计中的应用前景。
 生物通微信公众号
生物通微信公众号
知名企业招聘