
-

生物通官微
陪你抓住生命科技
跳动的脉搏
NEAR:基于神经嵌入的氨基酸关系模型显著提升蛋白质同源搜索效率与准确性
【字体: 大 中 小 】 时间:2025年07月16日 来源:Bioinformatics 4.4
编辑推荐:
本研究针对蛋白质语言模型(PLMs)在同源搜索中存在的高误报率和计算速度慢的问题,开发了NEAR(Neural Embeddings for Amino acid Relationships)方法。通过对比学习训练的ResNet模型生成残基级嵌入向量,结合k-NN搜索和邻居聚合策略,在保持pHMMs(profile Hidden Markov Models)高灵敏度的同时,将搜索速度提升5倍以上,误报率显著低于ProtTransT5等主流PLMs。该成果为大规模基因组注释提供了高效预过滤方案,代码已开源。
蛋白质同源搜索的困境与突破
随着DNA测序成本的降低,海量蛋白质序列数据对计算注释工具提出了严峻挑战。传统基于序列比对的同源搜索方法(如BLAST)虽快速但灵敏度有限,而高灵敏度的profile HMMs(pHMMs)又面临计算复杂度高的问题。近年来兴起的蛋白质语言模型(PLMs)虽能识别部分传统方法遗漏的同源关系,却普遍存在两大缺陷:计算速度比经典工具慢1-2个数量级,且误报率高达30-50%。美国蒙大拿大学(University of Montana)与亚利桑那大学(University of Arizona)的研究团队在《Bioinformatics》发表的研究,通过创新性的神经嵌入方法NEAR,实现了速度与精度的双重突破。
关键技术方法
研究团队采用1D残差卷积网络(ResNet)架构构建嵌入模型,利用HMMER3生成的600万对可信序列对齐数据指导对比学习。通过FAISS库实现高速k近邻搜索(k=150),结合tantan重复序列屏蔽技术降低噪声。评估使用Uniclust30数据集构建的10,000个查询序列和40,000个目标序列(含随机洗牌阴性对照),以phmmer E值≤10-10作为金标准。
研究结果
3.1 非同源诱饵序列过滤
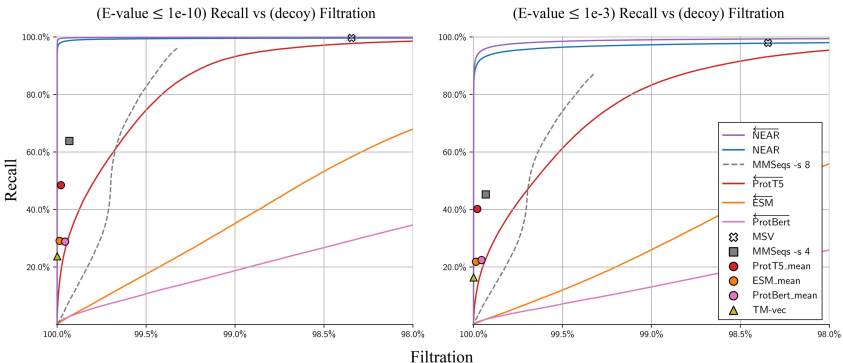
3.2 低相似度序列过滤

计算效率
表3显示,NEAR处理40,000序列的端到端耗时仅264秒(含12秒嵌入+26秒索引),参数规模(7M)仅为ProtTransT5(3B)的0.23%。反向搜索设计使GPU内存占用减少60%,特别适合大规模数据库应用。
结论与展望
NEAR通过"数据扩展"策略——将氨基酸序列转化为256维残基嵌入向量,开创性地平衡了搜索精度(N-Pair损失函数)与速度(FAISS近似搜索)。其创新性体现在:1)首次实现残基级可解释嵌入,每个向量对应局部对齐可能性;2)通过δ/√d噪声门控机制消除高维向量随机相似性;3)开源实现为pHMM工具(如nail)提供高效预过滤方案。未来研究可探索嵌入向量直接指导多序列比对(MSA),或开发稀疏表示方法以支持万亿级序列搜索。这项突破标志着蛋白质同源搜索从"序列比对时代"迈向"神经嵌入时代"的关键一步。
 生物通微信公众号
生物通微信公众号
知名企业招聘