
-

生物通官微
陪你抓住生命科技
跳动的脉搏
Beaver:基于短读长单细胞RNA测序数据的细胞特异性转录本组装新方法
【字体: 大 中 小 】 时间:2025年07月16日 来源:Bioinformatics 4.4
编辑推荐:
为解决单细胞RNA测序(scRNA-seq)中全长度转录本重建的难题,宾夕法尼亚州立大学团队开发了Beaver算法。该研究通过构建转录片段图整合跨细胞信息,结合动态规划与随机森林模型,实现了在保持细胞特异性表达模式的同时显著提升组装精度。实验表明,Beaver比现有工具精度提高9.8%-67.0%,为单细胞异构体分析提供了新工具。
单细胞RNA测序(scRNA-seq)技术的突破性进展,让科学家们得以窥见细胞间的异质性奥秘。然而在这项技术的光环背后,隐藏着一个长期悬而未决的核心难题——如何在单个细胞中准确重建全长度转录本?现有方法如同盲人摸象:单样本组装器因覆盖率不足产生碎片化结果,而元组装方法又难以兼顾共识构建与细胞特异性表达特征的保留。这种技术瓶颈严重制约了单细胞水平上异构体动态变化的研究。
针对这一挑战,宾夕法尼亚州立大学(The Pennsylvania State University)计算机科学与工程系的Qian Shi、Qimin Zhang和Mingfu Shao*团队在《Bioinformatics》发表了创新性解决方案。研究者开发的Beaver算法,通过巧妙整合跨细胞信息与机器学习预测,实现了单细胞分辨率下高精度转录组组装。实验数据证实,该工具在真实和模拟的Smart-seq3数据集上,比现有最佳工具的精度提升幅度达9.8%-67.0%,为单细胞转录组学研究提供了突破性技术手段。
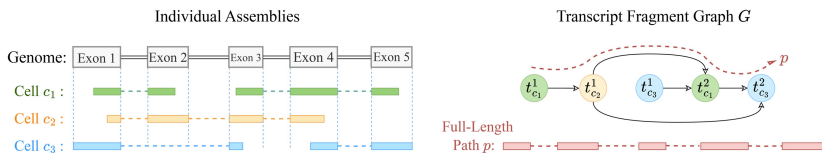
研究团队采用四步法技术路线:首先收集Aletsch生成的个体组装结果;构建转录片段图组织跨细胞片段;设计基于瓶颈连接评分的动态规划算法搜索候选全长转录本;最终通过两阶段随机森林模型(含51个工程化特征)进行细胞特异性评分。其中创新的转录片段图结构,通过顶点表示转录片段、边表示片段兼容性,有效解决了信息整合与特异性保留的矛盾。
转录片段图构建
研究构建的有向图G=(V,E)中,顶点代表来自不同细胞的转录片段,边连接满足后缀内含子链与前缀内含子链精确匹配的片段。如图1所示,该数据结构能有效组织来自192个HEK293T细胞的片段信息,路径搜索算法通过优化合并评分(瓶颈连接评分与连接数乘积),在保证连接可靠性的同时促进片段延伸。
机器学习评分系统
Beaver-General模型通过30个全局特征(如瓶颈连接评分、连接支持细胞数等)筛选候选转录本,Beaver-Specific模型则追加21个细胞特异性特征(如细胞特异的连接覆盖率)进行精细评分。在染色体1-9训练的数据显示,该双模型策略能有效区分真实表达与人工嵌合体。
性能验证
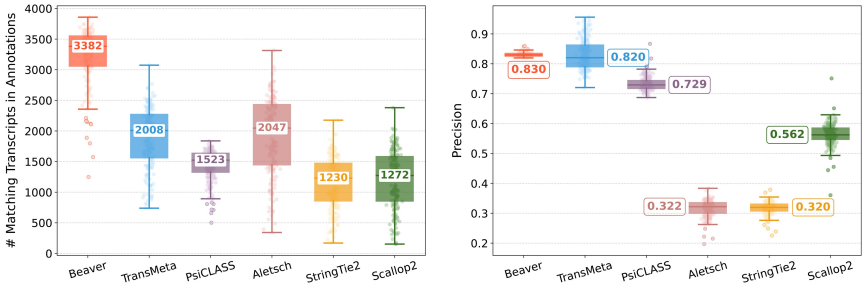
在HEK293T真实数据测试中,Beaver匹配转录本数较次优方法TransMeta提高68.4%,精度达83%(图2)。模拟数据集验证更显示其细胞特异性匹配优势:在Fibroblast-Sim数据中,Beaver较单样本组装器Scallop2提升36.3%精度(图7),显著缩小"通用匹配"与"细胞特异性匹配"的差距(图9)。这种优势源于其独特的片段连接策略与细胞特异性评分体系,避免了TransMeta等工具因简单连接阈值导致的特异性丢失问题。
这项研究标志着单细胞转录组分析的重要突破。Beaver的创新性体现在三个方面:首先,转录片段图的数据结构突破了传统元组装方法的局限,实现了跨细胞信息利用与细胞特异性保留的平衡;其次,动态规划算法通过优化合并评分,有效解决了覆盖率波动导致的组装碎片化问题;最后,两阶段随机森林模型建立的51维特征体系,为单细胞水平的转录本验证设立了新标准。这些技术进展将推动单细胞异构体发现、稀有细胞类型鉴定等研究领域的发展。
尽管当前训练依赖于参考注释,但模拟数据验证表明,当获得真实单细胞表达谱时,该框架能实现更精准的建模。未来整合长读长数据指导转录本选择,可能进一步减少错误连接。作为开源工具,Beaver的推广应用将助力实现单细胞转录组分析从基因水平到异构体水平的跨越,为精准医学研究提供新的技术支撑。
 生物通微信公众号
生物通微信公众号
知名企业招聘