
-

生物通官微
陪你抓住生命科技
跳动的脉搏
MetaGeno:基于染色体多任务基因组框架的缺血性卒中风险预测新方法
【字体: 大 中 小 】 时间:2025年07月19日 来源:Briefings in Bioinformatics 6.8
编辑推荐:
本研究针对传统多基因风险评分(PRS)在缺血性卒中(IS)风险预测中存在的线性模型局限性和非线性互作捕捉不足等问题,开发了MetaGeno框架。该研究通过染色体特异性嵌入层捕捉局部和全局遗传变异互作,结合Transformer模型分析IS及5种相关疾病(AF/HT/CAD/T2D/HCL)的共享遗传信息,在UK Biobank数据集上实现AUROC 0.809的预测性能,风险分层显示最高1%人群卒中风险增加2.14倍。研究为精准预防提供了新型临床决策支持工具。
在脑血管疾病研究领域,缺血性卒中(Ischemic Stroke, IS)的遗传风险评估长期面临重大挑战。尽管全基因组关联研究(GWAS)已鉴定出数十个IS相关基因位点,但当前主流的线性多基因风险评分(Polygenic Risk Score, PRS)方法存在明显局限:既无法捕捉单核苷酸多态性(SNP)间的非线性互作,也难以整合多种可调控风险因素(Modifiable Risk Factors, MRF)的协同效应。更棘手的是,约37.9%的IS遗传力仍无法通过现有模型解释,导致临床上大量具有遗传易感性的个体被传统风险评估工具低估。
针对这一现状,澳大利亚悉尼科技大学人工智能研究所的研究团队在《Briefings in Bioinformatics》发表了创新性研究成果。他们开发的MetaGeno框架通过三大突破性设计改进了IS风险预测:首先,首创染色体特异性嵌入层,依据SNP的染色体位置进行分组建模,符合连锁不平衡(Linkage Disequilibrium, LD)的生物学原理;其次,采用多任务学习策略同步分析IS及其五种关键MRF(心房颤动AF、高血压HT、冠状动脉疾病CAD、2型糖尿病T2D和高胆固醇血症HCL)的遗传关联;最后,系统比较了五种深度学习架构,证实Transformer模型在捕捉长程基因组互作方面具有独特优势。
研究团队运用了多项关键技术:从UK Biobank和All of Us项目中获取11,584例IS患者和460,985例对照的基因组数据;设计染色体分组嵌入矩阵处理3,025个GWAS筛选的SNP;采用五折交叉验证评估Transformer/LSTM/GRU等模型的预测性能;通过风险分层分析计算危险比(Hazard Ratio, HR)和累积发病率。特别值得注意的是,该方法仅需3,025个SNP就实现了优于传统百万级SNP模型的预测精度。
研究结果部分呈现了系列重要发现:
模型选择方面,Transformer以平均AUROC 0.792显著优于其他架构,对IS的预测性能达0.809。如图2所示,染色体嵌入策略(AUROC 0.809)较全局嵌入(0.791)和独热编码(0.776)展现出明显优势。
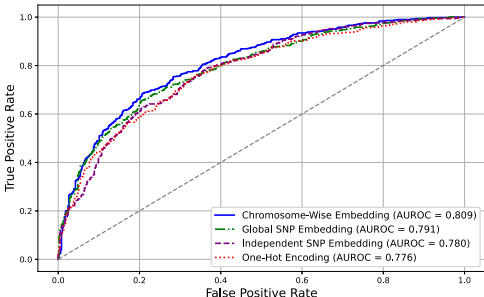
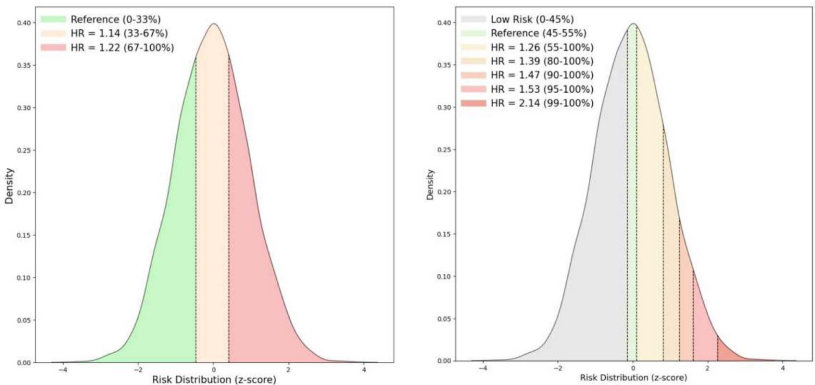
与传统方法对比中,MetaGeno(AUROC 0.809)全面超越PRS(0.650)、LDpred2(0.690)和DeepRisk(0.792)等基线模型。风险分层显示,最高1%人群的IS风险增加2.14倍(95%CI:1.81-2.46),如图3所示。
多疾病组合分析揭示,整合全部MRF时C-index达0.698,其中HT(0.661)和AF(0.646)贡献最显著。如图4所示,染色体1/4/6/7/9/12通过PHACTR1/ZFHX3等基因位点对预测贡献突出。

讨论部分强调了该研究的双重价值:方法学上,染色体嵌入层首次实现了基因组局部结构与全局互作的双重建模,将SNP需求从百万级降至数千级;临床上,如图5所示,同时携带HT/AF和卒中家族史的高遗传风险人群,80岁时累积发病率达4.9%,是低风险组的近5倍,为精准预防提供了量化依据。

该研究的局限性在于尚未整合影像组学等表型数据,且跨种族验证显示AUROC从UK Biobank的0.809降至All of Us的0.764,提示需进一步优化种群适应性。未来通过纳入多组学数据和扩大样本多样性,MetaGeno框架有望成为卒中精准预防的核心技术工具。
 生物通微信公众号
生物通微信公众号
知名企业招聘