
-

生物通官微
陪你抓住生命科技
跳动的脉搏
粪便免疫化学检测管样本在不同储存条件下微生物组稳定性研究及其在大规模人群研究中的应用价值
【字体: 大 中 小 】 时间:2025年07月25日 来源:Scientific Reports 3.8
编辑推荐:
本研究针对粪便免疫化学检测(FIT)管样本在结直肠癌筛查中残留物的微生物组稳定性问题,通过系统评估不同采样部位(表面vs核心)、短期(+20°C)和长期(-18°C/-80°C)储存条件及运输介质(缓冲液vs水)对全长16S rRNA基因测序结果的影响,证实FIT管样本微生物组在多种预处理条件下保持稳定特征。研究采用牛津纳米孔技术(ONT)完成V1-V9区测序,发现尽管存在肠球菌属(Enterococcus faecalis)等胶原酶产生菌的比例变化(0.2-2.6%),个体微生物组的丰富度(richness)、香农多样性(Shannon diversity)和整体特征仍高度稳定。该成果为利用现有结直肠癌筛查资源开展大规模微生物组研究提供了方法学基础。
在人类健康研究领域,肠道微生物组已成为解读疾病发生机制的关键"第二基因组"。随着测序技术的进步,全长16S rRNA基因分析能够提供比传统V3-V4区测序更精确的菌种鉴别能力。然而,大规模人群研究的样本采集标准化始终是制约微生物组研究可重复性的瓶颈——常规粪便采集需要严格冷链运输和复杂预处理,而临床筛查中广泛使用的粪便免疫化学检测(FIT)管因其简便性(仅需10mg粪便样本)可能成为理想的替代方案。但关键问题在于:这些非专门设计的采样管在现实场景中经历不可控的邮寄时间和温度波动后,其微生物组特征能否保持真实可靠?
来自丹麦哥本哈根大学医院系统(Department of Surgery, Copenhagen University Hospitals-Holbaek; Department of Clinical Biochemistry, Copenhagen University Hospital-Bispebjerg and Frederiksberg)的研究团队针对这一挑战展开攻关。他们创新性地利用结直肠癌筛查中OC-Sensor采样管的残留缓冲液,系统评估了全长16S rRNA测序在不同预处理条件下的稳定性表现。这项发表在《Scientific Reports》的研究为利用现有筛查资源开展微生物组流行病学研究扫清了方法学障碍。
研究团队采用三项核心技术路线:首先设计多因素对照实验,包括采样部位(粪便表面/核心)、短期室温储存(0-10天)、长期冷冻储存(-18°C/-80°C,1-6个月)和缓冲介质(HEPES缓冲液/无菌水)比较;其次使用AllPrep PowerFecal Pro DNA/RNA Kit进行DNA提取,通过Qubit荧光定量确保质量;最后采用牛津纳米孔技术(ONT)的SQK-RAB204试剂盒完成全长16S rRNA基因(27F-1492R引物,约1.5kb)扩增与测序,数据经EPI2ME平台进行生物信息学分析。
采样部位差异分析
通过比较8名健康志愿者粪便表面与核心样本,发现物种丰富度(richness)和α多样性无显著差异。主坐标分析(PCoA)显示个体间差异远大于采样部位引起的变异,证实OC-Sensor采样管无需严格 homogenization 即可获得代表性样本。
短期储存稳定性
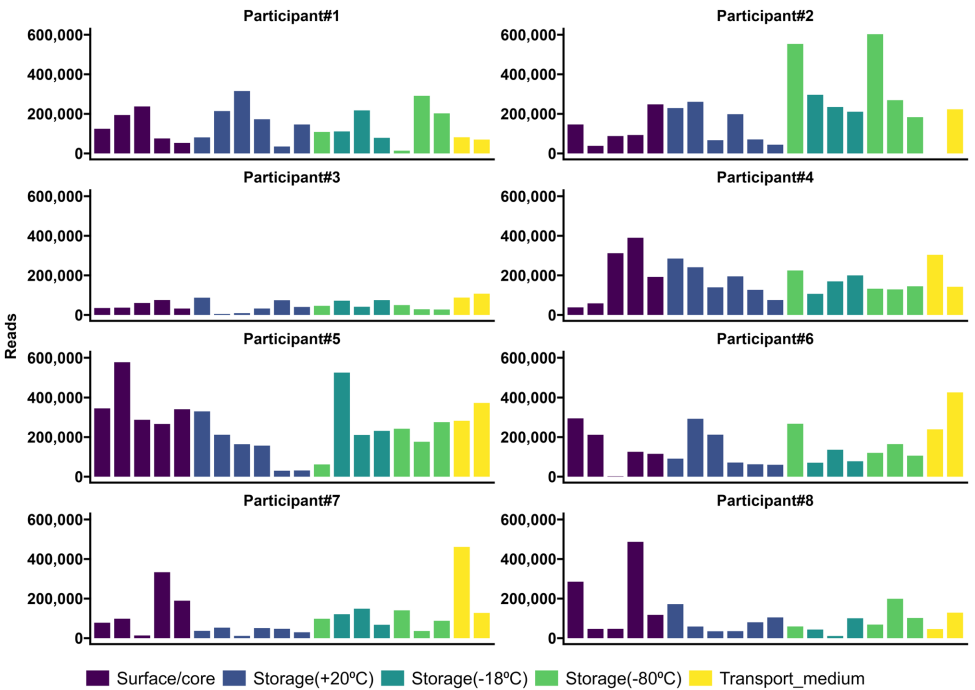
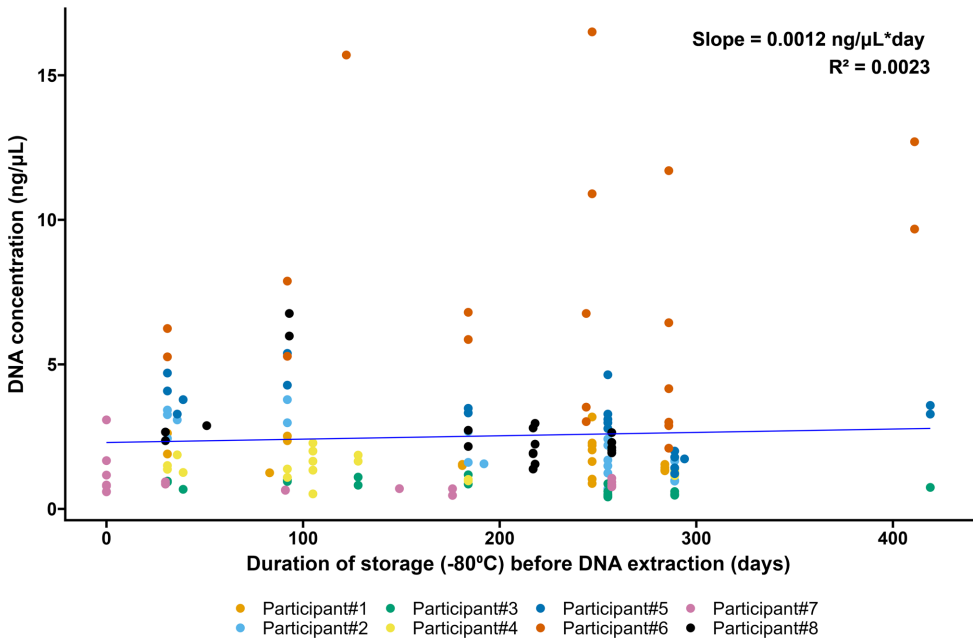
长期储存可靠性
缓冲介质影响
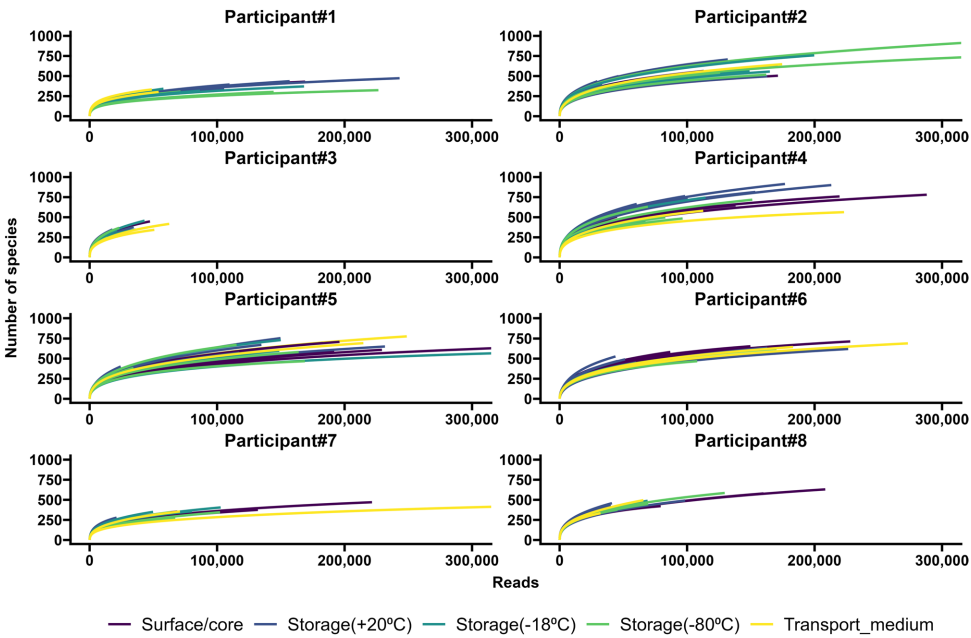
HEPES缓冲液显著抑制粪肠球菌生长(图6b),但无菌水组与缓冲液组的微生物组整体结构无统计学差异。稀有曲线分析(图3)显示两组测序深度相当,说明缓冲介质不影响DNA提取效率。
这项研究通过严谨的多维度验证,确立了FIT管样本在微生物组研究中的"三重稳定性"原则:采样部位灵活性、储存条件宽容性和介质兼容性。特别在结直肠癌筛查场景中,样本通常经历3-5天室温邮寄,本研究证实这种现实条件下的样本仍能保持核心微生物特征。研究同时揭示了特殊菌群(如胶原酶产生菌)可能成为术后并发症的生物标志物,为后续转化研究指明方向。
方法论上,全长16S rRNA测序相比传统V3-V4区分析具有物种分辨优势,但1.5kb扩增片段对DNA质量要求更高。本研究成功从低生物量(10mg粪便)样本获取高质量数据,为类似研究建立技术标杆。作者特别指出,虽然肠球菌等耐受力强的细菌可能在运输中增殖,但个体特征性的微生物组"指纹"仍能被准确识别,这种稳定性使得现有筛查项目的生物样本库成为挖掘微生物组-疾病关联的宝贵资源。
 生物通微信公众号
生物通微信公众号
知名企业招聘