
-

生物通官微
陪你抓住生命科技
跳动的脉搏
印度喜马拉雅拟南芥群体揭示深层次进化历史并可能是物种向东扩张的源头
【字体: 大 中 小 】 时间:2025年07月31日 来源:BMC Plant Biology 4.8
编辑推荐:
本研究针对拟南芥(Arabidopsis thaliana)在印度喜马拉雅地区的群体开展全基因组测序分析,解决了该物种在亚洲高海拔地区进化历史不明的关键问题。研究人员通过对38个样本的基因组数据分析,发现该群体具有比欧亚大陆群体更高的遗传多样性,并携带独特的S单倍型,证实其独立演化且未与欧亚非孑遗群体发生基因混合。研究首次揭示印度群体可能是物种向东扩张的源头,为理解拟南芥全球扩散历史提供了新视角。
拟南芥作为模式植物,其全球种群结构和进化历史一直是研究热点。然而,以往大规模基因组研究存在一个显著空白——缺乏对印度喜马拉雅地区群体的系统分析。这个位于物种分布边缘的高海拔区域,蕴藏着从亚热带到高山草甸的剧烈环境梯度,是研究物种适应性分化的天然实验室。更关键的是,现有理论认为现代拟南芥主要源自多个冰期避难所的孑遗种群,但物种如何扩散至东亚边缘仍存在争议,特别是跨越如此广阔气候带的长距离扩散机制尚不明确。
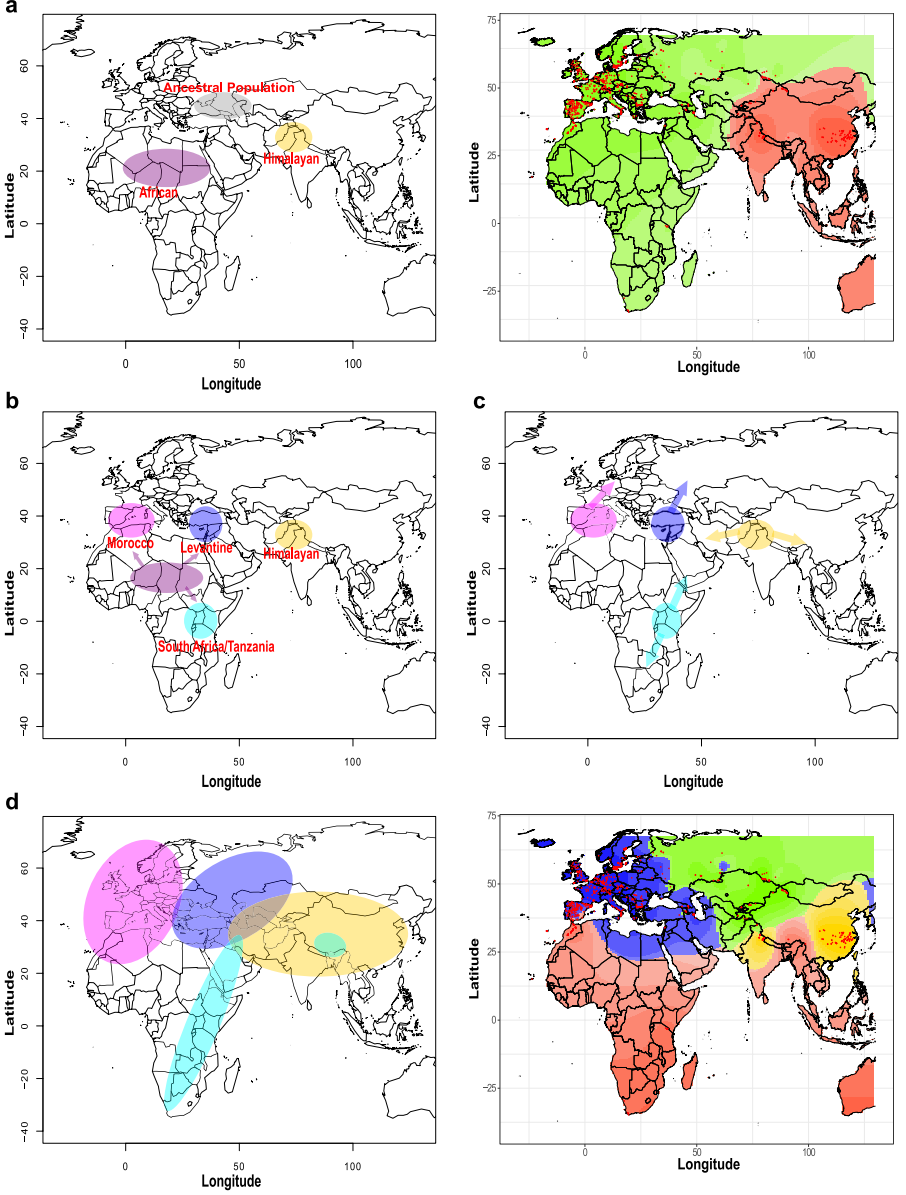
印度国家植物研究所(CSIR-National Botanical Research Institute)的研究团队通过分析印度西喜马拉雅地区8个海拔梯度(700-3400米)的38个拟南芥群体基因组,结合全球1135个基因组数据,揭示了令人惊讶的发现。该群体不仅形成了四个与海拔高度相关的独立亚群,其遗传多样性甚至超过了已知的欧亚大陆群体,与非洲祖先群体相当。更重要的是,研究首次证实印度群体携带独特的等位基因和罕见的S单倍型(如34%个体携带全球仅5.34%的C单倍型),且完全缺乏重组S单倍型——这些特征与已知的入侵谱系形成鲜明对比。
研究采用多项关键技术:Illumina HiSeqX10平台完成全基因组测序(平均深度37×);基于BWA和GATK的变异检测流程;Oxford Nanopore长读长测序解析S位点结构;MSMC和Fastsimcoal2进行群体历史重建;SweepFinder2检测选择信号;GEMMA进行环境关联分析。样本来自印度西喜马拉雅地区沿海拔梯度采集的8个自然群体。
主要研究结果:

多样性特征:印度群体核苷酸多样性(π=0.0031)高于周边中亚群体,且Tajima's D值(~1.9)显示其经历了瓶颈或平衡选择。
进化历史重建:MSMC分析表明印度群体有效种群规模在50-40万年前达到峰值,与非洲群体分化时间约10万年前。TreeMix检测到印度-长江群体与坦桑尼亚/云南/西藏群体间的基因流。
适应性特征:发现15,441个与环境相关的变异,其中5,156个在印度群体中达到固定(DAF=1),涉及光响应基因PHOT2、热激转录因子等。
结论与意义:
研究提出了修正的拟南芥扩散模型:非洲祖先分化为摩洛哥和坦桑尼亚-南非(TZSA)两支,后者通过东南走廊抵达印度并进一步向东扩散。印度群体因其高遗传多样性、独特S单倍型分布和海拔适应性,成为物种向东扩张的"跳板"。这一发现不仅填补了拟南芥在亚洲高海拔地区进化历史的空白,更挑战了"欧亚非孑遗群体主导扩张"的传统观点。
该成果发表于《BMC Plant Biology》,为理解物种在极端环境下的适应性分化提供了新案例,也为后续研究高山植物基因组进化与气候变化响应机制奠定了重要基础。
 生物通微信公众号
生物通微信公众号
知名企业招聘