
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于47个SNP位点的扩增子分型技术精准鉴别北美狼鲈属物种及其杂交种
【字体: 大 中 小 】 时间:2025年08月03日 来源:Conservation Genetics Resources 0.9
编辑推荐:
为解决北美狼鲈属(Morone)物种及其杂交种在管理保护中的鉴定难题,研究人员开发了一种基于47个SNP位点的GT-seq分型技术。该研究通过RAD-seq筛选物种特异性标记,结合PCA分析实现100%准确区分条纹鲈(M. saxatilis)、白鲈(M. chrysops)、黄鲈(M. mississippiensis)和白鲈杂交种,为种群评估提供高效低成本解决方案。
北美狼鲈属鱼类作为重要的管理和渔业资源,其物种鉴定长期依赖形态学特征,但杂交个体和幼体的鉴别存在重大挑战。随着条纹鲈(Morone saxatilis)与白鲈(M. chrysops)杂交种的大规模增殖放流,以及黄鲈(M. mississippiensis)等物种的人为扩散,传统方法已难以满足精准管理需求。尤其在水库生态系统和跨界水域中,杂交个体的基因渗入可能改变野生种群遗传结构,亟需开发高效可靠的分子鉴定工具。
针对这一科学问题,美国密歇根州立大学渔业与野生动物系(Jared J.Homola团队)联合密苏里大学合作鱼类与野生动物研究所,开发了首个覆盖北美全部四种狼鲈属物种的SNP分型技术。研究通过SbfI酶切的RAD-seq(限制性酶切位点关联DNA测序)对10条纹鲈、10白鲈及50疑似杂交个体进行初筛,基于条纹鲈参考基因组(PRJNA266827)定位候选位点。经PLINK关联分析和基因组控制校正,最终筛选出47个高特异性SNP位点构建GT-seq(Genotyping-in-Thousands by sequencing)分型体系。该成果发表于《Conservation Genetics Resources》,为北美鱼类资源管理提供了分子层面的"条形码"技术。
关键技术包括:1)基于SbfI-RAD-seq的SNP发现流程;2)GT-seq引物设计(产物长度140bp,退火温度60°C);3)Chelex法快速提取237份样本DNA;4)使用GTscore v1.3进行基因型分型;5)adegenet软件包进行PCA分析。样本涵盖已知物种(白鲈51、条纹鲈66、白鲈54、黄鲈50)和实验室验证杂交种16个。
主要研究结果
标记筛选验证
通过白鲈-条纹鲈群体全基因组关联分析,从11,418个候选SNP中筛选出96个物种特异性位点,经GT-seq验证后保留47个高区分度标记。这些SNP在reads 20-100bp区间均匀分布,确保扩增稳定性。
物种鉴别效能
PCA分析显示,前三个主成分累计解释82.3%遗传变异,清晰区分所有测试类群。
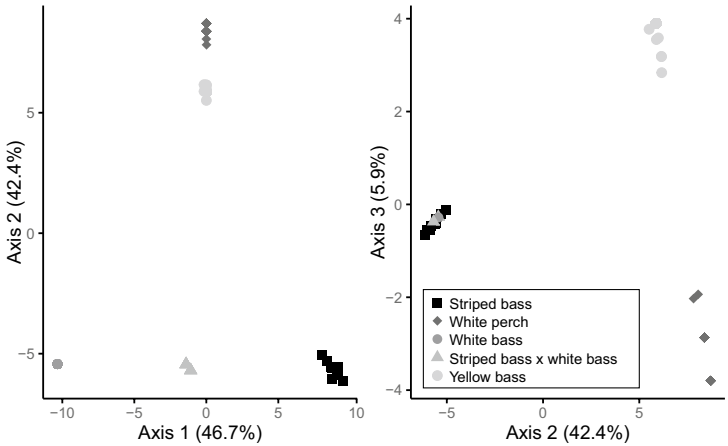
杂交检测应用
16个已知杂交个体在PCA图中均位于亲本物种中间位置,证实该面板可有效识别杂交事件。测试样本的缺失数据率<5%,分型成功率>95%。
该研究建立的SNP分型体系具有三大突破:首先,首次实现北美狼鲈属四物种+杂交种的同步鉴别;其次,GT-seq技术使单个样本成本降至传统方法的1/10;最后,47个SNP的组合克服了微卫星标记(Couch et al. 2006)跨物种扩增不稳定的缺陷。研究数据已开源共享(DOI:10.5281/zenodo.15297693),为各州自然资源部门开展种群监测、评估放流效果提供了标准化工具。值得注意的是,该方法在幼体鉴定和博物馆标本分析中展现特殊价值,未来可扩展应用于入侵物种溯源和杂交渐渗研究。
 生物通微信公众号
生物通微信公众号
知名企业招聘