
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于16S和23S rRNA反向疫苗学设计的鲍曼不动杆菌多表位疫苗构建与免疫信息学分析
【字体: 大 中 小 】 时间:2025年08月08日 来源:Future Journal of Pharmaceutical Sciences 3
编辑推荐:
本研究针对多重耐药(MDR)鲍曼不动杆菌(Acinetobacter baumannii)的临床威胁,采用反向疫苗学结合免疫信息学技术,以16S/23S rRNA为靶点设计新型疫苗。研究人员通过表位预测、分子对接及免疫模拟验证,构建出非过敏、高抗原性的多表位疫苗候选物,其与TLR-4强结合(-326.19 docking score)并能激发持久免疫记忆,为ESKAPE病原体防控提供新策略。
研究背景与意义
鲍曼不动杆菌(Acinetobacter baumannii)作为"ESKAPE"病原体成员,因其对碳青霉烯类等"最后防线"抗生素的耐药性,被WHO列为最高优先级威胁。该菌通过blaOXA-51基因和16S rRNA甲基化酶(armA)等机制获得耐药性,导致ICU内肺炎、败血症等院内感染死亡率攀升。传统疫苗策略如mRNA疫苗存在稳定性缺陷,而亚单位疫苗免疫原性不足。针对其高度保守的核糖体RNA(16S/23S rRNA)设计疫苗,可规避抗原变异难题,但相关研究尚属空白。
Helix Biogen Institute(尼日利亚奥格博莫索)的研究团队通过整合反向疫苗学与免疫信息学方法,成功构建靶向rRNA的多表位疫苗。该研究通过TLR-4激活验证和免疫模拟证实其诱导持久免疫的潜力,成果发表于《Future Journal of Pharmaceutical Sciences》,为耐药菌防控提供新思路。
关键技术方法
从NCBI获取93条16S/23S rRNA序列进行表位预测
使用ABCpred/Bepipred预测B细胞表位,NetMHCpan-4.0预测CTL表位
通过VaxiJen 2.0(阈值0.4)筛选抗原性表位,AllerTop 2.0排除过敏原
采用EAAAK/GPGPG/AAY连接子组装疫苗结构
AlphaFold2模拟三级结构,HDOCK验证TLR-4结合
C-ImmSim进行免疫响应动态模拟
主要研究结果
抗原表位筛选
从49条16S rRNA和44条23S rRNA序列中鉴定出核心表位:B细胞表位IHTYELERTHDARDVA(抗原性0.74)、CTL表位FEMDEEIEAL(结合HLA-A*02:01),以及HTL表位IGDRVLRTETAPVVA(诱导IL-4)。所有表位均通过非毒性、非过敏验证。
疫苗构建与特性
三组候选疫苗中,Construct 1含871个氨基酸,分子量91.25 kDa,不稳定指数36.25(<40为稳定)。其5'UTR-佐剂(LT-IIc B)-表位-3'UTR结构经RNAfold验证具有稳定二级结构
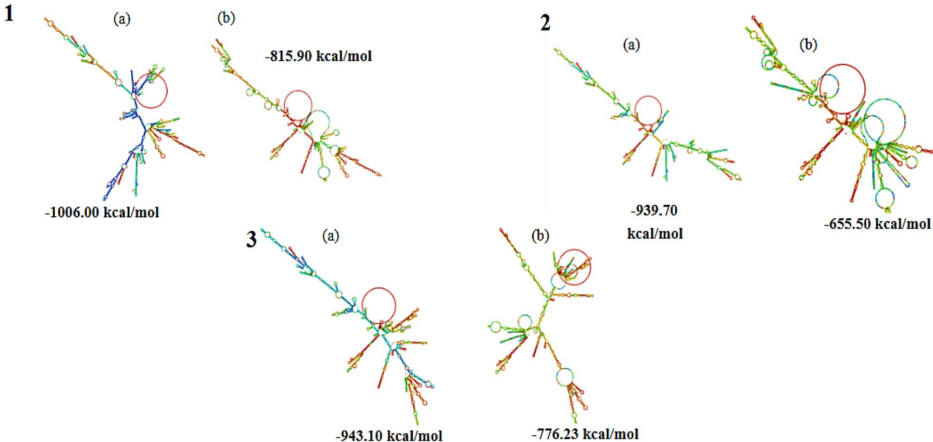
免疫原性验证
C-ImmSim模拟显示:
IgM/IgG抗体水平显著提升
记忆B细胞数量增加3倍
IFN-γ和IL-2分泌量超基准值200%
对亚洲人群覆盖率达95.15%
分子相互作用
Construct 1与TLR-4对接得分-326.19,优于其他构建体(-308.01/-309.89)。Ramachandran图显示94%残基处于优势构象
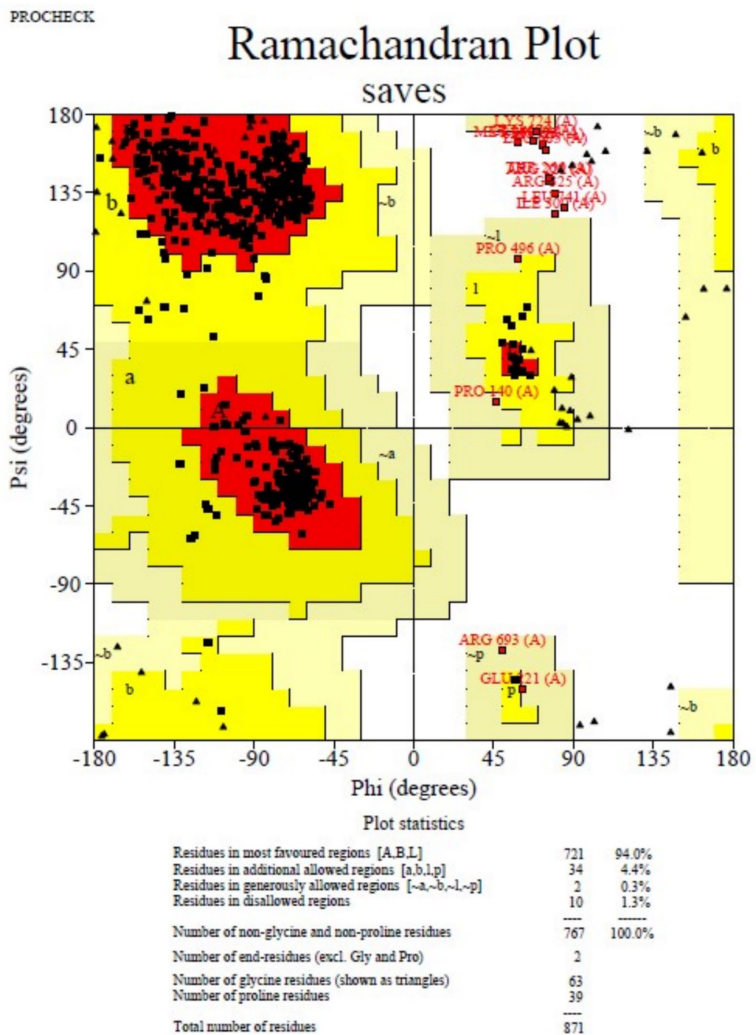
结论与展望
该研究首次证实rRNA疫苗对抗MDR鲍曼不动杆菌的可行性:
保守表位策略克服抗原变异挑战
多表位设计同时激活体液/细胞免疫
计算机优化使体外表达效率达99%(CAI=0.99)
局限在于5S rRNA数据缺失和部分地域人群覆盖率不足(欧洲仅12.46%)。后续需通过动物实验验证保护效力,并探索与碳青霉烯酶的协同抑制策略。这种反向疫苗学框架可扩展至其他ESKAPE病原体,为后抗生素时代提供新武器。
 生物通微信公众号
生物通微信公众号
知名企业招聘