
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于重构家系的长单倍型块分析与结构变异信号检测新方法
【字体: 大 中 小 】 时间:2025年08月08日 来源:Molecular Biology and Evolution 5.3
编辑推荐:
本研究针对基因组结构变异(SV)检测难题,开发了DoLoReS算法,通过分析重构家系(ARG)中单倍型块长度分布异常,成功识别出人类17q21.31倒位等50个重组抑制区域。该研究首次实现从SNP数据推断倒位变异,发现10号染色体760kb新倒位与南亚人群肺功能相关,为结构变异研究提供了全新视角。
基因组结构变异如同生命密码中的"错位拼图",长期以来因技术限制难以被完整解读。传统检测方法如配对末端测序存在高假阳性率,而基于连锁不平衡(LD)的分析仅适用于大片段高频变异。更棘手的是,当前祖先重组图(ARG)重建工具虽能处理数万样本,却普遍忽视结构变异的影响,导致深部分支重建存在偏差。
牛津大学统计系Anastasia Ignatieva团队在《Molecular Biology and Evolution》发表的研究,通过数学建模揭示了ARG中边缘跨度(edge span)与单倍型块长度的理论分布规律。研究人员开发出DoLoReS检测工具,利用Relate重建的2504例千人基因组计划(1KGP)数据,系统扫描了重组抑制信号。关键技术包括:SMC'模型推导边缘破坏概率、基于树序列(tskit)的ARG分析、SLiM软件模拟平衡选择下的倒位变异,以及HPRC长读长数据验证。

【边缘跨度分布重建】
通过解析SMC'模型发现,重组事件破坏边缘的概率与边缘年龄呈正相关。测试显示ARGweaver能准确重建边缘跨度,而tsinfer/tsdate会高估长跨度边缘。图2可视化显示,重建ARG中古老边缘的破坏概率被系统性低估,这与缺乏深层突变信号有关。

【单倍型块检测算法】
推导出单倍型块长度近似服从指数分布Exp(P??(G disrupted)·???·ρ/2)。针对Relate重建的ARG,开发了两种统计检验:检验1基于重组率积分计算跨度的显著性,检验2通过重组事件计数评估。图4显示经间断出现校正后,Relate重建的分布与理论值高度吻合。

【结构变异发现】
在1KGP数据中鉴定出50个显著区域(p<1·10-12),包括:
已知17q21.31倒位:在欧洲人群频率24%,年龄估算为8000-123000代,发现与CRHR1基因区古老的双交换事件证据(图7)。
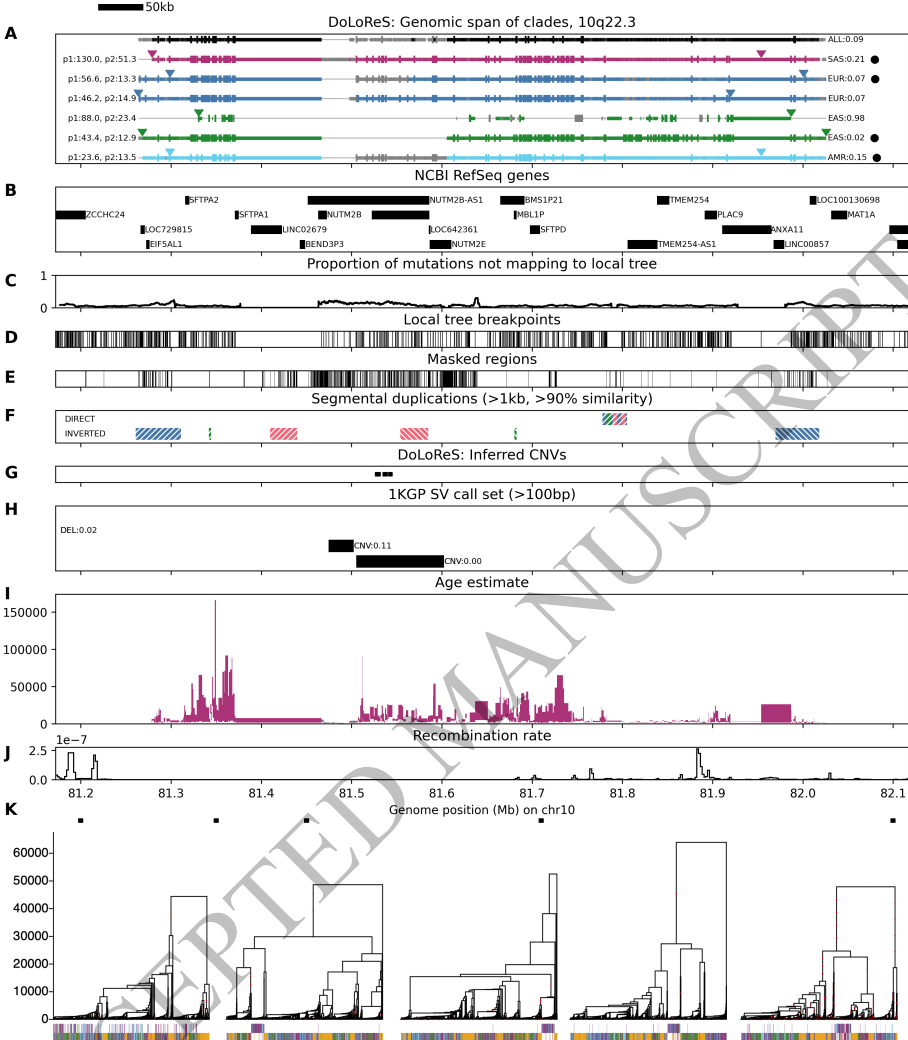
10q22.3新倒位:760kb片段在南亚人群频率21%,两侧存在50kb反向重复序列,HPRC数据验证5例携带者(图8)。该区域含肺表面活性蛋白基因,与血液尿素和血红蛋白水平显著相关。
16p12.2复杂多态性:原认为倒位,实为333kb片段重复导致的参考基因组错误组装。
【非结构变异机制】
26个无SV证据的区域显著富集于单基因边界(p=5·10-10),特别是雄性配子发生相关基因(如SCMH1、SPATA6),其eQTL与重组抑制显著相关(p=2·10-3),提示减数分裂基因表达量可能影响个体间重组率差异。
这项研究建立了ARG分析的新范式,证明SNP数据可揭示传统方法难以检测的结构变异。发现的10号染色体倒位为南亚人群疾病风险研究提供新线索,而减数分裂基因表达调控重组率的假说为群体遗传学开辟了新方向。该方法可推广至其他物种,为进化研究和医学遗传学提供强大工具。
 生物通微信公众号
生物通微信公众号
知名企业招聘