
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于深度对比学习的抗体NGS数据可视化工具deepNGS Navigator:实现高效克隆家族识别与功能聚类
【字体: 大 中 小 】 时间:2025年08月13日 来源:Bioinformatics 5.4
编辑推荐:
研究人员开发了deepNGS Navigator工具,通过语言模型和对比学习将抗体序列转化为直观的2D图谱,解决了大规模BCR序列分析中克隆家族检测和功能聚类难题。该工具在HER2靶向文库、COVID-19免疫序列等5类数据集验证中,展现出优于现有方法的聚类精度和生物学意义解析能力,为抗体发现提供了高效计算框架。
在抗体药物研发领域,海量的B细胞受体(BCR)测序数据就像一座未被充分挖掘的金矿。随着高通量测序技术的发展,单次实验就能产生数百万条抗体序列,这些数据蕴含着免疫系统对抗原应答的完整"作战方案"。然而,如何从这数据海洋中精准识别具有治疗潜力的抗体克隆,却成为困扰研究人员的难题。传统方法如系统发育树分析受限于计算效率和不完整的种系数据库,而常规降维可视化技术又难以保持序列间的功能关联性。更棘手的是,骆驼等非模式生物的抗体研究还面临种系注释缺失的挑战。
针对这些瓶颈,Genentech公司Prescient Design团队开发了革命性的deepNGS Navigator分析平台。这项发表在《Bioinformatics》的研究,创新性地将自然语言处理领域的BERT模型与对比学习相结合,实现了抗体序列空间的智能导航。就像为抗体数据打造了"谷歌地图",不仅能清晰标注各克隆家族的"地理位置",还能显示它们之间的"进化路线图"。
研究团队采用三大关键技术:1)基于Absolve的NGS数据预处理流程,支持核苷酸/氨基酸序列的合并与去重;2)定制化BERT语言模型训练,通过对比学习定义CDR3区域特异性邻域关系;3)改进的t-SimCNE算法进行2D投影,结合Leiden算法实现自动聚类。特别值得注意的是,该方法突破性地摆脱了对V(D)J基因注释的依赖,仅凭序列信息就能重建克隆关系。
研究结果通过五个典型案例验证了工具的普适性:
"酵母展示文库的FACS标记聚类"显示,deepNGS Navigator在HER2靶向文库分析中,相比传统方法产生更紧凑的簇(平均100簇),且结合标签熵降低30%。
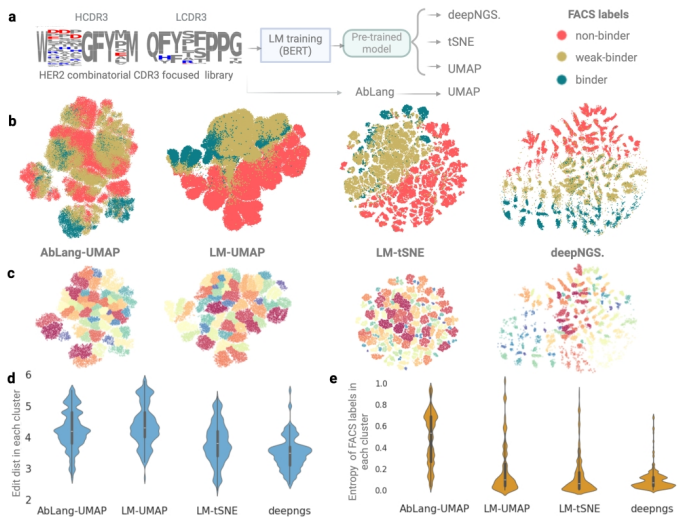
"合成数据集层次结构分析"证实,该方法2D投影与序列编辑距离的相关系数达0.94,准确反映了GP/EN等生成模型产生的层级关系。

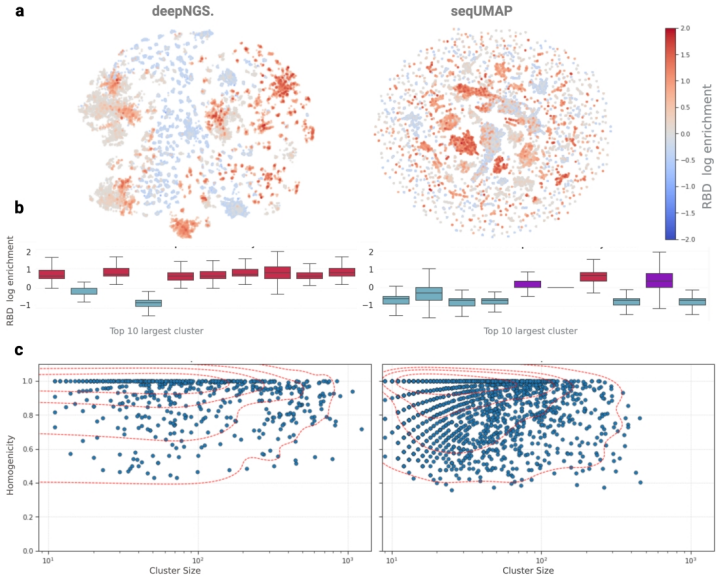
"骆驼抗COVID-RBD抗体挖掘"实验中,deepNGS Navigator识别出的前10大簇中有8个为富集簇,纯度显著高于seqUMAP方法。
"初始与记忆B细胞区分"研究显示,仅凭原始序列就能分离IGHV3/4/5基因亚型,并再现从初始到记忆B细胞的进化轨迹。

"模拟克隆谱系重建"测试中,在线噪比1:1的挑战性数据下,仍达到99%的聚类准确率,优于现有最佳方法。

这项研究的突破性在于建立了抗体序列分析的"通用坐标系"。通过语言模型捕捉的"抗体语法"和对比学习构建的进化拓扑,研究者首次实现了:1)万级序列的分钟级可视化;2)无监督的克隆家族识别;3)跨物种应用的普适性分析。该工具已开源(GitHub: prescient-design/deepngs-navigator),其模块化设计支持整合结构预测等扩展功能。对于抗体发现流程,这意味着可以从浩如烟海的候选分子中,快速锁定最具开发潜力的克隆家族,将传统需要数月的筛选工作压缩至数天,为下一代抗体药物的理性设计开辟了新途径。
 生物通微信公众号
生物通微信公众号
知名企业招聘