
-

生物通官微
陪你抓住生命科技
跳动的脉搏
LABind:基于图Transformer与跨注意力机制的蛋白质-配体结合位点智能预测新方法
【字体: 大 中 小 】 时间:2025年08月21日 来源:Nature Communications 15.7
编辑推荐:
本研究针对蛋白质-配体结合位点预测中实验成本高、单配体方法泛化性差、多配体方法缺乏配体编码等难题,开发了名为LABind的深度学习模型。该模型通过图Transformer捕捉蛋白质局部空间特征,结合跨注意力机制学习配体特异性结合模式,在DS1-DS3基准测试中全面超越现有方法(平均MCC提升21.8%),首次实现未见配体的高精度预测,并为分子对接提供关键支持。
蛋白质与配体的相互作用是生命活动的核心环节,从酶催化到信号传导都依赖这种分子识别过程。虽然X射线晶体学等技术能精确解析结合位点,但高昂的成本限制了其广泛应用。更棘手的是,现有计算方法要么像IonCom、MIB等单配体方法只能预测特定配体,要么如P2Rank等多配体方法忽视配体特性差异。这种困境随着蛋白质结构数据库的爆炸式增长(UniProt已收录超2亿条序列)愈发凸显——我们亟需能同时理解蛋白质结构和配体化学特性的智能预测工具。
为此,深圳大学Zhijun Zhang团队在《Nature Communications》发表研究,提出了革命性的LABind模型。该研究整合了三大技术创新:首先采用Ankh和MolFormer预训练模型分别提取蛋白质序列和配体SMILES的深度特征;其次构建包含原子空间坐标的蛋白质图网络;最终通过跨注意力机制实现蛋白质-配体的动态特征交互。这种"预训练+几何深度学习+注意力机制"的架构,使模型既能捕捉结合位点的共性规律,又能区分不同配体的特异性。
关键技术包括:1) 使用2626个实验解析的蛋白质链构建DS1-DS3基准数据集;2) 通过DSSP算法提取蛋白质二级结构特征;3) 基于MSMS计算的蛋白质表面距离特征;4) 图Transformer编码残基-残基相互作用;5) 多任务学习框架整合3651种配体数据。
性能验证
在包含474种未见配体的DS3测试集上,LABind的MCC(0.510)显著优于LigBind(0.379),特别是对Zn2+等金属离子的预测精度提升34.4%。如图1所示,模型能准确区分5XWK_A蛋白中Zn2+与PO43-的重叠位点和Ca2+的独立位点。
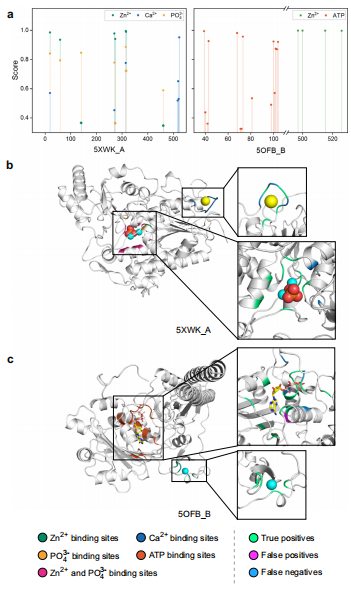
应用拓展
将预测结果用于分子对接时,LABind使Smina的对接成功率提升19.1%(图5)。在SARS-CoV-2 NSP3宏结构域案例中,模型对未训练配体QU3的预测精准度达100%,而传统方法出现8个假阳性。
机制解析
图6的消融实验揭示:蛋白图网络贡献最大(移除后AUPR下降0.284),配体特征使Ca2+识别率提升57.5%。t-SNE可视化显示,跨注意力机制使结合/非结合残基的特征分布显著分离。
这项研究创立了首个真正意义上的"配体感知"预测范式,其创新性体现在三方面:1) 突破单配体方法的局限性,统一模型支持>3000种配体;2) 通过预训练特征融合解决小样本配体学习难题;3) 开发出适用于预测结构的鲁棒性算法。该成果不仅为药物靶点发现提供新工具,其"几何深度学习+注意力"框架更为蛋白质功能预测开辟了新方向。
 生物通微信公众号
生物通微信公众号
知名企业招聘