
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于动态对齐的DANet模型:从H&E组织学图像预测空间基因表达的新方法
【字体: 大 中 小 】 时间:2025年08月22日 来源:Briefings in Bioinformatics 7.7
编辑推荐:
本研究针对病理组织学图像与空间基因表达预测中的关键挑战,提出创新性解决方案DANet模型。通过引入密集连接结构(DenseNet)实现特征高效复用,结合状态空间模型(SSM)捕捉基因间相关性,并设计残差Kolmogorov-Arnold网络(RKAN)实现双模态动态对齐。在GSE240429和HER2+数据集上的实验表明,该方法较现有最优模型BLEEP在标记基因(MG)预测准确率提升22%,为临床诊断和个性化医疗提供新工具。
在生物医学领域,病理切片与基因表达的关系犹如"形态"与"功能"的密码本。传统空间转录组技术如MERFISH和Visium虽能解码这种关联,却面临设备昂贵、耗时长的瓶颈。更棘手的是,病理图像中密集复杂的细胞结构使得局部精细特征提取困难,而基因间相互作用又涉及全局生物学机制,传统CNN和MLP难以捕捉。这些挑战催生了Yulong Wu等学者在《Briefings in Bioinformatics》发表的突破性研究。
研究团队创新性地构建了DANet模型,通过三大核心技术实现突破:1)采用DenseNet架构提取组织图像特征,通过密集跨层连接实现特征高效复用;2)引入基于状态空间模型(SSM)的Mamba模块,通过选择性状态转移机制建模基因长程依赖关系;3)设计残差Kolmogorov-Arnold网络(RKAN),利用可学习激活函数动态调整双模态映射。实验采用GSE240429(人类肝脏组织)和HER2+(乳腺癌)两个公开数据集,通过对比学习框架进行训练验证。
方法创新
研究采用对比学习框架,图像特征通过DenseNet提取后,基因表达数据经SSM模块处理,两者通过RKAN投影到256维空间进行对齐。RKAN模块创新性地结合了KAN网络的函数分解能力和残差连接,通过下采样-上采样操作和GELU激活实现动态特征调整。损失函数采用改进的CLIP对比损失,引入温度系数τ调节相似度分布。
关键结果
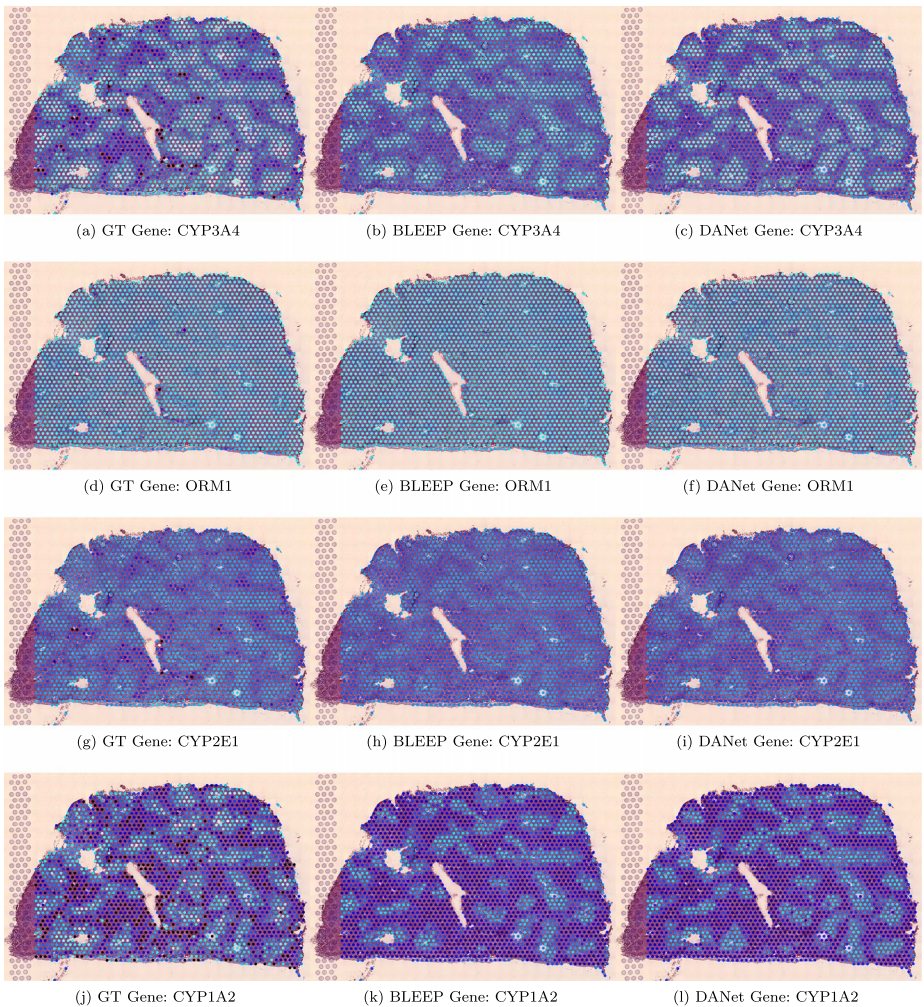
性能优势:在GSE240429数据集上,DANet对标记基因(MG)预测的Pearson相关系数(PCC)达0.265±0.003,较BLEEP提升22%。如图2所示,CYP3A4等基因表达预测结果与真实值(GT)的空间分布高度一致。
模块贡献:消融实验显示,移除SSM模块使PCC下降12.5%,证实其对基因相关性建模的关键作用。RKAN模块在HER2+数据集E1子集使PCC提升14.6%,体现动态对齐优势。
生物学意义:如图4所示,经基因计数均值归一化后,预测值(红点)与真实值(蓝线)高度吻合,表明模型能准确反映基因表达水平差异。
结论与展望
该研究通过动态对齐策略成功建立了病理图像与基因表达的跨模态关联,其创新性体现在:1)首次将SSM应用于基因关系建模,突破传统神经网络局部感知局限;2)RKAN模块为多模态医学数据分析提供新范式。临床层面,该方法可降低对昂贵基因测序的依赖,助力肿瘤微环境研究。未来研究可探索图神经网络与SSM的融合,进一步提升对空间异质性的建模能力。
这项由重庆大学团队主导的研究,通过开源代码(https://github.com/202324131016T/DANet)推动领域发展,为精准医疗时代的跨模态分析树立了新标杆。
 生物通微信公众号
生物通微信公众号
知名企业招聘