
-

生物通官微
陪你抓住生命科技
跳动的脉搏
芹菜与欧芹根际土壤微生物组16S rRNA和ITS基因扩增子测序数据集揭示间作系统的微生物群落动态
【字体: 大 中 小 】 时间:2025年08月26日 来源:BMC Genomic Data 2.5
编辑推荐:
本研究通过16S rRNA(V3-V4区)和ITS扩增子测序技术,解析了芹菜(Apium graveolens)与欧芹(Petroselinum crispum)单作和间作系统下根际微生物群落结构。研究发现Actinobacteria和Proteobacteria为优势细菌门,Ascomycota和Mortierellomycota为优势真菌门,为可持续农业中植物-微生物互作机制研究提供了重要数据支撑。
研究背景
芹菜和欧芹作为全球广泛使用的药食同源植物,常被农民采用间作模式种植以优化资源利用。然而,这种耕作方式如何影响根际微生物组的动态变化尚不明确。根际作为植物与土壤相互作用的"热区",其微生物群落结构直接影响作物健康和土壤肥力。尤其在南非Gauteng省(26°05'41.3"S 27°38'03.3"E)的特定农业生态系统中,理解这两种植物间作对微生物组的影响,对开发可持续种植策略具有重要意义。
关键技术方法
研究团队采集了单作、间作及对照土壤样本,使用NucleoSpin Soil试剂盒提取DNA后,采用Illumina NovaSeq 6000平台进行16S rRNA和ITS扩增子测序。通过QIIME 2(2019.1版)流程处理数据,应用DADA2算法生成高精度ASV(Amplicon Sequence Variants),并利用Silva 138.1和UNITE 8.0数据库进行物种注释。功能预测分别采用PICRUSt2和FUNGuild工具。
研究结果
微生物群落组成
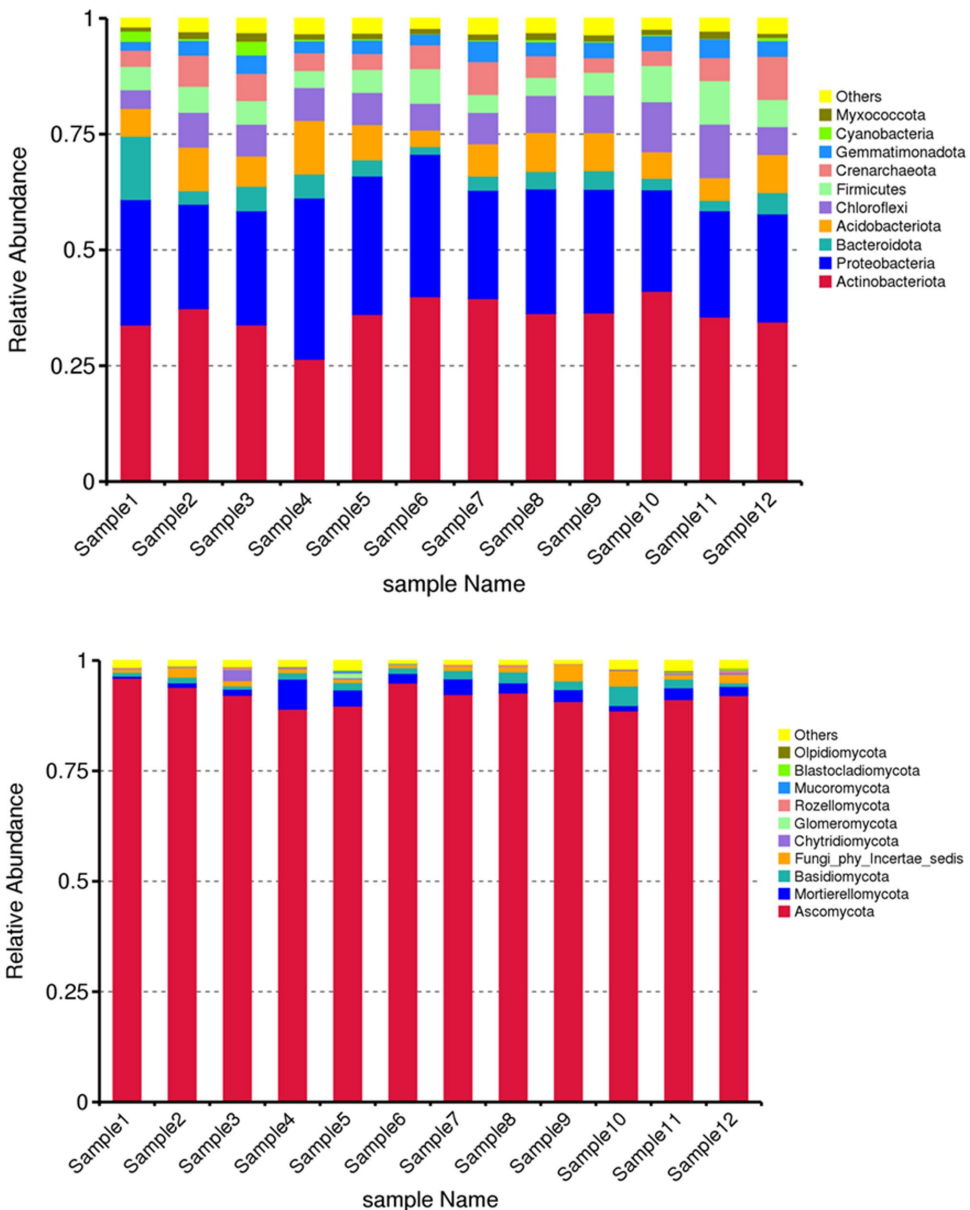
细菌群落以Actinobacteria(28.7%)和Proteobacteria(25.3%)为主,间作系统显著提高了Gemmatimonadota的丰度。真菌群落中Ascomycota占比达61.2%,间作使Mortierellomycota相对丰度提升1.8倍。
α多样性分析
Chao1指数显示间作系统细菌多样性较单作提高17.3%,而真菌Shannon指数在欧芹单作中最高。系统发育多样性(PD whole tree)表明间作改变了微生物功能基因的分布格局。
β多样性差异
PCoA分析揭示间作与单作样本在坐标轴上明显分离(ANOSIM R=0.42,p<0.01),表明耕作方式显著影响微生物群落结构。
功能预测
PICRUSt2分析显示间作系统富集了氮循环相关基因(nifH、narG),FUNGuild预测病原真菌在单作系统中相对丰度更高。
研究结论
Oluhukola Oluranti Babalola团队首次系统揭示了芹菜-欧芹间作通过改变根际微环境,显著提高了有益微生物(如Plant Growth Promoting Rhizobacteria)的丰度,同时降低了土传病原菌负荷。该研究为开发基于微生物组的精准间作方案提供了理论依据,相关数据已存入NCBI SRA(SRP540554和SRP540675)。发表在《BMC Genomic Data》的这项成果,对实现资源高效利用的生态农业具有重要指导价值。
 生物通微信公众号
生物通微信公众号
知名企业招聘