
-

生物通官微
陪你抓住生命科技
跳动的脉搏
钙成像网络分析新利器:CalciumNetExploreR助力神经元功能连接研究
【字体: 大 中 小 】 时间:2025年08月27日 来源:BMC Bioinformatics 3.3
编辑推荐:
研究人员针对钙成像数据下游分析工具匮乏的现状,开发了R语言包CalciumNetExploreR(CNER),整合归一化、二值化、网络构建与拓扑分析等功能,通过斑马鱼模型验证发现AKT1过表达导致神经元集群系数(Cg)降低41%、全局效率(Gg)下降33%,为研究神经退行性疾病和肿瘤机制提供新工具。
在神经科学研究中,钙离子(Ca2+)动态是解码神经元通信的核心线索。尽管GCaMP等荧光探针技术已能捕获毫秒级的钙信号,但海量的时序数据处理仍令研究者头疼——现有工具如Suite2p、CaImAn仅擅长信号提取,而Brain Connectivity Toolbox等网络分析平台又缺乏钙成像适配性。这种"数据丰富但工具割裂"的现状,促使爱丁堡大学的Simone Lenci和Dirk Sieger团队开发了CalciumNetExploreR(CNER),这一成果近期发表于《BMC Bioinformatics》。
研究团队采用模块化设计思路,将分析流程浓缩为三大核心技术:1)自适应预处理(含min-max归一化xt'=(xt-min(x))/(max(x)-min(x))和2σ阈值二值化);2)基于互相关分析(ccf()函数计算时滞τ=-1,0,+1的ρij,max)构建功能网络;3)拓扑量化(含聚类系数C(g)=∑2ei/ki(ki-1)、全局效率G(g)=∑1/dij/N(N-1)等)。所有分析均通过Tg(NBT:H2B-GCaMP6s)斑马鱼模型获取的450帧时序数据验证。
数据预处理
通过min-max归一化消除GCaMP6s表达差异,采用μ+2σ阈值识别活性事件,发现AKT1过表达组(TREAT)标记神经元活动频率达4.00±0.10次/分,较对照组(CTRL)2.89±0.09次显著升高(p<0.001)。
网络特征
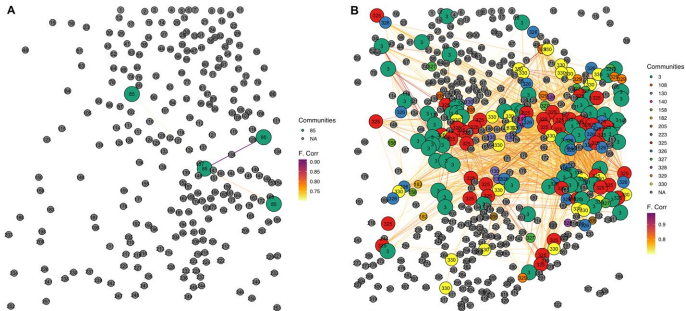
拓扑分析显示TREAT组聚类系数Cg=0.547±0.011较CTRL组0.772±0.010降低41%(p=0.0009),网络图显示其大集群数量从14个锐减至1个。标记-未标记神经元连接比例(LtU)下降50%,提示AKT1导致功能隔离。
动态模式
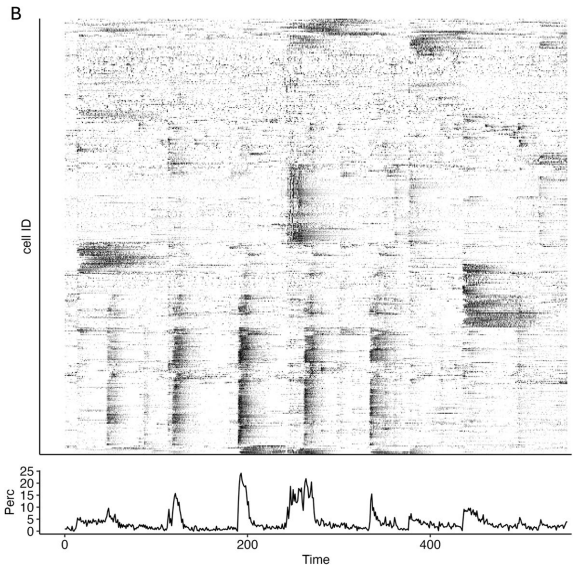
主成分分析揭示TREAT组前5主成分解释方差仅8.80%±0.35%(CTRL组12.38%±1.02%),Scree曲线更平缓,反映其活动模式复杂性增加。功率谱分析则显示γ波段活动紊乱。
这项研究创新性地将图论分析与钙成像数据深度融合,CNER不仅解决了多工具切换的痛点,其发现的AKT1致网络碎片化现象为肿瘤神经侵袭机制提供了新视角。作者特别指出,软件内置的subset_connections()功能可精准量化特定神经元亚群(如肿瘤相关神经元)的整合程度,这对研究癫痫、阿尔茨海默病等网络性疾病具有普适价值。未来通过集成机器学习算法,或将实现从静态网络到动态预测的跨越。
 生物通微信公众号
生物通微信公众号
知名企业招聘