
-

生物通官微
陪你抓住生命科技
跳动的脉搏
染色体水平基因组揭示砂仁(Wurfbainia villosa)药用价值与育种潜力
【字体: 大 中 小 】 时间:2025年08月27日 来源:Scientific Data 6.9
编辑推荐:
本研究针对传统中药砂仁(Wurfbainia villosa)产业面临的种质退化、产量低下等瓶颈问题,通过ONT+NGS+Hi-C多组学技术,完成2.73Gb染色体水平基因组组装(Contig N50达30.23Mb),锚定24条假染色体并注释42,713个基因。首次揭示该物种88.83%的重复序列特征及LTR转座子主导的基因组结构,为解析其活性成分生物合成通路及分子育种奠定基础。
作为"四大南药"之一的砂仁(Wurfbainia villosa),其干燥果实Fructus Amomi在胃肠疾病治疗和食品保健领域应用广泛,年产值超千亿元。然而自然授粉困难、萌发率低等农业难题,加之种质资源遗传多样性匮乏导致的品质退化,严重制约产业发展。更关键的是,作为主要活性成分的挥发油生物合成机制尚未在基因组层面阐明,传统育种缺乏分子标记支撑。
为解决这些问题,Yingmin Zhang等研究团队在《Scientific Data》发表了首个染色体水平的砂仁基因组。研究者整合Oxford Nanopore长读长测序、Illumina短读长校正和Hi-C染色体构象捕获技术,从云南文山采集的样本中构建了2,731.09Mb的高质量基因组。通过19-mer分析预估基因组大小为2,729.62Mb,杂合率仅0.63%。采用NextDenovo组装结合Racon抛光,最终获得Contig N50达30.23Mb的组装结果,其中99.91%序列成功锚定至24条假染色体。
关键方法:样本采自云南文山(23°34'N,104°45'E),使用ONT PromethION P48平台获取108×长读长数据,结合DNBSEQ-T7产出的84×短读长进行校正。Hi-C数据通过Juicer等工具辅助染色体挂载,EDTA软件注释重复序列,MAKER整合三种基因预测方法。
基因组特征:组装评估显示BUSCO完整性达95.1%,重复序列占比88.83%(其中LTR/Copia占55.19%)。Hi-C互作热图证实染色体组装准确性,24条染色体长度介于66.82-155.81Mb之间。与既往研究相比,本组装Contig N50提升3倍以上(表1)。
基因注释:预测42,713个蛋白编码基因,平均含4.97个外显子,平均CDS长度1,105.59bp。注释完整性经BUSCO验证达97.6%,显著优于同类研究(表5)。特别值得注意的是,LTR反转座子占比达78.38%,这种转座子爆发可能驱动了物种适应性进化。
讨论与意义:该研究创制了目前最完整的砂仁基因组资源,其染色体水平组装为解析萜类合成通路(如调控挥发油生成的terpene synthases)提供分子基础。基因组揭示的LTR转座子扩张模式,为解释Zingiberaceae科物种进化提供新视角。提出的DNA-informed breeding策略可突破传统育种瓶颈,对保障"药食同源"作物品质安全具有重大应用价值。
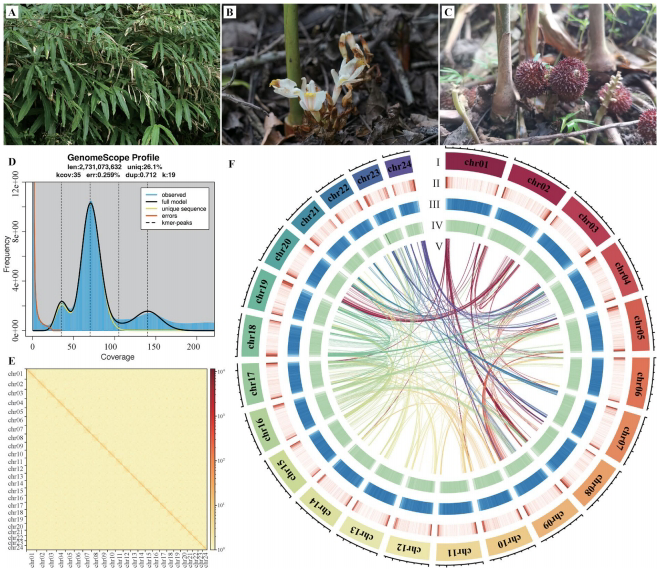
研究首次将砂仁染色体可视化呈现(图1F),其中III-V轨道展示的基因密度与重复序列分布规律,为后续比较基因组学研究提供重要参照。团队特别指出,该资源将加速开发与挥发油含量相关的分子标记,推动实现《中国药典》规定的品质标准化控制。
 生物通微信公众号
生物通微信公众号
知名企业招聘