
-

生物通官微
陪你抓住生命科技
跳动的脉搏
AlphaFold 3在糖链建模中的应用:能力、局限与糖蛋白相互作用研究新突破
【字体: 大 中 小 】 时间:2025年08月29日 来源:Glycobiology 3.3
编辑推荐:
本研究针对糖链结构复杂性和动态性带来的建模挑战,利用AlphaFold 3(AF3)开发了基于bondedAtomPairs(BAP)语法的糖链建模方法。通过系统评估不同输入格式(SMILES/userCCD/CCD),研究人员成功构建了N-糖链、O-糖链及脂质连接糖复合物的立体化学精确模型,并验证了其与糖基转移酶、凝集素的相互作用。该研究为糖生物学机制研究提供了标准化建模工具,填补了AI预测在糖科学领域的应用空白。
糖链作为生命体中结构最复杂的生物分子之一,其立体化学多样性和动态特性长期制约着糖科学的发展。超过半数人类蛋白质存在糖基化修饰,这些糖链在免疫调控、病原体识别等过程中发挥关键作用,但传统实验方法难以捕捉其瞬时构象。随着AlphaFold系列算法在蛋白质结构预测领域的突破,最新发布的AlphaFold 3(AF3)首次扩展了多生物分子建模能力,为糖链研究带来了新机遇。
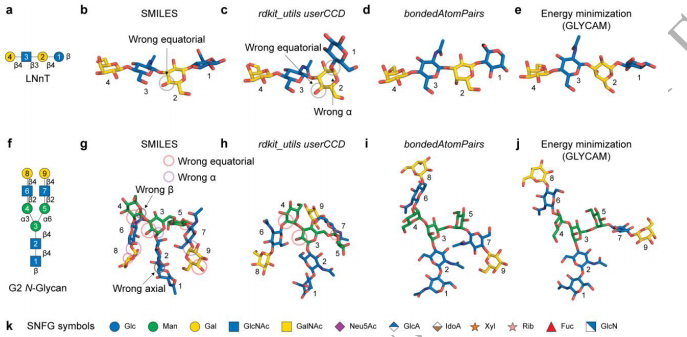
美国乔治亚大学的Chin Huang、Natarajan Kannan和Kelley W. Moremen团队在《Glycobiology》发表的研究,系统探索了AF3在糖链建模中的潜力。研究人员发现,直接使用简化分子线性输入规范(SMILES)或用户自定义化学组分词典(userCCD)会导致糖链立体化学错误,如半乳糖(Gal)C2羟基轴向/赤道取向错误、糖苷键α/β构型误判等。通过创新性地采用化学组分词典(CCD)编码的单糖模块与bondedAtomPairs(BAP)语法联用,成功构建了乳糖-N-新四糖(LNnT)、双天线N-糖链G2等复杂糖链的精确模型。
关键技术包括:1)基于GLYCAM的能量最小化糖链结构验证;2)通过JSON文件定义CCD编码与BAP共价连接;3)与PDB晶体结构(如MAN1A1/M9/La3+复合物)进行RMSD比对;4)跨膜蛋白CD22等多结构域糖蛋白的全体系建模。
研究结果揭示:
糖链输入语法的优化验证
通过比较SMILES、rdkit_utils转换的userCCD和CCD+BAP三种输入方式,证明BAP语法能准确保持糖链立体化学特性。如M9 N-糖链在α-甘露糖苷酶MAN1A1结合时,末端Man9残基从溶液相的4C1椅式构象转变为催化所需的3OB船式构象,与GH47家族酶反应坐标理论相符。
糖-蛋白复合物的精准预测
AF3成功复现了糖基转移酶MGAT2与A1 N-糖链的催化复合物(RMSD 0.241 ?),其中3-臂GlcNAc结合于外显位点,6-臂Man伸入活性中心与UDP-GlcNAc配位。对于唾液酸转移酶ST6GAL1,模型准确捕捉了其对G2F糖链3-臂的优先识别特性。
脂质连接糖链的拓展应用
研究首次实现神经节苷脂GP1c、Globo H等糖鞘脂的完整建模。当Globo H与伯克霍尔德菌凝集素Bc2L-C形成三聚体复合物时,模型与晶体结构(6TIG)的RMSD仅0.137 ?。多萜醇焦磷酸(Dol-PP)连接的几丁二糖则正确呈现了N-糖链生物合成中罕见的α-糖苷键构型。
O-糖链的动态建模突破
针对难以结晶的O-连接糖链,AF3构建了核心1-4结构、硫酸角质素(KS)及O-Man核心M3糖链模型。其中包含磷酸核糖醇的非经典糖链骨架建模,为肌营养不良相关糖链研究提供新工具。
跨膜糖蛋白的构象解析
全长CD22(Siglec-2)的11个N-糖基化位点被同时建模,首次可视化其IgV结构域通过G2S2糖链介导的顺式相互作用,以及6-O-硫酸化GlcNAc(GlcNAc6S)对反式结合的调控机制。
讨论部分指出,AF3的扩散生成架构使其能超越训练集范围预测新型糖-蛋白相互作用,如对2023年后新发布的B3GALT5/核心3 O-糖链复合物(8ZX3)仍能达到0.096 ?精度。但需注意静态模型的局限性:1)Fc结构域N-糖链因晶体数据偏倚难以模拟加工酶作用;2)硫酸乙酰肝素(HS)长链建模需手动修正;3)matriglycan中α-木糖易被误判为β构型。
该研究建立的糖链建模标准流程,不仅推动了糖科学从"结构盲区"向机制研究的转变,其CCD+BAP的模块化策略更为复杂生物分子体系(如GPCR-配体复合物)的AI预测提供了范式。随着糖链数据库的扩充与算法迭代,AF3有望成为糖疫苗设计、糖基化抗体优化等转化医学研究的核心工具。
 生物通微信公众号
生物通微信公众号
知名企业招聘