
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于ddRAD测序的亚马逊河虾Macrobrachium amazonicum复合体遗传结构与物种界定研究
【字体: 大 中 小 】 时间:2025年08月30日 来源:Scientific Reports 3.9
编辑推荐:
本研究通过ddRAD测序技术获取969个SNP标记,首次从基因组层面解析了Macrobrachium amazonicum复合体(含M. pantanalense)的种群结构与系统发育关系,证实了M. pantanalense的独立物种地位,揭示了亚马逊内陆与沿海种群间的遗传分化,为淡水虾类物种界定和资源管理提供了分子依据。
淡水虾类Macrobrachium因其生态与经济价值备受关注,但该属物种存在显著的分类学争议。以广泛分布于南美洲的亚马逊河虾Macrobrachium amazonicum为例,其内陆淡水种群与沿海种群虽形态相似,却表现出不同的生理适应性。更复杂的是,原被认为属于该物种的巴拉圭河种群因独特的生殖隔离特征被命名为新种M. pantanalense,但线粒体标记(如COI基因)仅显示0-3.3%的遗传差异,难以支撑物种划分。这种矛盾使得"Macrobrachium amazonicum复合体"的分类地位长期悬而未决,也阻碍了针对不同种群的精准保护策略制定。
为破解这一难题,Gabriel Monteiro de Lima等团队在《Scientific Reports》发表研究,首次采用双酶切简化基因组测序(ddRAD-Seq)技术,对来自巴西三个地理种群(Mosqueiro沿海型、Tucuruí人工隔离种群、Santarém内陆型)的M. amazonicum及M. pantanalense(Bela Vista de Goiás)共39个样本进行全基因组SNP分析。通过群体遗传学与系统发育学方法,揭示了该复合体的真实演化关系。
关键技术方法
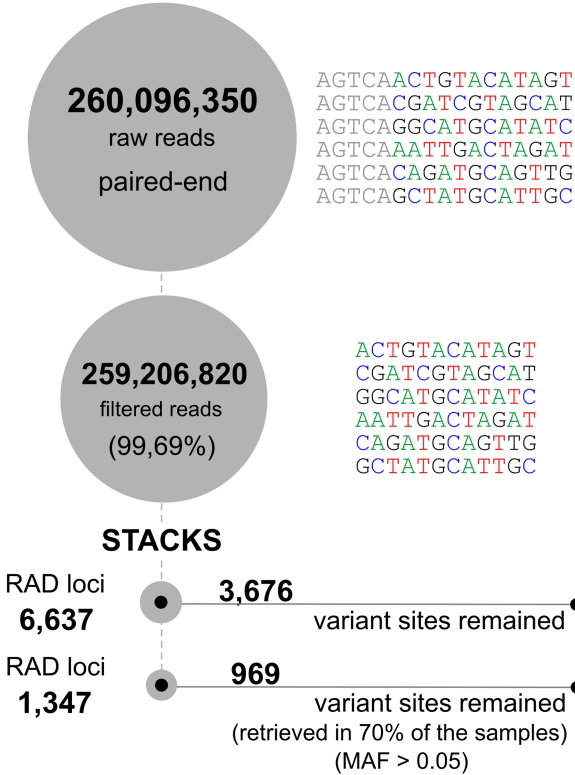
研究通过EcoRI和MspI酶切构建ddRAD文库,使用Illumina NextSeq500平台进行双端测序。经STACKs流程比对参考基因组(Macrobrachium nipponense)后,筛选出969个高质量SNP位点(MAF>0.05,检出率≥70%)。采用STRUCTURE、DAPC分析种群结构,RAxML和MrBayes构建系统发育树,并通过BFD*进行物种界定检验。
研究结果
1. 分子鉴定与SNP标记特征
COI序列分析确认样本分类地位,但仅显示0-0.32%的种内差异。而ddRAD数据获得562个转换和407个颠换位点,其中M. pantanalense独有235个私有等位基因,显著高于M. amazonicum种群(89-107个)。
2. 种群遗传分化
沿海与内陆M. amazonicum种群呈现高遗传分化(FST=0.184-0.237),尤以Santarém内陆种群最为突出。M. pantanalense与所有M. amazonicum种群的FST高达0.870-0.928,基因流仅0.04-0.07。
3. 遗传结构与系统发育
STRUCTURE和DAPC分析均支持K=4的最佳分组,对应三个M. amazonicum地理种群和一个M. pantanalense单系群。最大似然法和贝叶斯推断均以100%支持度确认M. pantanalense的独立分支地位。
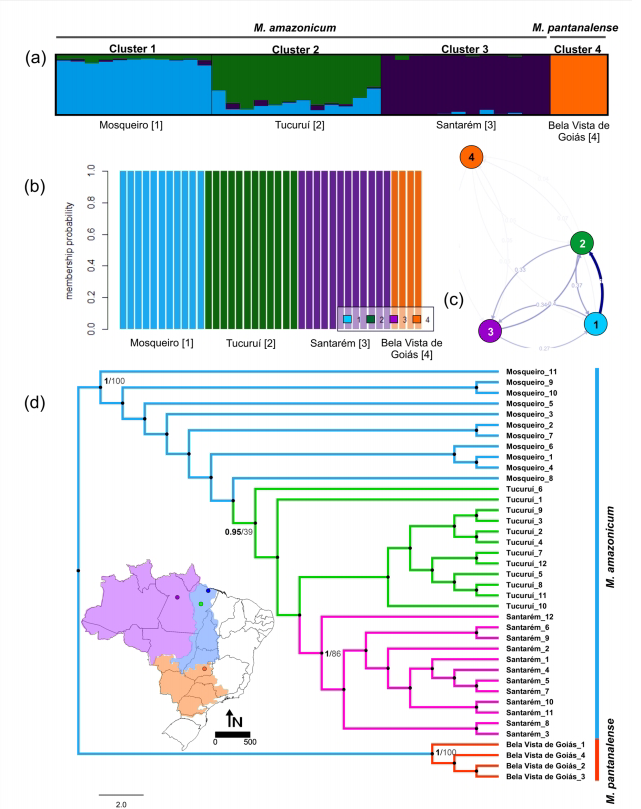
4. 物种界定检验
BFD*分析强烈支持三物种模型(边际似然值-3881.7764),将M. pantanalense、沿海型(Mosqueiro+Tucuruí)和内陆型(Santarém)划分为独立演化单元。
结论与意义
该研究通过基因组SNP数据首次从分子层面验证了M. pantanalense的物种有效性,解决了线粒体标记分辨率不足导致的分类争议。同时发现亚马逊内陆Santarém种群可能正处于与沿海种群的分化过程中,这与其独特的寄生等位基因(如Probopyrus sp.寄生虫特异性)和淡水适应表型相吻合。研究为淡水甲壳动物物种形成机制提供了新案例,强调需针对不同遗传种群制定差异化管理策略——特别是对过度捕捞的北部沿海种群。技术层面,ddRAD-Seq在解决低分歧类群系统发育问题上的优势得到充分体现,为同类研究提供了范式。
研究也存在样本量局限(如M. pantanalense仅4个样本),未来需扩大地理采样以验证遗传分化格局。该成果不仅推动了对Macrobrachium属演化历史的认知,也为生物多样性保护与水产资源可持续利用提供了科学依据。
 生物通微信公众号
生物通微信公众号
知名企业招聘