
-

生物通官微
陪你抓住生命科技
跳动的脉搏
狐猴基因组结构变异图谱揭示濒危物种的遗传多样性危机
【字体: 大 中 小 】 时间:2025年08月30日 来源:Genome Biology and Evolution 2.8
编辑推荐:
本研究首次系统描绘了濒危物种指猴(Daubentonia madagascariensis)全基因组结构变异(SV)图谱。研究人员通过整合DELLY/LUMPY/MANTA多算法分析14个个体的高通量测序数据,鉴定出1,133个影响240kb基因组的SV事件,发现85%为短片段缺失(<200bp),并鉴定出两个位于疾病相关基因的大片段缺失。该研究为理解灵长类SV进化模式提供了重要基线数据,对濒危物种保护具有指导意义。
在生物多样性热点地区马达加斯加,指猴(Daubentonia madagascariensis)作为全球25种最濒危的灵长类动物之一,其种群数量已锐减至不足万只。这种夜行性灵长类不仅具有独特的形态特征——如特化的中指用于敲击树干觅食,更是生态系统中重要的种子传播者。然而栖息地碎片化导致的遗传多样性丧失,使得该物种面临严重的近交衰退风险。传统保护遗传学研究多聚焦单核苷酸变异(SNV),而对影响更大基因组区域的结构变异(Structural Variation, SV)却知之甚少。
随着指猴高质量基因组组装(DMad_hybrid)的完成,来自亚利桑那州立大学的Cyril J. Versoza团队在《Genome Biology and Evolution》发表了开创性研究。研究团队采用三重技术路线:首先通过Illumina NovaSeq平台获取14个个体(来自10个亲子 trio)平均46.1x覆盖度的全基因组数据;随后运用BWA-MEM比对和GATK标准流程进行质控;最终整合DELLY、LUMPY和Manta三种SV检测算法,通过SURVIVOR软件构建高置信度SV数据集。
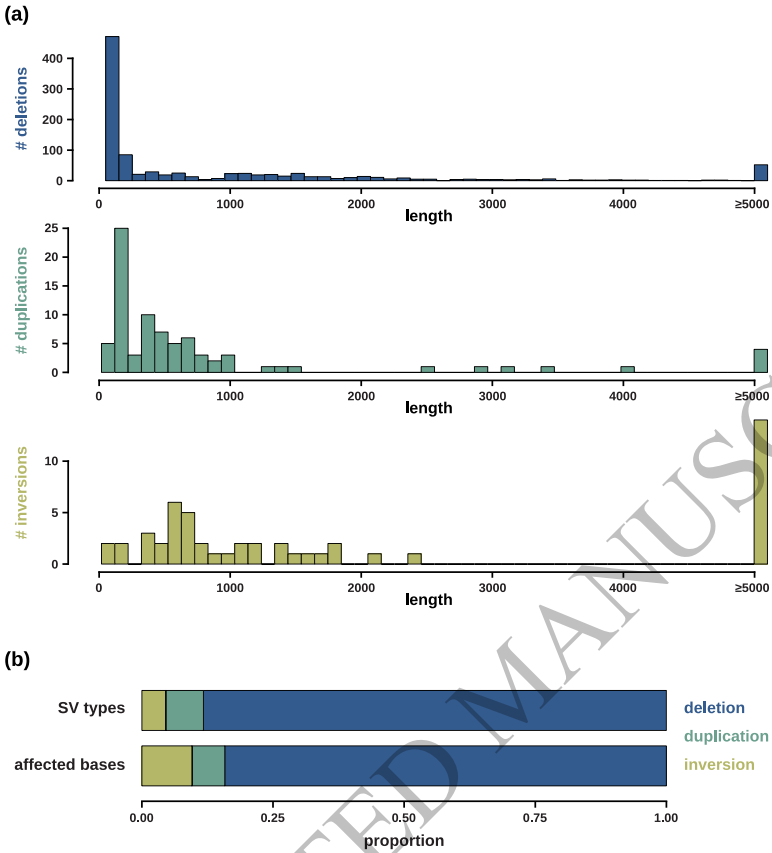
研究共鉴定1,133个常染色体SV,包含1,000个缺失(88.3%)、81个重复(7.2%)、51个倒位(4.5%)和1个插入。这些变异呈现典型长度分层现象:85%缺失短于200bp,而重复和倒位的中位长度分别达424bp和1,237bp。通过SnpEff注释发现,36.8%的SV位于内含子区,6个导致移码突变,4个影响外显子区。特别值得注意的是两个长度超过850bp的缺失,分别位于CLP1(与神经退行性疾病相关)和OPCML(肿瘤抑制基因)内部,可能通过移码突变导致功能丧失。
SV在基因组中呈非随机分布,12个SV密集区(每10Mb≥10个SV)富集于亚端粒区域——这些区域通常富含转座元件,易发生非等位同源重组。与人类和恒河猴数据相比,指猴个体SV数量(360-523个)显著偏低,这与该物种较低的SNV多样性一致,反映了其种群瓶颈效应。
通过1,596个基因型的手动验证发现,短读长数据存在35.7%的基因型错误率,主要源于复杂区域的比对困难。研究特别排除了275-325bp的Alu元件,因其高重复性会导致假阳性。这些发现强调了长读长测序在未来研究中的必要性。
这项研究首次绘制了指猴全基因组SV图谱,揭示了:1) SV类型和长度分布与其他灵长类保守;2) 疾病相关基因存在大效应SV;3) 当前短读长技术在SV检测中的局限性。这些发现不仅为理解灵长类基因组进化提供了新视角,更重要的是为指猴保护计划提供了分子层面的决策依据——例如通过SV监测评估近交程度,或针对关键基因设计保护策略。随着长读长测序技术的普及,未来研究有望发现更复杂的SV类型,从而更全面地评估这个"活化石"物种的遗传健康状态。

 生物通微信公众号
生物通微信公众号
知名企业招聘