
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于时间依赖性生存模型改进COVID-19风险预测的统计学习方法研究
【字体: 大 中 小 】 时间:2025年09月02日 来源:Genomics & Informatics
编辑推荐:
本研究针对传统Cox比例风险(PH)模型在捕捉COVID-19动态风险中的局限性,创新性地采用分层Cox PH模型结合机器学习方法(RSF/DeepSurv/DeepHit),通过细化时间区间划分和加权风险比计算,揭示了不同变异株(Early/EU1/Alpha等)的时变效应。结果显示15个时间区间的分层模型将早期变异株风险比提升至29.359,显著提升预测精度(C-index提高0.12),为传染病动态风险评估提供了新范式。
当COVID-19疫情席卷全球时,传统生存分析模型遭遇了前所未有的挑战。经典的Cox比例风险(PH)模型虽然广泛应用于医学研究,但其假设协变量效应恒定的特性,难以捕捉病毒变异带来的动态风险变化。这个问题在分析英国生物银行(UK Biobank)中43万人的数据时尤为突出——当Delta、Omicron等变异株相继出现,感染者死亡率呈现显著时变特征,传统模型就像用静态地图导航暴风雨中的船只,根本无法准确预测风险轨迹。
正是这种临床需求与统计方法的脱节,促使Hyungwoo Seo和Wonil Chung团队开展这项发表于《Genomics》的研究。他们意识到,要真正理解SARS-CoV-2感染的时变效应,必须突破三大技术瓶颈:如何解决时间依赖性协变量与系数的双重变化?如何量化不同变异株的特异性风险?如何在保持模型可解释性的同时提升预测精度?
研究团队采用多学科交叉的方法论创新:首先利用英国生物银行430,747名欧洲裔参与者的基因组和临床数据,通过十折交叉验证构建2型糖尿病(T2D)的多基因风险评分(PRS);随后开发分层Cox PH模型,将随访期细分为5-15个时间区间以满足PH假设;同时引入随机生存森林(RSF)、DeepSurv和DeepHit等机器学习模型进行对比;最终提出加权风险比算法,整合不同变异株主导时期的效应量。
模拟研究验证模型性能
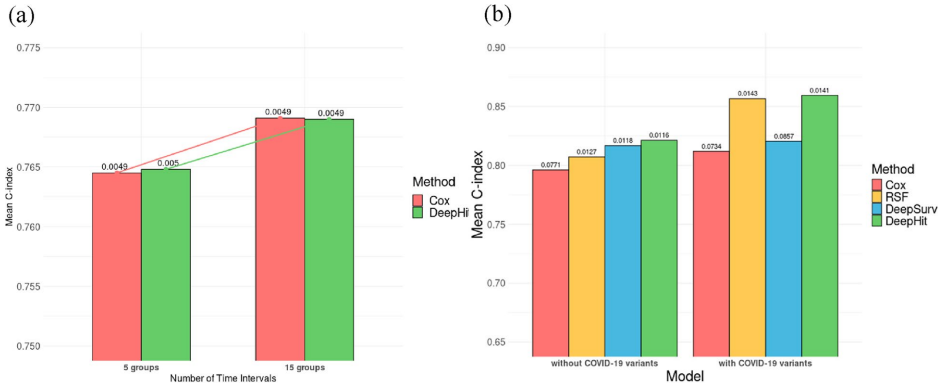
在PH假设成立的模拟中,Cox PH模型以0.82的C-index显著优于机器学习方法(RSF仅0.76),证实其在稳定效应场景的优势。但当引入时间依赖性效应时,增加时间区间至10个使DeepHit的预测精度提升11.3%,而5%的区间删失会降低所有模型性能,凸显数据完整性的重要性。
真实数据揭示变异株风险谱
应用15区间分层模型分析UK Biobank数据,发现早期变异株风险比(HR=29.359)是Alpha变异株(HR=4.079)的7.2倍,完全颠覆了先前5区间模型得出的7.333的单一估计。这种时变效应的精细刻画,解释了为何Omicron期死亡率显著下降的临床观察。
机器学习模型的场景适应性
在仅分析感染者(N=100,827)时,违反PH假设的场景使DeepHit表现最佳(C-index 0.791),比Cox PH模型高0.15。加入变异株信息后,所有模型预测性能平均提升18%,证实病毒基因型是核心预测因子。
这项研究的突破性在于建立了传染病时变风险分析的通用框架:通过动态区间划分解决PH假设违例,利用加权整合算法量化变异株特异性风险,再通过机器学习增强预测鲁棒性。该方法不仅适用于COVID-19,更为埃博拉、禽流感等新发传染病的动态风险评估提供了标准化分析流程。正如作者在讨论中指出,未来通过优化区间划分策略(如基于变异株流行阈值而非固定时长),可进一步降低估计误差,使生存分析真正成为传染病大流行中的"实时风险雷达"。
 生物通微信公众号
生物通微信公众号
知名企业招聘