
-

生物通官微
陪你抓住生命科技
跳动的脉搏
人源蛋白质合成中氨基酰-tRNA校对过程的动态旋转机制解析
【字体: 大 中 小 】 时间:2025年09月04日 来源:Nature Communications 15.7
编辑推荐:
本研究针对真核生物蛋白质合成过程中氨基酰-tRNA(aa-tRNA)选择机制的独特特征展开。研究人员通过1856次结构模拟实验,首次揭示人类核糖体A位点接纳aa-tRNA时需要30°动态旋转的关键机制。该发现阐明了亚基滚动(subunit rolling)导致的容纳通道空间限制是校对步骤速率降低十倍的结构基础,同时鉴定出延伸因子eEF1A通过保守碱性氨基酸与aa-tRNA的相互作用模式。研究成果发表于《Nature Communications》,为理解真核生物翻译保真度调控提供了新范式。
在生命活动的核心过程——蛋白质合成中,核糖体如同精密分子机器,需要准确解码mRNA信息并将对应氨基酸装配成多肽链。这一过程的保真度至关重要,细菌和真核生物分别能达到10-4和10-5的错误率。然而长期以来,人们对真核生物特别是人类aa-tRNA选择机制的理解远不如细菌深入。近期研究发现人类aa-tRNA校对步骤比细菌慢十倍,且伴随独特的亚基滚动现象,但具体结构动态机制仍是未解之谜。
为揭示这一科学问题,Diyva Sapkota、Karissa Y. Sanbonmatsu和Dylan Girodat团队采用结构模拟技术,对aa-tRNA进入人源核糖体A位点的过程进行系统研究。通过建立两种势能模型(Potential 1和Potential 2),分别捕捉可逆波动事件和早期/晚期接纳事件,共模拟1856次aa-tRNA接纳过程。研究结合距离测量(Relbow和RCCA)和角度分析(θtRNA),并引入亚基滚动距离(Rrolling)等创新性参数。
关键实验技术
研究主要采用结构分子动力学模拟技术,基于人源核糖体(PDB id: 6YOG)和兔源eEF1A复合物(PDB ID: 5LZS)构建初始模型。通过SWISS-MODEL建立人源eEF1A同源模型,使用GROMACS 2021进行显式溶剂平衡。模拟系统包含20 mM Mg2+和200 mM K+离子环境,采用AMBER 99力场。通过定义A/A态原生接触(4.5 ?内原子对)和非原生接触,设置不同权重(Potential 1: 0.13;Potential 2: 0.4)模拟可逆波动与单向接纳过程。
主要研究结果
人类aa-tRNA接纳需要肘部结构域和3'-CCA端的协同运动
通过测量aa-tRNA与肽基-tRNA间U60-U8距离(Relbow)和A76间距(RCCA),发现人类aa-tRNA采用"3'-CCA端优先"的独特路径。与细菌相比,人类EA-1中间态(Relbow=42?,RCCA=60?)显示出更大空间位移,自由能垒高达11-13.4 kBT(细菌为9.3-10.4 kBT)。
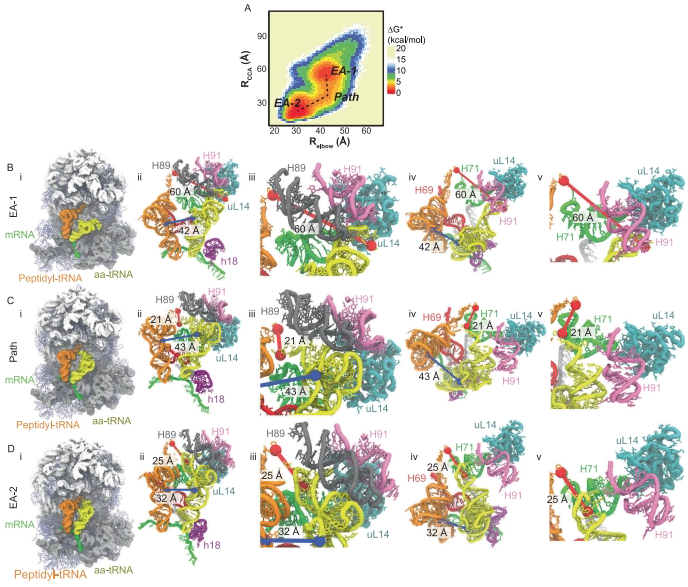
aa-tRNA在接纳过程中发生特征性30°旋转
通过测量tRNA平面法向量角度变化(θtRNA),首次发现人类aa-tRNA需绕反密码子茎旋转约30°才能通过接纳通道。自由能景观显示该旋转在Relbow=38?时达到能量最低点(θtRNA≈0°),此时tRNA与28S rRNA的H89发生关键相互作用。
亚基滚动导致接纳通道空间压缩
测量h14与SRL间距(Rrolling)显示亚基滚动(33.4?→27.5?)先于aa-tRNA接纳发生。溶剂可及表面积(SASA)分析显示人类tRNA接触面积(13079±121?2)显著大于细菌(14390±206?2),tRNA压缩程度增加3.7?。
eEF1A通过保守碱性氨基酸与aa-tRNA相互作用
发现eEF1A结构域III(DIII)的K378、K395等保守碱性氨基酸(保守度>94%)与aa-tRNA肘部磷酸骨架相互作用。开关I区R69仅能通过3'-CCA端小沟(不同于细菌的双路径),限制了tRNA初始运动轨迹。
研究结论与意义
该研究建立了人类aa-tRNA选择的结构动力学模型,阐明亚基滚动通过增加H89空间位阻,迫使aa-tRNA旋转调整以通过狭窄接纳通道,这合理解释了校对步骤速率降低的现象。发现eEF1A DIII与tRNA的持续性相互作用为真核生物特有调控机制,其保守性提示该机制在进化中的重要性。
研究成果不仅完善了蛋白质合成理论体系,更对相关疾病治疗具有指导价值:一方面为氨基糖苷类抗生素的物种特异性提供结构解释;另一方面为靶向翻译过程的抗癌(如plitidepsin)、抗病毒药物设计提供新思路。研究揭示的tRNA几何排列原则,为理解神经退行性疾病中蛋白质错误折叠机制开辟了新视角。