
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于GPU加速的JEMRIS平台实现高效MRI模拟:在动态效应及多物种定量研究中的应用
【字体: 大 中 小 】 时间:2025年09月05日 来源:Magnetic Resonance Materials in Physics, Biology and Medicine 2.0
编辑推荐:
本研究针对传统MRI模拟工具JEMRIS计算效率低下的问题,开发了支持CUDA的GPU加速版本(GPU-JEMRIS)。通过将计算密集型任务迁移至GPU、实现异步通信和混合精度支持,该研究在保持Bloch方程高精度求解(NRMSE<1%)的同时,使双精度和单精度模拟分别获得3-12倍和7-65倍的加速。研究成功模拟了呼吸运动导致的肝脏质子密度脂肪分数(PDFF)量化误差,为复杂多物种系统和大规模动态模拟提供了新工具。
磁共振成像(MRI)技术的发展始终伴随着对精确模拟工具的追求。Bloch方程作为描述核磁共振现象的核心数学模型,其数值求解在序列设计、伪影分析和定量成像研究中具有不可替代的作用。然而,随着研究问题日益复杂——涉及多组织模型、多线圈系统、动态效应(如呼吸运动)等,传统CPU-based模拟工具面临巨大计算挑战。特别是对于需要高时间分辨率的自适应步长ODE求解器,计算耗时已成为制约研究进展的瓶颈。
现有解决方案主要分为两类:基于矩阵运算的快速模拟器(如KomaMRI.jl)牺牲了部分理论精度换取效率;而基于ODE求解的JEMRIS虽精度优异,却因缺乏GPU支持难以应对大规模计算。这种矛盾在需要同时考虑多化学物种(如水/脂肪)和器官运动的肝脏脂肪定量等临床问题上尤为突出——既需要精确建模复杂物理过程,又受限于实际可承受的计算时间。
针对这一技术困局,来自德国慕尼黑工业大学的Aizada Nurdinova团队在《Magnetic Resonance Materials in Physics, Biology and Medicine》发表研究,通过深度改造开源框架JEMRIS,成功实现了基于CUDA的GPU加速方案。研究团队重构了Model、Bloch_CV_Model等核心类,将样本参数、线圈灵敏度图等数据异步传输至GPU,利用CUDA流实现并行操作,并开发了支持混合精度的ODE求解器。关键技术包括:1)采用SUNDIALS库的GPU-aware ODE求解器;2)使用CUB库优化多线圈信号接收时的归约操作;3)通过cuRAND在GPU端生成随机变量;4)对2.25百万自旋的几何模型进行加速测试;5)构建包含10种化学组分的腹部数字体模(基于DUKE数据库)模拟呼吸运动效应。
【主要结果】
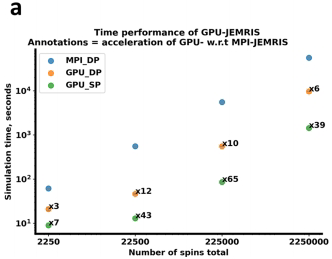
性能基准测试
通过EPI化学位移伪影、bSSFP带状伪影等经典案例验证,双精度GPU-JEMRIS与CPU版本结果差异可忽略(NRMSE<0.1%)。
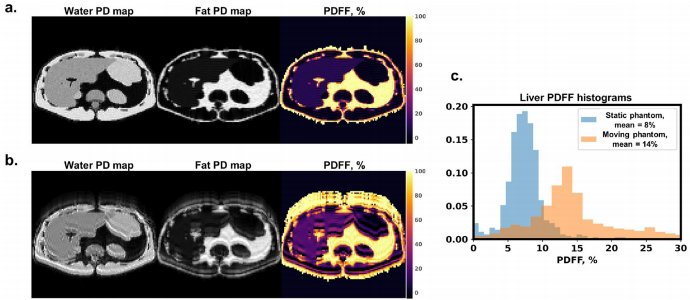
动态定量模拟
在六回波梯度回波(MEGRE)序列中,3像素的呼吸运动导致肝脏PDFF估值出现系统性偏移(均值从8%升至14%)。
精度-效率平衡
单精度运算虽引入约0.8%的k-space信号误差,但将7-8小时的肝脏模拟缩短至1.25小时,使大规模动态模拟在临床可接受时间内完成成为可能。
这项工作的突破性在于首次将自适应步长ODE求解器成功部署于GPU平台,既保留了JEMRIS"全物理建模"的核心优势,又通过异构计算解决了其传统弱点。研究者特别指出,当前版本尚未实现扩散、化学交换等扩展功能,且受限于单GPU内存。未来通过MPI-GPU混合并行,有望进一步拓展其在超大型体模、并行发射等场景的应用边界。该工具已开源发布,将为MRI物理研究、序列优化及临床前验证提供新的计算范式。
 生物通微信公众号
生物通微信公众号
知名企业招聘