
-

生物通官微
陪你抓住生命科技
跳动的脉搏
实验反馈驱动的生成模型优化显著提升生物序列设计成功率
【字体: 大 中 小 】 时间:2025年09月05日 来源:Nucleic Acids Research 13.1
编辑推荐:
本研究针对生成模型在生物序列设计中假阳性率高的问题,提出基于似然的实验反馈整合方案。研究人员通过DCA(Direct Coupling Analysis)模型对RNA(核糖核酸)和蛋白质序列进行设计,结合Group I内含子核酶等实验验证,将功能性序列生成率从6.7%提升至63.7%。该成果为克服进化数据局限性、实现精准生物分子设计提供了新范式。
在人工智能与合成生物学交叉领域,生成模型已成为设计功能性生物序列的重要工具。然而,这些模型面临一个致命缺陷:它们像一位想象力丰富但缺乏实践经验的画家,虽然能绘制出绚丽的蓝图,却常常无法通过实验验证——假阳性率居高不下。Francesco Calvanese等人在《Nucleic Acids Research》发表的研究,通过巧妙整合实验反馈,让生成模型真正"学会"区分功能性与非功能性序列,将设计成功率提升近10倍。
传统生成模型(如DCA、变分自编码器)依赖多重序列比对(MSA)数据训练,但自然进化数据存在两大局限:一是功能序列的采样稀疏性,二是实验室条件与生物体原生环境的差异。这导致模型生成的序列在计算机中"看起来很美",却在试管中"黯然失色"。正如研究者比喻:"进化约束如同模糊的地图,而实验反馈则是精准的GPS"。
为解决这一难题,团队开发了基于似然的反馈整合框架。其核心在于构建动态调整的权重函数w(b):实验验证的功能序列获得正权重(+1),失败序列被赋予负权重(-1)。通过调节强度参数λ,模型在保持Potts模型数学结构不变的前提下,重新校准参数,使生成序列向功能区域集中。
关键技术方法包括:1)采用直接耦合分析(DCA)构建初始生成模型P1;2)基于RNAeval计算RNA折叠自由能作为计算验证指标;3)对Group I内含子核酶开展高通量自剪接实验;4)通过主成分分析(PCA)可视化序列空间分布演变;5)开发两种反馈整合策略(标准REINT和分箱平衡REINT SB0)。实验数据来源于Rfam数据库的tRNA、核糖开关等RNA家族,以及chorismate mutase(CM)蛋白质家族。
材料与方法
研究创新性地将实验数据转化为修正频率?f,通过公式(10)-(12)实现DCA参数的迭代优化。针对RNA设计,采用eaDCA(edge activation DCA)提升效率;蛋白质案例则使用adabmDCA(adaptive Boltzmann Machine DCA)的GPU加速实现。
结果与讨论
计算验证
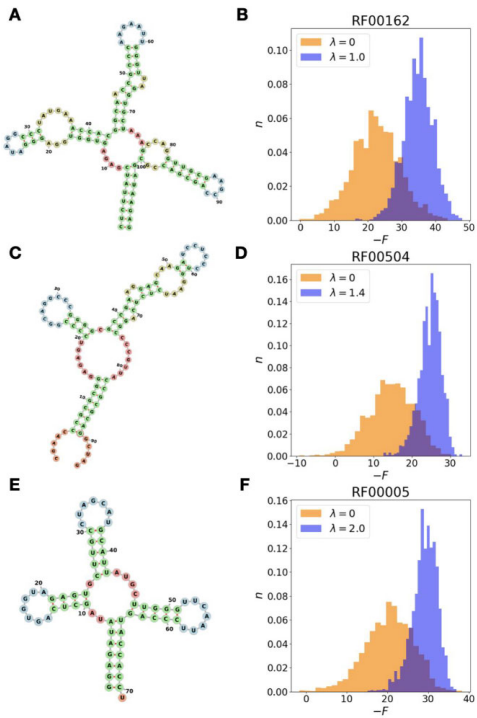
在SAM核糖开关(RF00162)测试中,反馈整合使模型熵S从63.4降至53.3,但功能性序列比例从49.9%飙升至99.4%。
蛋白质设计突破
对CM酶的设计中,逻辑回归评估的功能序列比例从36.6%提升至66.3%。PCA分析揭示,新模型生成的序列(图3D蓝色点)主动避开实验验证的非功能区域(图3E红色点)。
实验验证里程碑
Group I内含子核酶设计中,REINT SB0模型在60个突变位点仍保持23.6%活性,远超基础模型2.0%的水平(表3)。
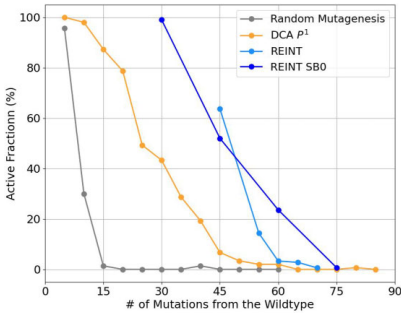
这项研究证实:通过"计算设计→实验验证→反馈优化"的闭环,即使不增加模型复杂度,也能大幅提升设计可靠性。其意义不仅在于技术突破,更揭示了生物序列设计的本质——自然进化数据只是起点,实验反馈才是划定功能边界的"金标准"。未来,这种迭代框架有望应用于CRISPR引导RNA设计、人工酶开发等领域,加速合成生物学从经验摸索向理性设计的跨越。
 生物通微信公众号
生物通微信公众号
知名企业招聘