
-

生物通官微
陪你抓住生命科技
跳动的脉搏
CORAL方法实现25万种稀有节肢动物的高效分布建模与预测
【字体: 大 中 小 】 时间:2025年09月24日 来源:Nature Methods 32.1
编辑推荐:
面对DNA宏条形码技术产生的大规模物种数据中绝大多数为稀有物种的挑战,研究人员开发了“从常见到稀有迁移学习”(CORAL)方法。该方法通过常见物种构建骨干模型,利用系统发育和性状信息为稀有物种建立信息先验,成功对马达加斯加255,188种节肢动物进行了分布推断,显著提升了稀有物种预测精度,解决了生态学中的“稀有物种悖论”。
随着DNA宏条形码等高通量监测技术的迅猛发展,生态学家们能够获取前所未有的生物多样性数据规模——单次研究即可记录数万至数十万个物种。然而这些海量数据中隐藏着一个根本性矛盾:绝大多数物种都是极其稀有的,在255,188个物种级操作分类单元(OTUs)中,有71%的物种仅在单个样本中出现,传统物种分布模型(SDMs)往往因为数据稀疏而放弃对这些稀有物种的建模分析。这种现状形成了所谓的“稀有物种悖论”:最需要保护信息的物种恰恰是最缺乏研究数据的物种。
以往解决这一难题的尝试包括使用集成小模型(ESMs)或利用系统发育信息进行推断,但面对数十万量级的物种时,这些方法在计算效率和统计效能上都面临巨大挑战。联合物种分布模型(JSDMs)虽然能通过共享环境响应实现物种间信息借用,但其计算复杂度随物种数量增长呈指数级上升,使得包含全部物种的完整模型几乎不可能实现。
针对这一瓶颈,由芬兰于韦斯屈莱大学Otso Ovaskainen和美国杜克大学David Dunson领衔的国际研究团队在《Nature Methods》发表了创新性研究方法。他们开发了“从常见到稀有迁移学习”(Common to Rare Transfer Learning, CORAL)框架,通过三阶段建模策略巧妙解决了大规模稀有物种的分布预测问题:首先利用876个常见物种(出现次数≥50)构建HMSC骨干模型,获取潜在环境因子估计;然后通过系统发育相关性和性状信息构建稀有物种的条件先验分布;最后对每个稀有物种独立进行贝叶斯probit回归,但使用来自常见物种的强信息先验。
研究人员为验证CORAL方法的有效性,将其应用于马达加斯加节肢动物群落数据。该数据集包含53个地点2,874个样本的COI基因序列,经OptimOTU流程处理得到255,188个物种级OTUs。环境预测因子包括年降水量和年平均温度的二次多项式及交互项、采样日期的周期函数(sin/cos)以及测序深度对数转换值。作为系统发育的代理,使用了从界到种的全级别分类信息,并为未明确分类的序列创建了伪分类单元。
主要技术方法包括:基于R包Hmsc的层次群落模型(HMSC)构建骨干模型;使用MCMCpack进行单物种贝叶斯probit建模;通过系统发育相关矩阵实现物种间信息传递;采用交叉验证比较模型预测性能(AUC、Tjur's R2、PRAUC等指标)。
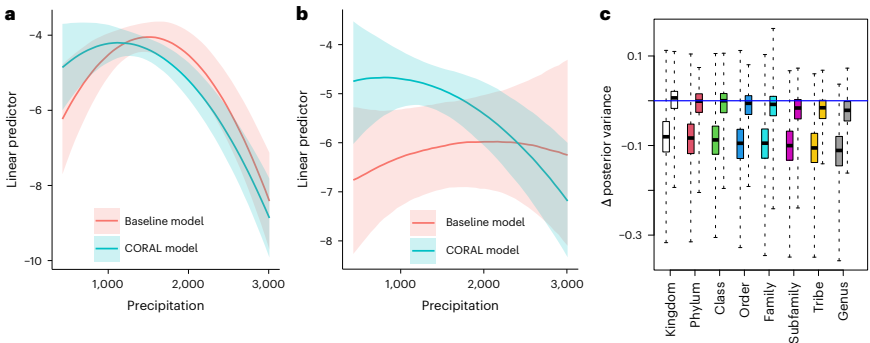
骨干模型分析显示,常见物种的分布主要受站点水平变异驱动,其中气候变量解释了48%的变异,站点水平潜在因子解释了42%的变异。物种对环境预测因子的响应呈现强烈的系统发育结构(后验平均ρ=0.65,Pr(ρ>0)=1.00),为从近缘物种借用信息提供了基础。
方差缩放因子k介于0.13-0.70之间(均值0.34),表明通过CORAL先验实现了显著的方差缩减。与骨干模型中近缘物种关系越密切的稀有物种,其先验模型的预测能力越强(AUC>0.5),且出现次数超过10次的物种预测精度高于极度稀有物种。

通过CORAL方法成功获得了所有物种(常见和稀有)对环境预测因子的响应模式。常见物种的响应倾向会传递给分类学上相近的稀有物种,但当稀有物种数据提供足够证据时,其后验估计仍可偏离先验预期。
与基线模型相比,CORAL模型显著提高了参数估计精度,特别是对极度稀有物种(出现次数≤10)。CORAL后验方差平均降低幅度与物种和骨干模型的亲缘关系密切程度正相关。
模型验证表明,CORAL后验预测与观测数据高度一致,仅在最稀有物种的丰富度估计上存在轻微高估。模型在不同温度和湿度范围内拟合均匀,说明协变量效应设定无明显误设。
对22,140个出现次数≥5但未纳入骨干模型的物种进行交叉验证,CORAL模型在所有指标上均显著优于基线模型:AUC从0.86提升至0.94,Tjur's R2从0.03提升至0.08。性能提升主要来源于常见物种潜在因子的纳入,而非直接从近缘物种借用信息。

为进一步验证CORAL推断稀有物种环境响应的能力,研究人员对常见物种数据进行90%随机掩蔽处理,模拟常见物种变为稀有物种的情景。CORAL估计的环境响应与全数据骨干模型估计值高度相关(气候和季节预测因子相关性0.68,样本水平潜在因子0.69,站点水平潜在因子0.73)。

CORAL方法突破了传统联合群落模型对物种数量的限制,通过从常见物种骨干模型中借用信息,使对极度稀有物种的统计有效建模成为可能。该方法成功实现了对25万种马达加斯加节肢动物的分布建模,其中99.7%的物种传统方法会因数据稀疏而放弃分析。
研究发现节肢动物对气候、季节和潜在预测因子的响应具有显著系统发育结构,表明物种分布受到祖先生态位保守性的约束。这一发现对理解马达加斯加高特有化现象的形成机制具有重要意义——即使在适应本地环境过程中,物种仍保持了强烈的祖先生态位信号。
方法学上,CORAL的三大优势值得关注:一是计算效率高,稀有物种模型可独立并行拟合,使数百万物种的推断成为可能;二是存储高效,通过多元正态近似后验分布,极大减少了存储需求;三是灵活性好,即使没有系统发育信号或性状信息,仍可通过潜在因子和物种平均响应实现信息借用。
该研究不仅提供了方法论突破,也为全球生物多样性监测提供了实用工具。随着DNA宏条形码技术的普及,生态学家将面临越来越多的大规模、高稀疏度物种数据,CORAL框架为此类数据的全面分析提供了可行路径,真正实现了对“生物多样性”中“多样性”成分的完整建模。
未来研究方向包括将CORAL扩展至丰度数据、零膨胀模型以及其他类型的生物多样性数据(如声学监测、图像识别等)。同时,如何进一步提高超大规模数据集的计算效率,以及如何整合多源异构数据,仍是需要持续探索的问题。
 生物通微信公众号
生物通微信公众号
知名企业招聘